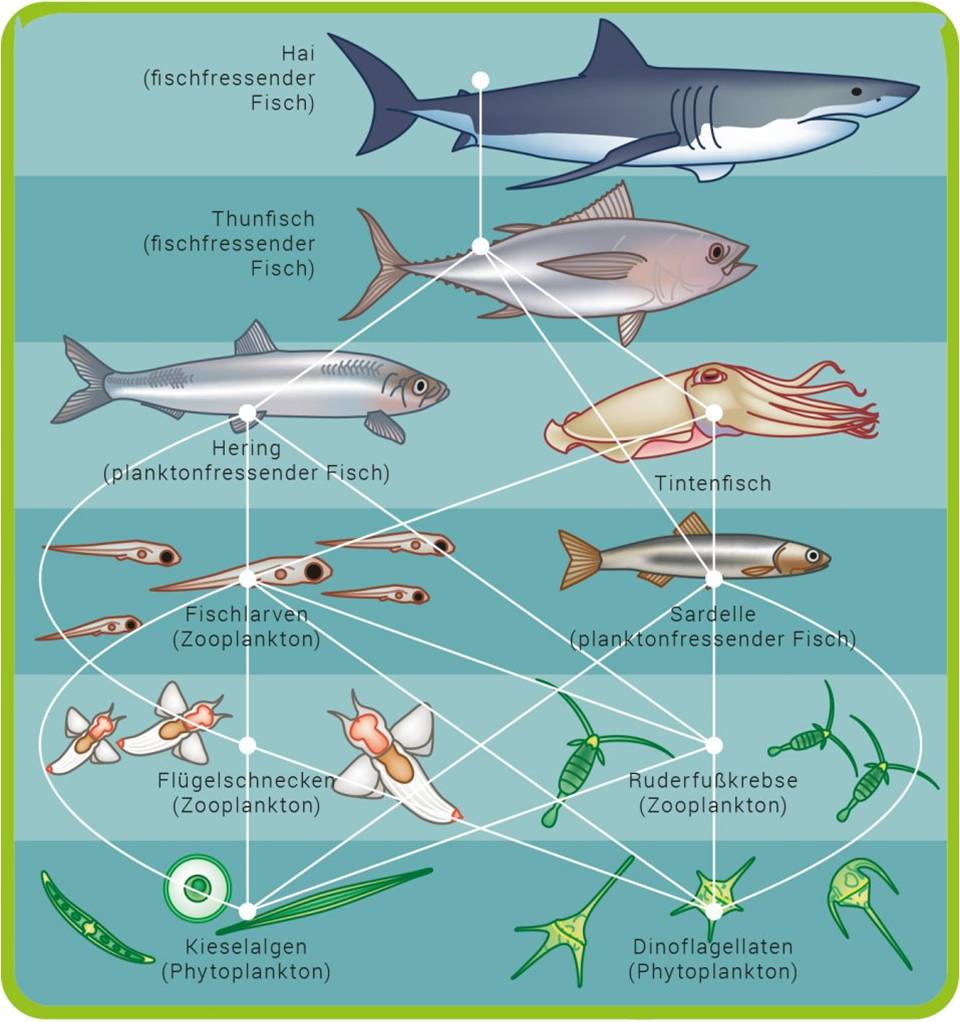
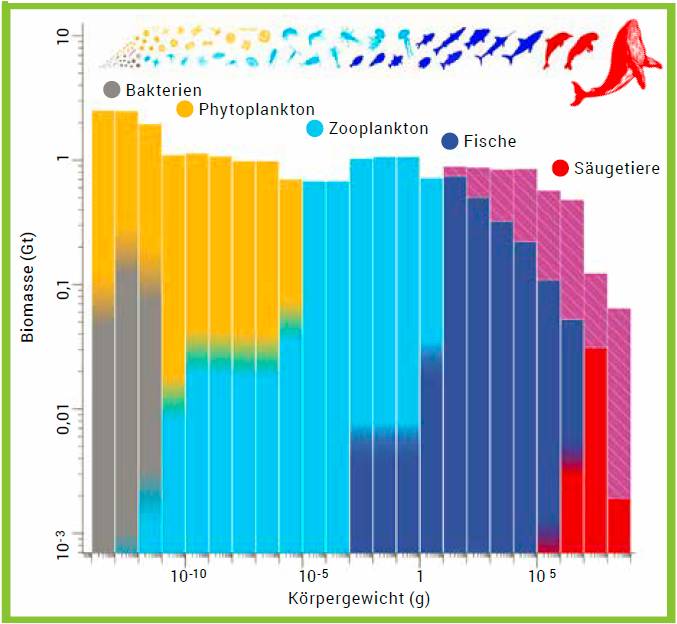
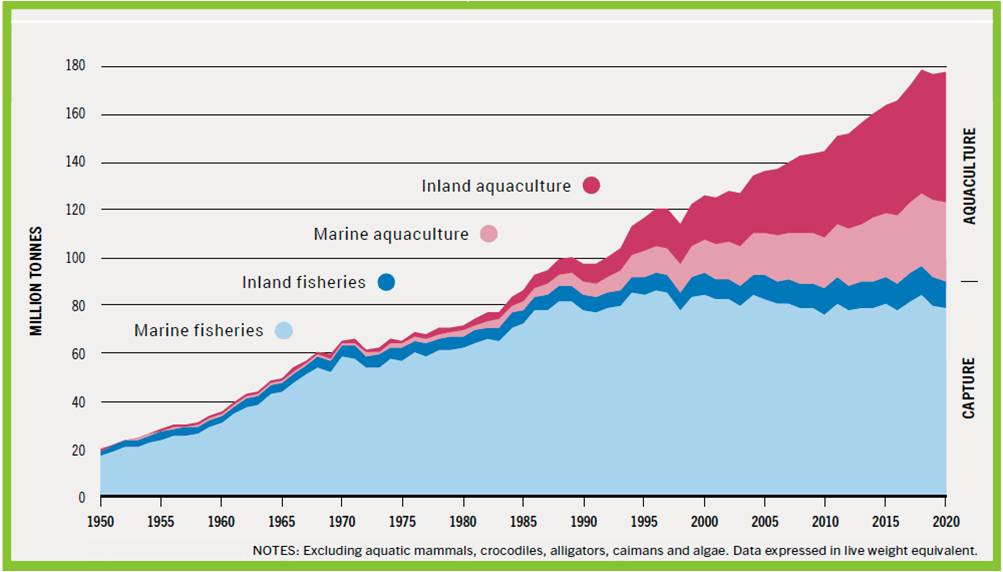
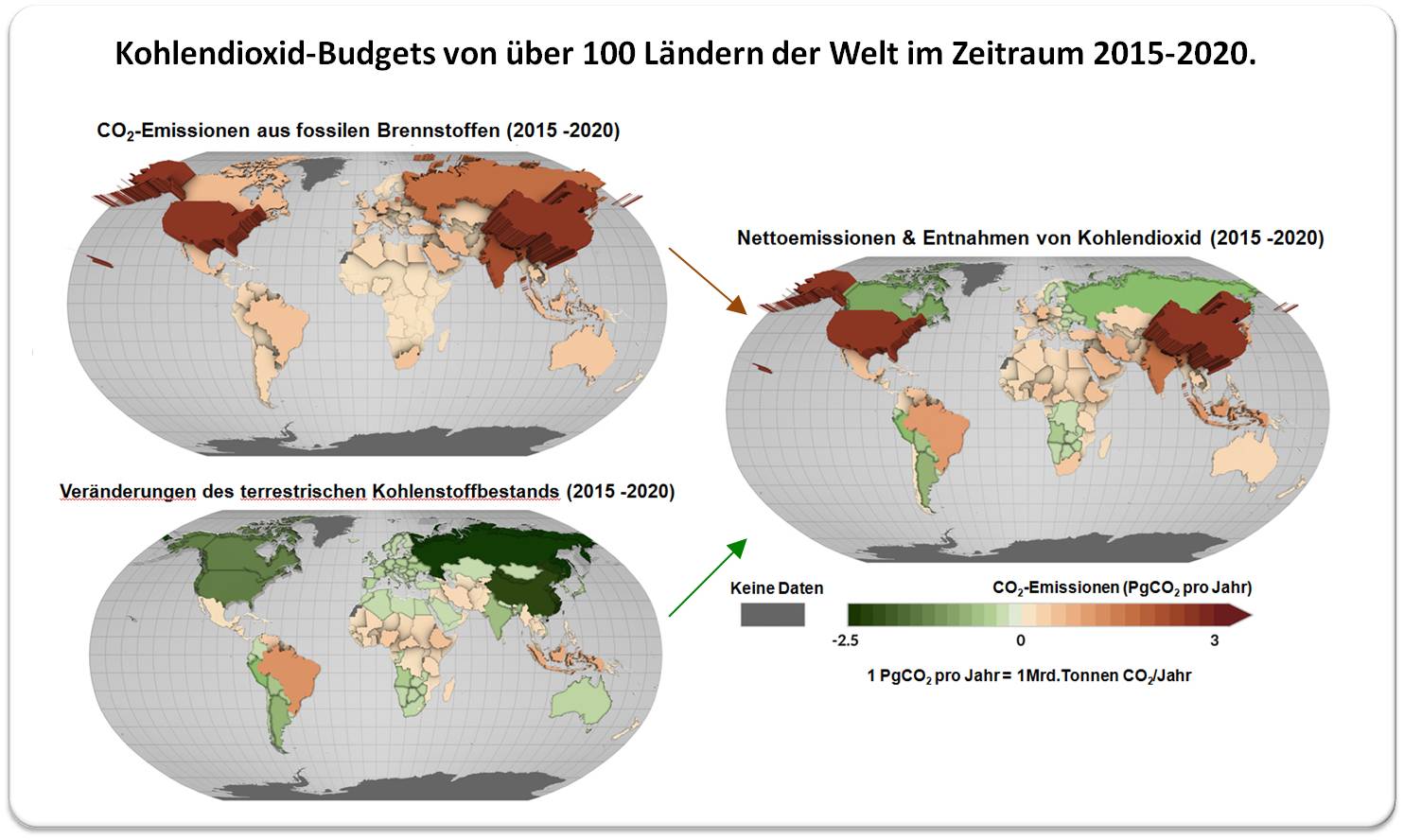
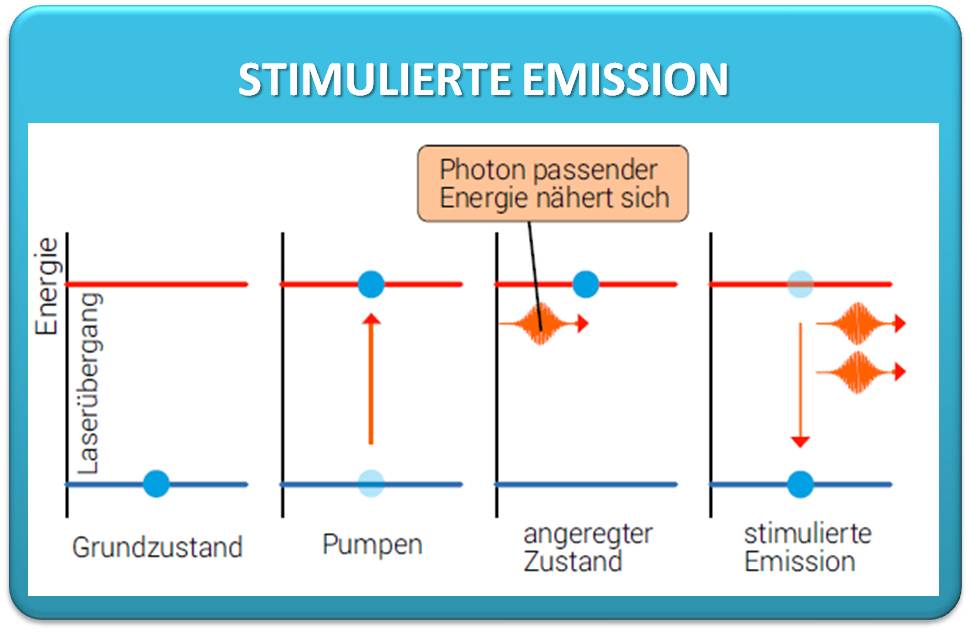
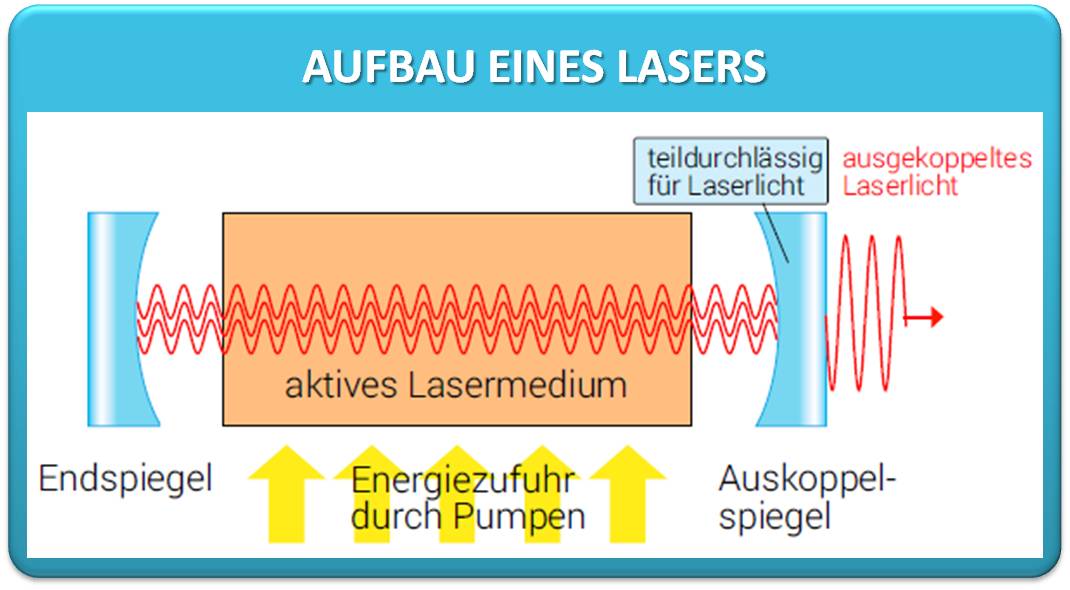
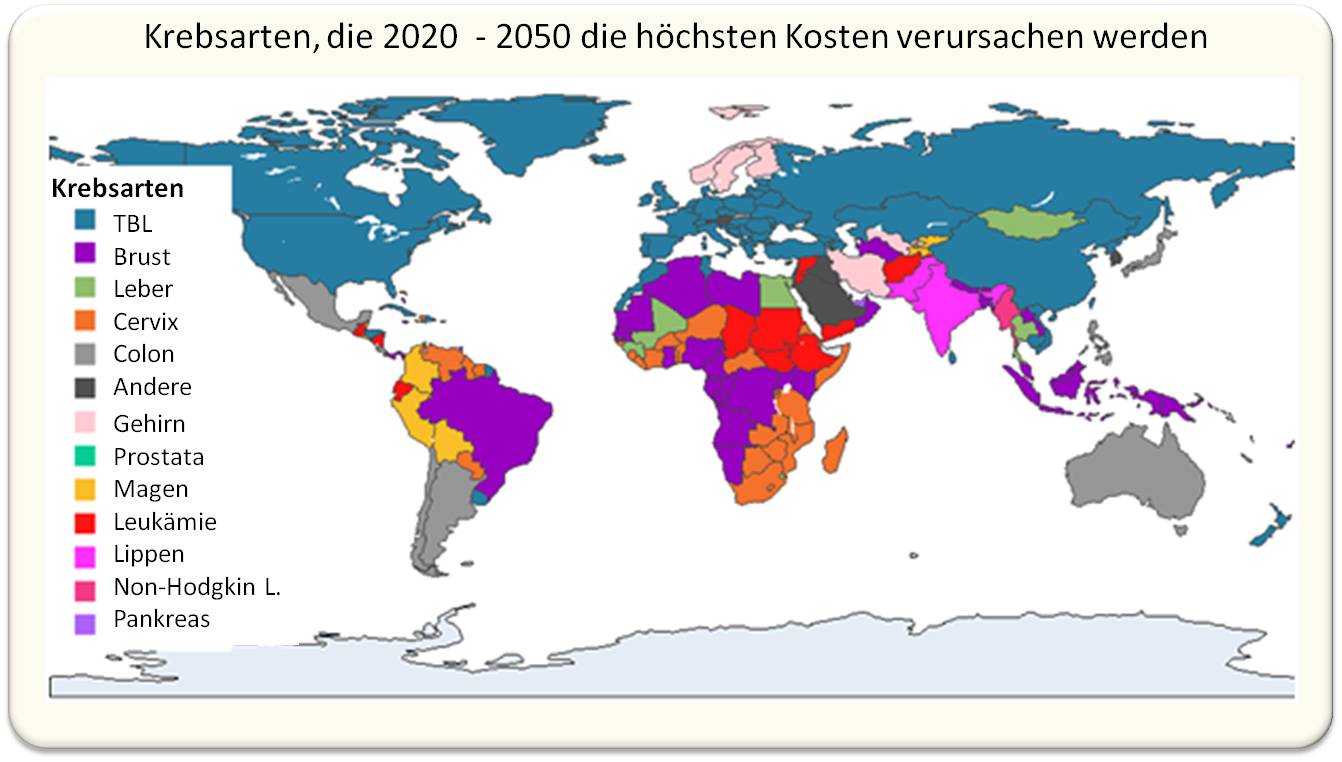
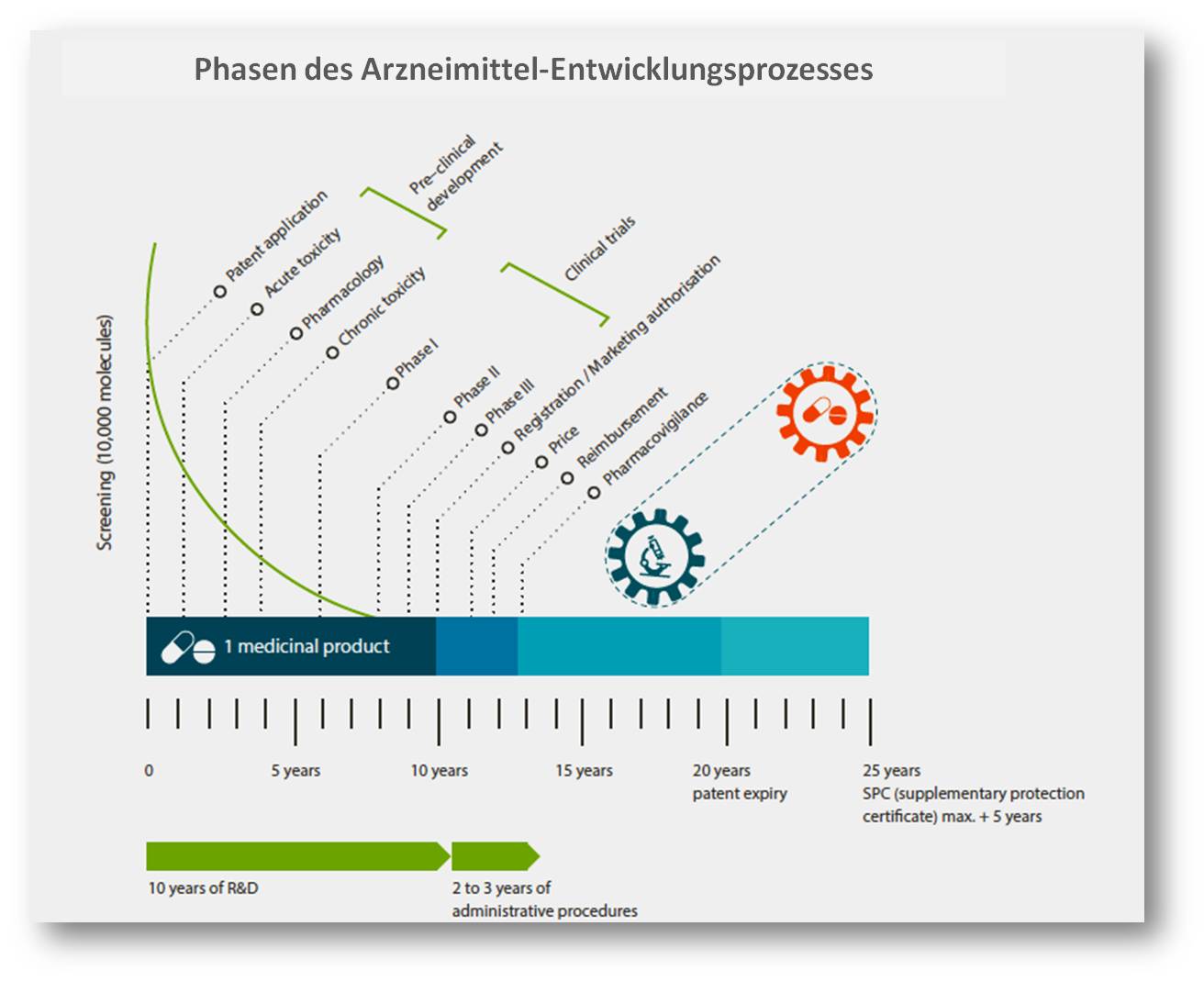
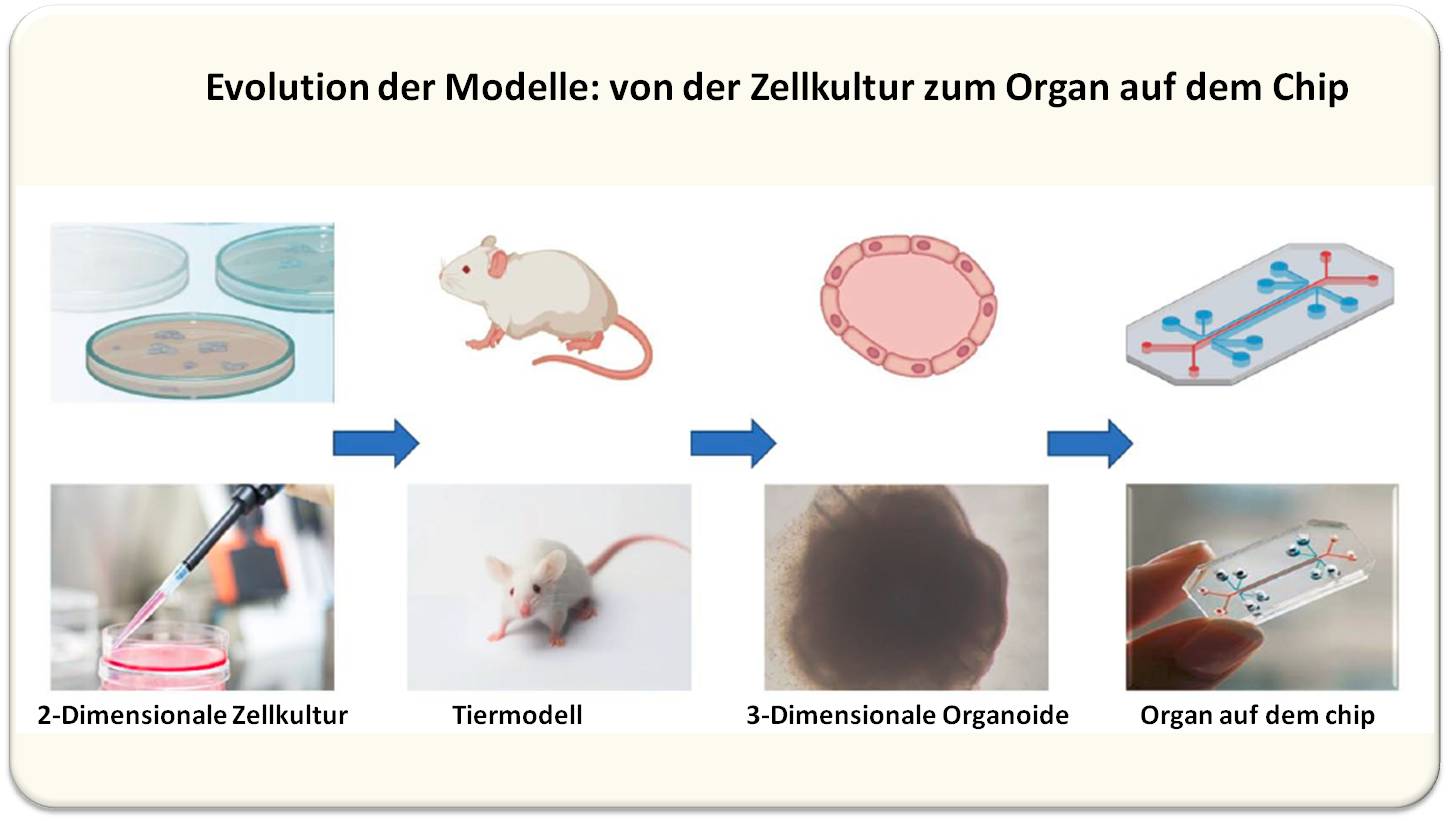
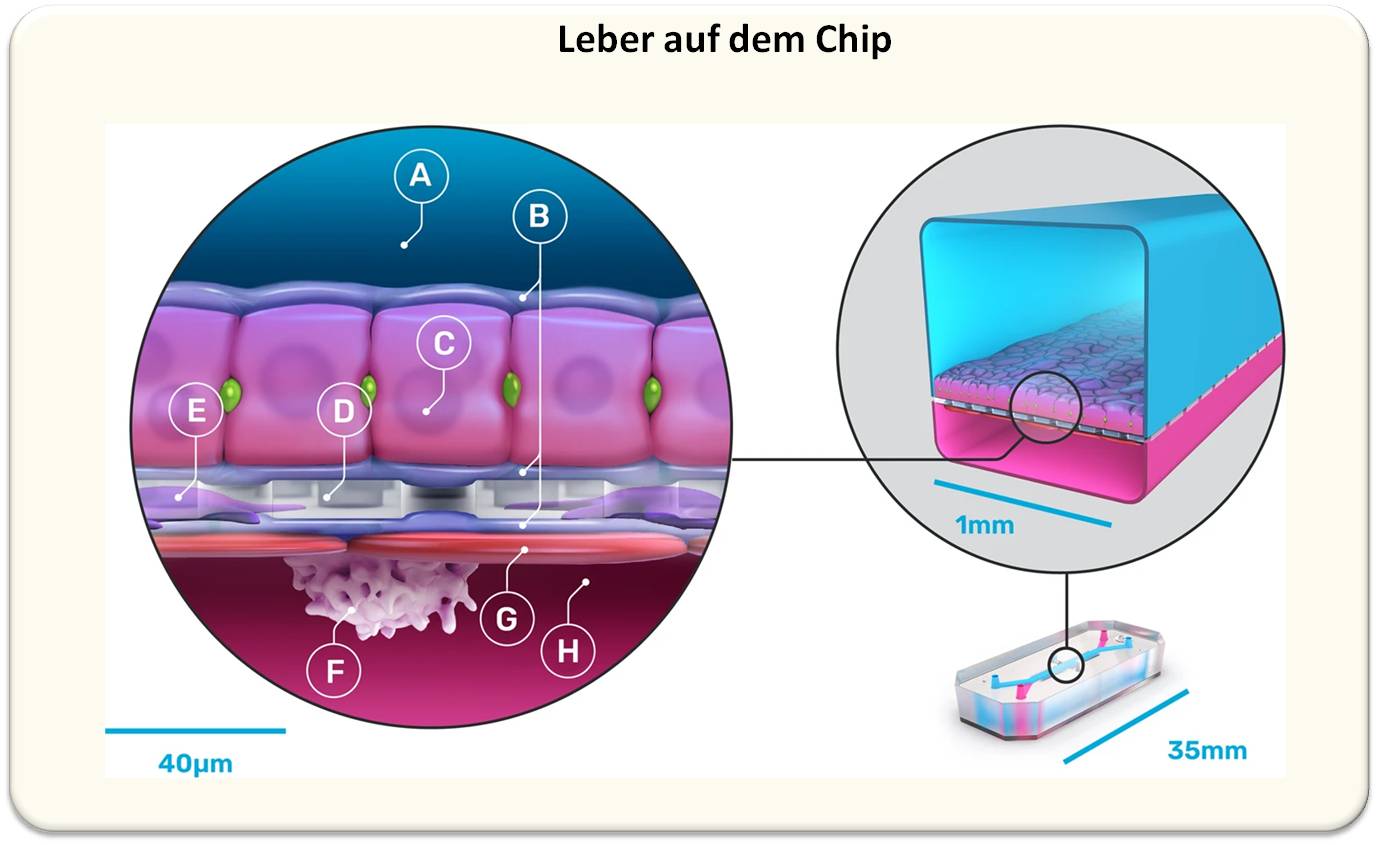
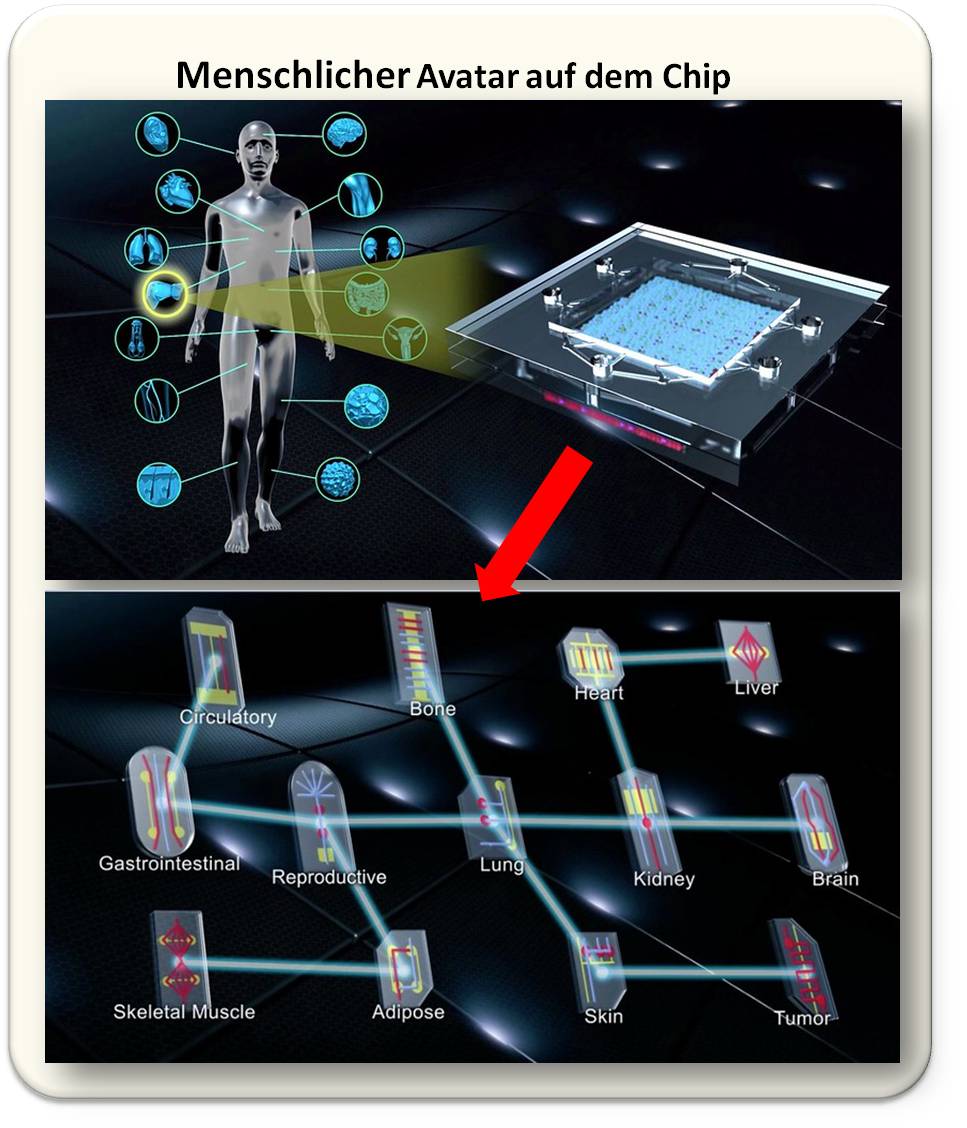
2023
2023 inge Thu, 05.01.2023 - 16:31Hämoglobin trägt zur Barrierefunktion unserer Haut bei
Hämoglobin trägt zur Barrierefunktion unserer Haut beiFr.29.12.2023 — Inge Schuster
Hämoglobin, der in den Erythrozyten zirkulierende Transporter von Sauerstoff, wird offensichtlich auch in anderen Zelltypen produziert. Eine neue Untersuchung zeigt erstmals, dass Hämoglobin in den obersten Schichten der Epidermis und auch in den Haarfollikeln gebildet wird. Es wird als Antwort auf oxidativen Stress - wie er beispielweise durch UV-Bestrahlung entsteht - in den Keratinozyten hochreguliert und kann dort die Generierung von reaktiven Sauerstoff-Spezies hemmen und die Zellen vor deren Folgen schützen. Die Expression von Hämoglobin ist somit eine neue Facette in der Barrierefunktion der Epidermis.
Mit Hämoglobin verbindet wohl jeder sofort den Blutfarbstoff in den roten Blutkörperchen, den Erythrozyten, der den in der Lunge eingeatmeten Sauerstoff (O2) über den Blutkreislauf zu den Zellen unserer Gewebe und Organe transportiert. Die ununterbrochene Versorgung mit Sauerstoff ist unabdingbar, um die zur Instandhaltung und zum Funktionieren unserer Körperzellen nötige Energie in den Mitochondrien (über Zellatmung und oxydative Phosphorylierung) zu erzeugen; Sauerstoff ist aber auch unentbehrlich, um die zahllosen Oxydationsvorgänge im Stoffwechsel - zu Synthese und Metabolismus von körpereigenen Substanzen und zum Abbau von Fremdstoffen - zu ermöglichen. Für den Sauerstofftransport steht dabei eine sehr hohe Kapazität zur Verfügung: Erythrozyten bestehen zu etwa 90 % (ihres Trockengewichts) aus Hämoglobin und machen bis zu 50 % des Blutvolumens aus; das entspricht 120 bis 180 Gramm Hämoglobin pro Liter (von insgesamt 5 Liter) Blut, wobei ein Gramm Hämoglobin 1,34 ml Sauerstoff binden kann, und ein Erwachsener bei körperlicher Ruhe rund 280 ml Sauerstoff pro Minute einatmet. Im Gegenzug zur Abgabe von Sauerstoff bindet Hämoglobin einen Teil (rund 23 %) des bei der Zellatmung freiwerdenden CO2, das dann über das Blut in die Lunge transportiert und dort abgeatmet wird.
Zur Charakterisierung von Hämoglobin
Seine lebensnotwendige Rolle und hohe Verfügbarkeit haben Hämoglobin zu einem der meistuntersuchten Proteine der letzten 150 Jahre gemacht. Dazu gehört auch, dass die ersten Röntgenkristall-Analysen von 3D-Proteinstrukturen an Hämoglobin (durch Max Perutz) und dem strukturverwandten, einfacher aufgebauten Myoglobin - dem Sauerstoff-Speicher in der Muskulatur - (durch John Kendrew) stattfanden und 1962 mit dem Nobelpreis ausgezeichnet wurden.
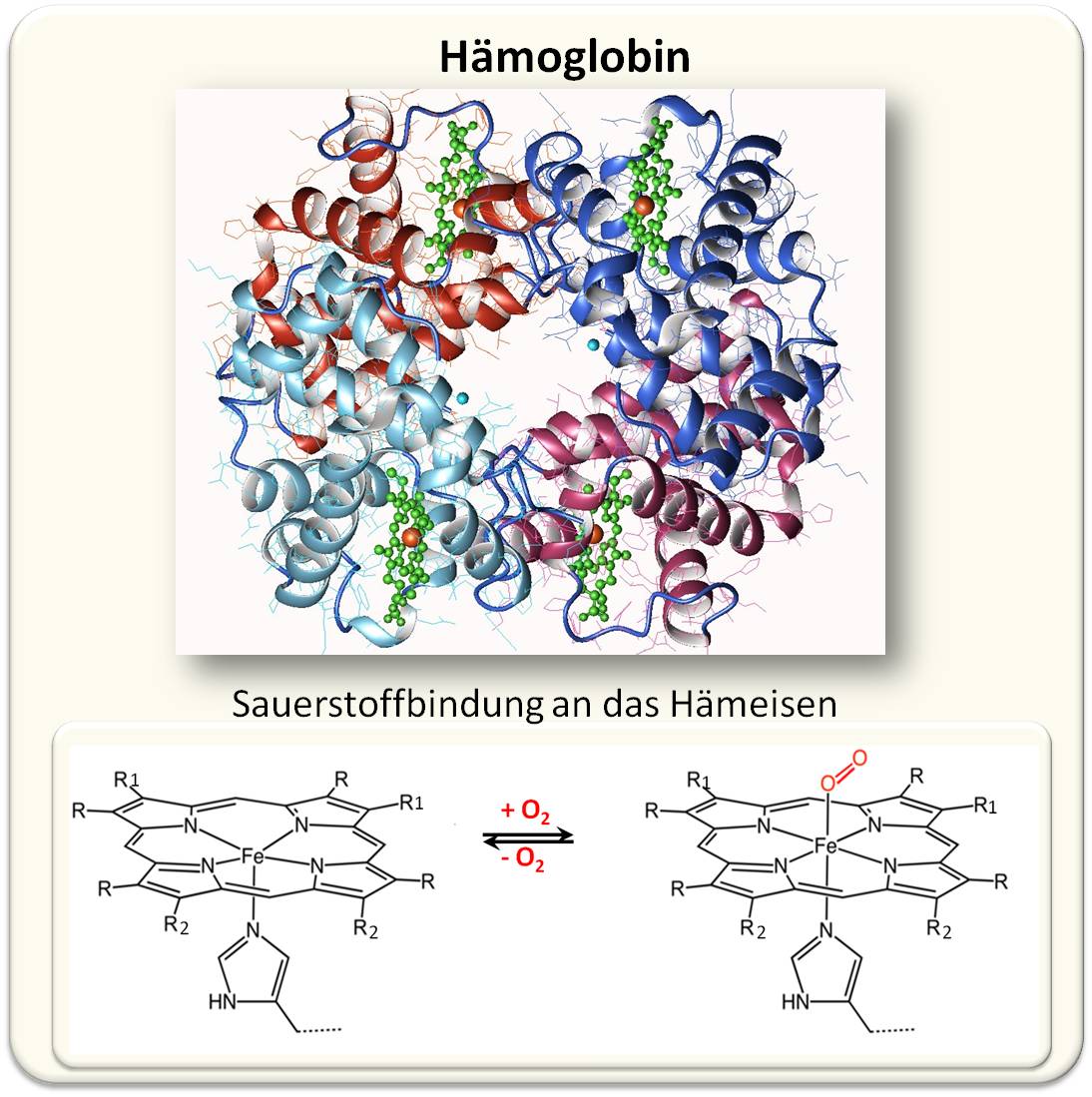
| Abbildung 1. Hämoglobin A (adulte Form - HbA) als Sauerstofftransporter. Das Bändermodell zeigt ein Tetramer aus je 2 αund 2 β Untereinheiten (Rot- und Blautöne), in die jeweils eine Hämgruppe - ein Protoporphyrin (grün) mit zentralem Eisenatom (rot) - über einen Histidinrest des Globins gebunden vorliegt. Sauerstoff bindet reversibel in der O2-Form an die 6. Koordinationsstelle des Hämeisens. (Bild: Fermi, G., Perutz, M.F. https://www.rcsb.org/structure/3hhb Lizenz: cc-by-sa) |
Demnach ist Hämoglobin aus 4 Untereinheiten aufgebaut; beim erwachsenen Menschen sind das jeweils 2 Hb α und 2 Hb β Globine. Jede dieser Untereinheiten besteht aus 142 bis 146 Aminosäuren langen Peptidketten, die jeweils 8 Helices bilden mit einer Tasche, in der das Häm - ein Porphyrin-Molekül mit einem Eisen Fe2+ als Zentralatom - eingebettet ist. Abbildung 1.
An Hämoglobin wurde in den 1960er-Jahren auch erstmals das Phänomen einer kooperativen Bindung entdeckt; im konkreten Fall: Wenn Sauerstoff als O2-Molekül an das Hämeisen einer Untereinheit bindet, induziert es darin eine Konformationsänderung, die sich auf die anderen 3 Untereinheiten überträgt und dort eine erleichterte Bindung der anderen 3 Sauerstoffmolküle zur Folge hat.
Hämoglobin kann auch andere chemische Gruppen binden und transportieren: Direkt an das Hämeisen binden beispielsweise Kohlenmonoxid (CO), Cyanid (CN-) oder Sulfid (S2-), hemmen dadurch die Bindung von Sauerstoff mit schwerwiegenden Folgen. An Aminogruppen der Globine binden Moleküle wie CO2 und das Signalmolekül Stickstoffmonoxid (NO), ein gefäßerweiterndes Gas.
Hämoglobin fungiert nicht nur als Transporter
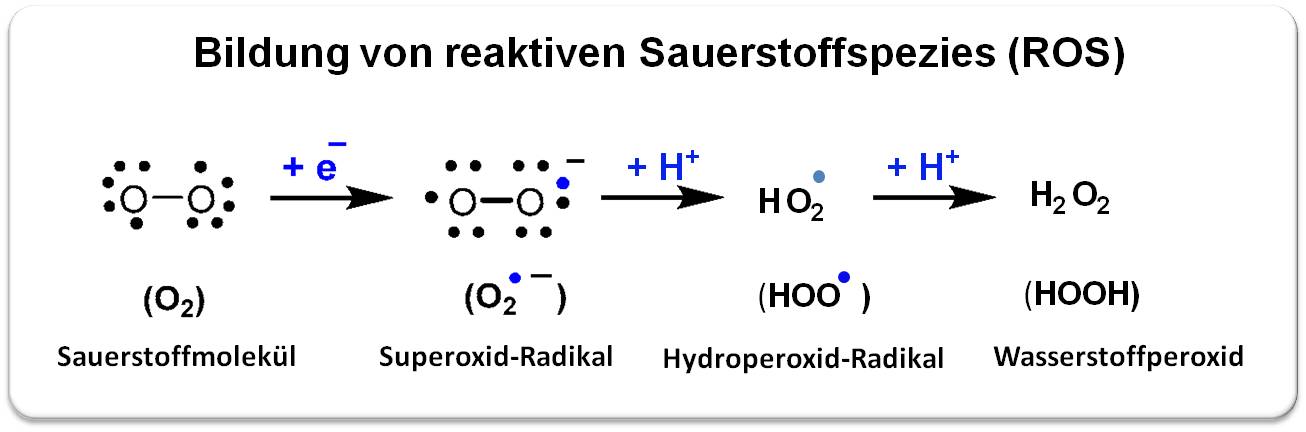
Es generiert auch reaktive Sauerstoffspezies. Bei der Bindung von Sauerstoff kommt es häufig zur Autooxidation von Hämoglobin: dabei wird das Hämeisen Fe2+ zu Fe3+ oxidiert (Methämoglobin) und gleichzeitig Sauerstoff zum Superoxid-Radikal reduziert, aus dem dann das Hydroperoxid-Radikal und in weiterer Folge Wasserstoffperoxid entstehen. Abbildung 2.
| Abbildung 2. Die Bindung von Sauerstoff an das Hämeisen führt häufig zur Generierung von reaktiven Sauerstoffspezies (ROS). Vereinfachte Darstellung. |
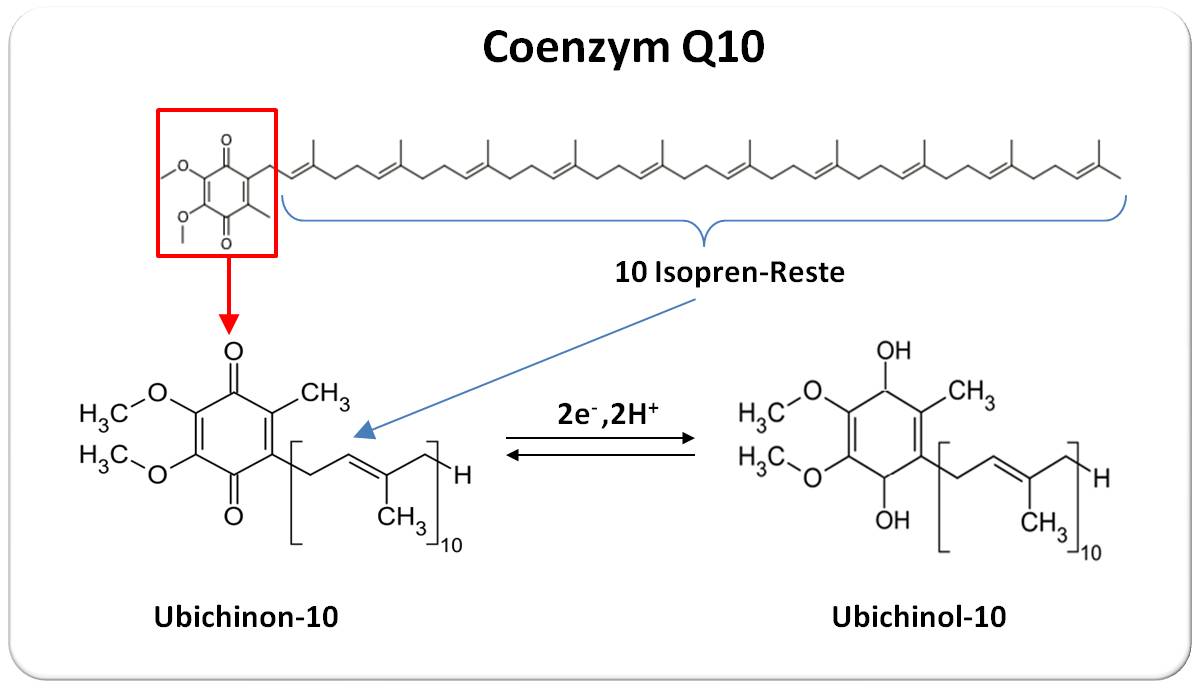
Die reaktiven Sauerstoffspezies reagieren (nicht nur) mit Hämoglobin und lösen eine Kaskade oxidativer Reaktionen aus, die zum Abbau des Häms, Freisetzung des Hämeisens (das freie Radikalreaktionen katalysiert), irreversiblen Vernetzungen der Globinketten und schlussendlich zum Abbau von Hämoglobin führen. Diese Reaktionen treten auch auf, wenn Hämoglobin oder Erythrocyten mit exogenem Wasserstoffperoxid reagieren, wie er bei anderen metabolischen Vorgängen (beispielsweise in den Mitochondrien) produziert wird und sogar, wenn Erythrozyten gelagert werden. Allerdings - solange Hämoglobin eingeschlossen in den Erythrozyten zirkuliert, schützt ein äußerst wirksames antioxidatives Abwehrsystem aus diversen Enzymen und endogenen niedermolekularen Substanzen wie Glutathion, Vitamin C, Coenzym Q10, etc. vor den kontinuierlich entstehenden reaktiven Sauerstoffspezies. Übersteigt die ROS-Produktion die Kapazität des Abwehrsystems, kommt es zu einer Beeinträchtigung der Sauerstoffzufuhr, Schädigung der Erythrozytenmembran verbunden mit dem Austritt von (geschädigtem) Hämoglobin und anderen Biomolekülen sowie ROS, die Entzündungsprozesse auslösen können.
Die Reaktion von Hämoglobin mit H2O2, die zur Zerstörung des Proteins führt, bedeutet gleichzeitig ein hochwirksame Neutralisierung von ROS und Schutz vor deren schädlichen Auswirkungen.
Hämoglobin wird auch außerhalb der Erythrozyten produziert.....
Dass Hämoglobin außerhalb der im Blutkreislauf zirkulierenden Erythrozyten eine Rolle spielt, war lange unbekannt. Erst in den letzten beiden Jahrzehnten zeigen mehr und mehr Untersuchungen, dass Hämoglobin in vielen, nicht den Erythrozyten oder ihren Vorläuferzellen angehörenden Zelltypen exprimiert wird [1]. Detektiert wurde Hämoglobin oder seine Untereinheiten Hb α und Hb β u.a. in Makrophagen, in Epithelzellen der Alveolen in der Lunge, im Pigmentepithel der Retina, in den Mesangialzellen der Niere, in Leberzellen, in Neuronen und Gliazellen, in der Schleimhaut der Gebärmutter und des Gebärmutterhalses, in den Chondrozyten des Knorpelgewebes und nun kürzlich in den Keratinozyten der obersten Hautschichten der Epidermis [2].
Die Funktion, die Hämoglobin in den einzelnen Zelltypen ausüben dürfte, reicht von Speicherung und Abgabe von Sauerstoff in spärlich durchbluteten Geweben (z.B. durch Hämoglobinkondensate in den Chondrozyten der Knorpelsubstanz [3]) über die Inaktivierung von reaktiven Sauerstoffmolekülen (H2O2, Hydroxyl-Radikal, Superoxid-Anion) bei oxidativem Stress (z.B: in Hepatozyten [1]) und von Stickoxiden (z.B. in der Lunge [4]) bis hin zu antibiotischen und antiviralen Eigenschaften von Peptiden, die durch Proteasen vom Hämoglobin abgespalten werden [5]. Je nach Zellstatus kann das Ausmaß der Hämoglobin-Expression variieren und bei erhöhtem Sauerstoffbedarf oder als Antwort auf oxidativen Stress/Entzündung hochreguliert werden.
... in Keratinozyten trägt es zur epidermalen Barriere gegen Umweltbelastungen bei....
Unsere Haut bildet eine hochwirksame Barriere gegen Umweltbelastungen - von Trockenheit, hoher Sauerstoffbelastung über Fremdstoffe, Mikroorganismen bis hin zur UV-Bestrahlung. Anpassung an diese Gegebenheiten haben im Laufe der Evolution die ausgeprägte mehrschichtige Struktur der obersten Hautschichte, der Epidermis geschaffen. Dieses hauptsächlich aus Keratinozyten bestehende Epithel, das bei uns Menschen unglaublich - im Mittel nur rund 0,1 mm - dünn ist [6], ist die eigentliche Barriere - die epidermale Barriere - gegen die Außenwelt. Die tiefste einzellige Schicht, das Stratum basale, besteht aus Keratinozyten, die sich aus Stammzellen heraus kontinuierlich und rasch teilen. Dadurch werden Zellen laufend nach außen in obere Schichten gedrängt, wobei sie aufhören sich zu teilen und einen Differenzierungsprozess durchlaufen, der sie innerhalb von rund einem Monat von Zellen des Stratum spinosum über Zellen des Stratum granulosum schließlich zu abgestorbenen verhornten Zellen im Stratum corneum umwandelt, die als Schuppen abgeschiefert werden (siehe dazu Abbildung 3 und auch [7]). Seit mehr als 50 Jahren wird über das Differenzierungsprogramm, die darin vom Stratum basale weg bis hin zur Verhornung involvierten Gene und die sich daraus entwickelnden Barrierefunktionen der epidermalen Schichten geforscht - viele Gene sind jedoch noch nicht entdeckt, weil es schwierig war terminal differenzierte Keratinozyten in ausreichender Menge für die Transkriptomanalyse zu erhalten.
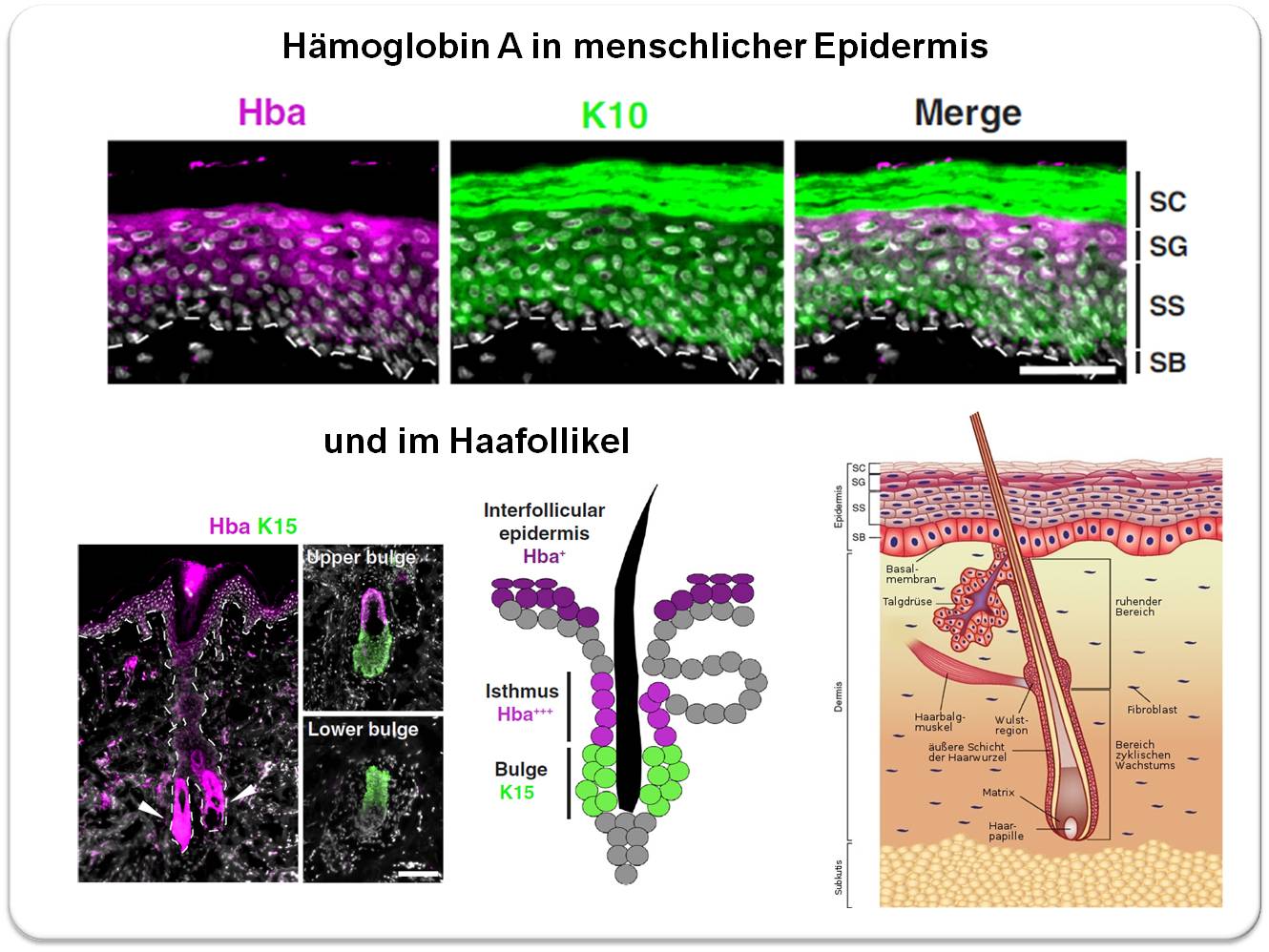
Ein japanisches Team um Masayuki Amagai (RIKEN Center for Integrative Medical Sciences, Yokohama) hat kürzlich über eine umfassende Untersuchung der Gene, die in den zunehmend differenzierten Schichten der oberen Epidermis exprimiert werden, berichtet [2]. Die Forscher haben dabei eine vergleichende Analyse der transkribierten Gene in der gesamten Epidermis und in ihren abgetrennten oberen Schichten aus Hautproben von Menschen und Mäusen durchgeführt. Ein unerwartetes Ergebnis war die Expression des Hämoglobin A Gens in der Epidermis, das in der oberen Epidermis im Vergleich zur gesamten Epidermis angereichert war. Mit immunhistochemischen Methoden wurde auch eine hohe, vom Stratum spinosum zum Stratum granulosum zunehmende Konzentration des Hämoglobin Proteins (HbA) bei Mensch und Maus nachgewiesen. Auch in den Keratinozyten von Haarfollikeln wird HbA produziert, insbesondere in der Isthmusregion, nicht aber in der Wulstregion, die Stammzellen (Marker Keratin 15) und prolifierende Keratinozyten enthält. Abbildung 3.
|
Abbildung 3. Lokalisierung von Hämoglobin A in Keratinozyten der menschlichen Epidermis und des Haarfollikels (Die Haut stammte von der Hüfte einer 62 jährigen Frau).Oben: Das lila angefärbte HbA-Protein wird mit zunehmender Differenzierung in den Zellen der oberen Schichten zunehmend stärker exprimiert, fehlt jedoch in den toten Zellen des verhornten Stratum corneum. Im Vergleich dazu wird Keratin 10 (K10) gezeigt (grün), das als Marker der Differenzierung in allen Zellen ab dem Stratum spinosum (SS) aufscheint und im toten verhornten Stratum corneum akkumuliert. Der weiße Balken entspricht einer Distanz von 0,05 mm. Unten: Im Haarfollikel ist HbA besonders stark in den Keratinozyten der Isthmus Region (d.i, zwischen Talgdrüse und Haarbalgmuskel) exprimiert, dagegen nicht im unteren Bereich der Wulstregion, der Stammzellen und proliferierende Keratinozyten (Indikator Keratin 15, grün) enthält. (Bilder aus Tahara U., et al. 2023; [2. Lizenz cc-by]). Rechts unten: Schematische Darstellung eines Haarfollikels in der Haut (Bild: A. Friedrich, https://de.wikipedia.org/wiki/Haar#/media/Datei:Anatomy_of_the_skin_de.jpg. Lizenz cc-by) SB, Stratum basale; SS, Stratum spinosum, SG, Stratum granulosum; SC, Stratum corneum. |
Wie früher erwähnt können von HbA abgespaltene Peptide antimikrobielle Aktivitäten aufweisen [5]. In Hinblick auf die ausgeprägte HbA-Expression im Isthmus des Haarfollikels könnte dies - nach Meinung der Autoren - auf den Bedarf an Stammzellschutz gegen die Invasion der Hautmikrobiota an der Follikelöffnung zurückzuführen sein.
...... und schützt vor allem gegen durch UV-Strahlung generierte reaktive Sauerstoffspezies
Die auf unsere Haut auftreffende UV-Strahlung des Sonnenlichts, besteht zu etwa 95 % aus UVA (350-400 nm) Strahlung, der Rest aus UVB (290-320 nm) Strahlung. Die energiereichere UVB-Strahlung verursacht direkte DNA-Schäden, UVA kann dagegen in viel stärkerem Maße als UVB ROS erzeugen.
In Primärkulturen menschlicher Keratinozyten und auch in einem 3D-Epidermis Modell haben die Forscher gesehen, das UVA-Bestrahlungen bereits bei der minimalen erythematischen Dosis, aber nicht UVB-Bestrahlungen die Expression von Hämoglobin hochregulierten. Gleichzeitig wurden durch die UVA-Bestrahlung Wasserstoffperoxid und andere ROS generiert, offensichtlich aber zum Großteil von Hämoglobin abgefangen/neutralisiert. Dies ging aus Versuchen hervor, in denen die Expression von Hämoglobin ausgeschaltet (mittels knockdown) war und die UV-induzierten ROS um ein Vielfaches anstiegen.
Diese Ergebnisse deuten darauf hin, dass die epidermale HbA-Expression durch oxidativen Stress induziert wird und als Antioxidans zur Barrierefunktion der Haut beiträgt; nach Meinung der Autoren stellt dies einen körpereigenen Schutzmechanismus gegen Hautalterung und Hautkrebs dar.
[1] Saha D., et al. Hemoglobin Expression in Nonerythroid Cells: Novel or Ubiquitous? International Journal of Inflammation, vol. 2014, Article ID 803237, https://doi.org/10.1155/2014/803237.
[2] Tahara U., et al. Keratinocytes of the Upper Epidermis and Isthmus of Hair Follicles Express Hemoglobin mRNA and Protein. J Invest. Dermatol. 143 (12) 2023, DOI: https://doi.org/10.1016/j.jid.2023.08.008.
[3] Zhang, F., et al. An extra-erythrocyte role of haemoglobin body in chondrocyte hypoxia adaption. Nature 622, 834–841 (2023). https://doi.org/10.1038/s41586-023-06611-6
[4] M.P.Sumi et al.,Hemoglobin resident in the lung epithelium is protective for smooth muscle soluble guanylate cyclase function. Redox Biology, 07,2023, 63, https://doi.org/10.1016/j.redox.2023.102717
[5] Olari L-R., et al., The C‑terminal 32‑mer fragment of hemoglobin alpha is an amyloidogenic peptide with antimicrobial properties. Cellular and Molecular Life Sciences (2023) 80:151. https://doi.org/10.1007/s00018-023-04795-8
[6] Lintzeri D.A. et al., Epidermal thickness in healthy humans: a systematic review and meta-analysis (2022) J. Eur.Acad.Dermatol. Venereol. 36: 1191 -1200. https://doi.org/10.1111/jdv.18123
[7] Inge Schuster, 17.07.2015: Unsere Haut - mehr als eine Hülle. Ein Überblick.
Verwandte Themen im ScienceBlog
Gottfried Schatz, 23.01.2015: Der besondere Saft
Inge Schuster, 06.09.2018: Freund und Feind - Die Sonne auf unserer Haut
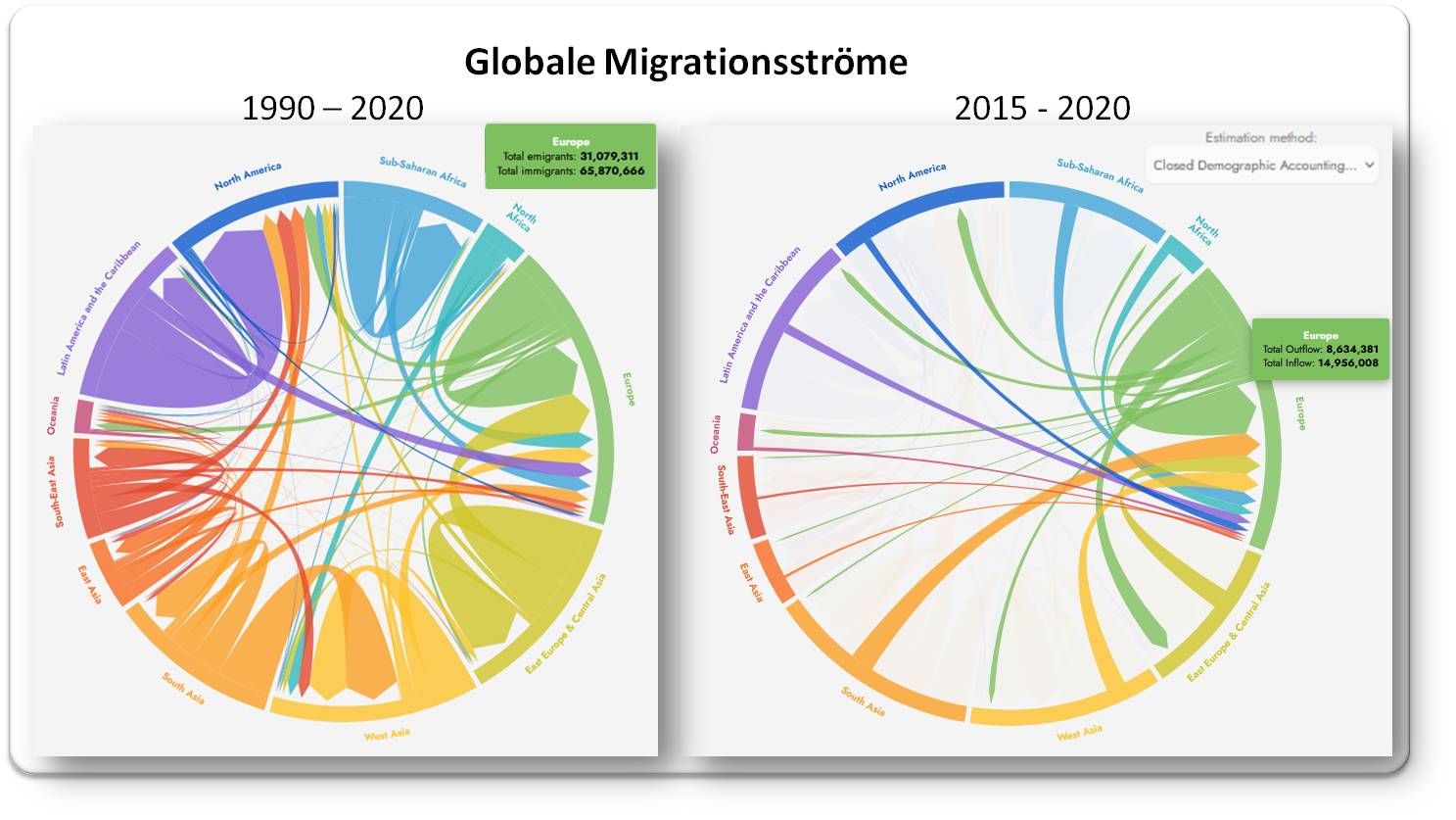
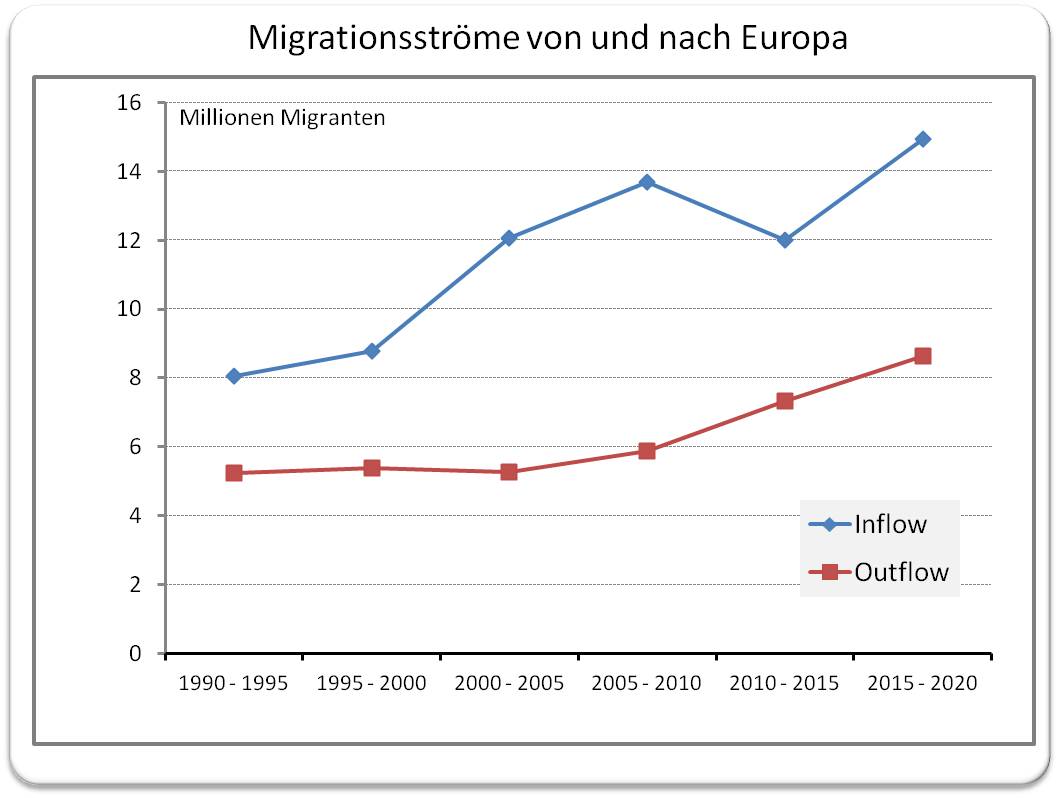
Extreme Hitze und Dürre könnten in Europa schon früher auftreten als bislang angenommen
Extreme Hitze und Dürre könnten in Europa schon früher auftreten als bislang angenommenFr, 22.12.2023 — Redaktion
Extreme Hitze und Dürre, wie sie für das Klima am Ende des 21. Jahrhunderts typisch sein werden, könnten in Europa schon früher als angenommen auftreten und dies wiederholt in mehreren aufeinander folgenden Jahren. Das ist die kürzlich publizierte Schlussfolgerung eines Forscherteams vom Max-Planck-Institut für Meteorologie (Hamburg), das eines der weltweit besten hochkomplexen Erdsystemmodelle "MPI Grand Ensemble" für seine Berechnungen einsetzte. Demnach liegt die Wahrscheinlichkeit, dass Hitze und Dürre ein Niveau erreichen, wie man es für das Ende des Jahrhunderts angenommen hatte - selbst bei einer moderaten Erwärmung -, bereits in den 2030er Jahren bei 10 %. Um 2040 könnten ganze Dekaden mit Hitzestress ihren Anfang nehmen, wobei ein warmer Nordatlantik den Wandel beschleunigen wird.*
Wenn die Erderwärmung weiter zunimmt, wird abnorme Hitze häufiger auftreten und extremer werden. Dass Hitze, die unsere bisherige Anpassungsfähigkeit deutlich übersteigt, zu erhöhter Morbidität und Sterblichkeit führt, ist erwiesen - bei den Hitzewellen 2015 in Indien und Pakistan und 2003 in Europa sind Tausende Menschen gestorben. Die Lage wird verschärft, wenn zu extremer Hitze noch hohe Luftfeuchtigkeit, mangelnde nächtliche Abkühlung oder anhaltende Trockenheit hinzukommen. Höchsttemperaturen und zunehmende Schwankungen zwischen extremer Trockenheit und immensen Niederschlägen werden zudem massive Auswirkungen auf Sozioökonomie und Ökologie haben und ganze Landstriche unbewohnbar machen. Die Bedrohung für Mensch und Umwelt wird noch ärger, wenn solche extremen Ereignisse in aufeinanderfolgenden Jahren wiederholt auftreten. Wie sich bei zunehmender Erwärmung die Wahrscheinlichkeit einer solchen mehrjährigen Abfolge von extremer Hitze und Trockenheit verändert, ist allerdings unklar, auch wie bald solche Ereignisse bereits auftreten können und wie diese Wahrscheinlichkeiten außerdem von der internen Variabilität des Klimasystems - spontan erzeugt durch Prozesse und Rückkopplungen im Klimasystem selbst — beeinflusst werden.
Das MPI-GE-Ensemble
Diese interne Variabilität des Klimasystems stellt heute eine der größten Unsicherheiten bei der Beurteilung aktueller Klimaschwankungen und bei den modellbasierten Projektionen der zukünftigen Klimaentwicklung dar. Das Max-Planck-Institut für Meteorologie (MPI-M) in Hamburg entwickelt und analysiert hochkomplexe Erdsystemmodelle, welche die Prozesse in der Atmosphäre, auf dem Land und im Ozean simulieren. Das MPI Grand Ensemble (MPI-GE) ist eines der größten derzeit verfügbaren Ensembles für die Anfangsbedingungen eines umfassenden, vollständig gekoppelten Erdsystemmodells, mit dem u.a. die Unsicherheiten der internen Variabilität des Klimasystems untersucht werden können [1].
An Hand von hundert Simulationen mit diesem Erdsystem-Modell hat nun ein Forscherteam vom MPI für Meteorologie die Wahrscheinlichkeit für das (wiederholte) Auftreten von extremen Hitze- und Dürrestress berechnet. Die Ergebnisse wurden eben im Fachjournal Nature Communications Earth & Environment publiziert [2].
Die wichtigsten Aussagen
Selbst bei einer moderaten Erwärmung wird ein noch nie dagewesenes Ausmaß an Hitze- und Dürrestress, wie dies für das Klima am Ende des Jahrhunderts typisch sein wird, in naher Zukunft europaweit möglich sein.
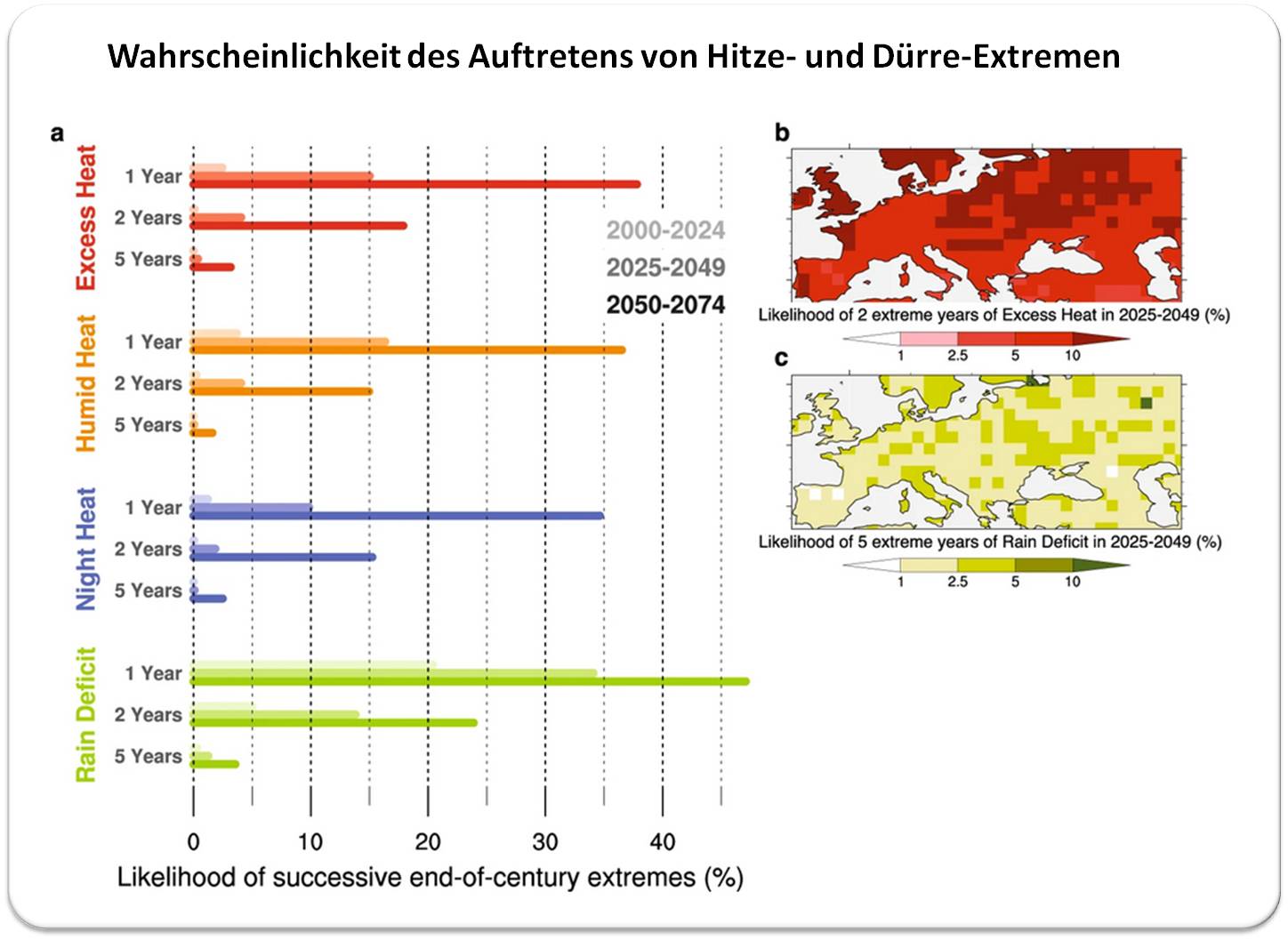
Den Projektionen zufolge werden alle untersuchten Formen von Hitzestress bereits im Jahrzehnt 2030-2039 mit einer Wahrscheinlichkeit von 1:10 ein Niveau erreichen oder übertreffen, das vor 20 Jahren praktisch unmöglich war. Darüber hinaus wird der für das Ende des Jahrhunderts typische, wiederholt in aufeinanderfolgenden Jahren auftretende extreme Hitze- und Dürrestress bereits ab 2050 mit einer Wahrscheinlichkeit von mehr als 1:10 möglich sein (dergleichen war seit Beginn der Aufzeichnungen noch nie beobachtet worden). Bis dahin werden in 20 % der Fälle zwei aufeinanderfolgende Jahre mit für das Ende des Jahrhunderts typischen Niederschlagsdefiziten prognostiziert, und es besteht ein nicht zu vernachlässigendes Risiko für 5 Jahre anhaltende extreme Dürre. Abbildung.
| Wahrscheinlichkeit, dass es in Europa zu extremen, für das Ende des Jahrhunderts typischen Hitze- Dürreperioden kommt. a) Wahrscheinlichkeit für das Auftreten extremer Hitze (rot),verbunden mit hoher Luftfeuchtigkeit, mangelnder nächtlicher Abkühlung oder anhaltender Trockenheit in einem Jahr und in den folgenden 2 oder 5 Jahren, für die Zeiträume 2000-2024 (helle Farben), 2025-2049 (mittel-helle Farben) und 2050-2074 (dunkle Farben). Wahrscheinlichkeit von extremem Hitzestress in Europa in 2 aufeinanderfolgenden Jahren (b) und von extremer Hitze und Trockenheit in 5 aufeinanderfolgenden Jahren (c) im Zeitraum 2025-2049. (Quelle: L. Suarez-Gutierrez et al., https://doi.org/10.1038/s43247-023-01075-y [2]; Lizenz cc-by.) |
Bei gleichbleibender globaler Erwärmung wird nicht nur die Häufigkeit von Hitze- und Dürreextremen zunehmen, auch deren Bandbreite wird von Jahrzehnt zu Jahrzehnt größer werden.
Diese Spanne wird so groß werden, dass Hitze- und Dürreperioden, wie sie für das Klima am Ende des Jahrhunderts typisch sein werden, in Europa bereits im Jahr 2040 Realität werden könnten. Im günstigsten Fall werden dann die Höchtstemperaturen bei Tag und Nacht im Bereich der Werte des wärmsten bislang in Europa aufgezeichneten Jahrzehnts von 2010 - 2019 liegen; im schlimmsten Fall wird die Häufigkeit und Heftigkeit der extremen Hitze- und Dürreperioden die für das Ende des Jahrhunderts typischen Ereignisse bei weitem übersteigen.
Der Einfluss des Nordatlantiks
Jedes dieser verheerenden Ereignisse könnte durch die interne Variabilität des Klimasystems früher als erwartet nach Europa gebracht werden können. Die über Dekaden zunehmende Variabilität des europäischen Hitze- und Dürrestresses wird stark von der multidekadischen Variabilität des Nordatlantiks beeinflusst. Diese zyklisch auftretende Zirkulationsschwankung der Ozeanströmungen im Nordatlantik (atlantic multidecadal variability - AMV) hat eine Periodendauer von 50 bis 70 Jahren und besitzt „warme“ und „kalte“ Phasen mit veränderten Meeresoberflächentemperaturen des gesamten nordatlantischen Beckens, die sich auf die Temperatur der Atmosphäre auswirken. Die Simulationen am MPI-GE zeigen, dass bei gleichzeitig wärmerem Nordatlantik bereits im Jahr 2030 ein Überschreiten der für das Ende des Jahrhunderts typischen Werte für einfache und komplexe Hitze- und Dürreperioden doppelt so wahrscheinlich ist, als bei einem kalten Nordatlantik.
*Diese Forschung wurde im Rahmen des vom deutschen Bundesministerium für Bildung und Forschung geförderten Projekts ClimXtreme durchgeführt und durch das Rahmenprogramm Horizon Europe der Europäischen Union unter den Marie Skłodowska-Curie Maßnahmen kofinanziert.
„ClimXtreme – Klimawandel und Extremereignisse“ hat unter Beteiligung von 39 Forscherteams in einer ersten Phase von 2019 - 2023 erforscht, wie sich der Klimawandel auf die Häufigkeit und Intensität von Extremwetterereignissen auswirkt. Nun startet die zweite Phase, die auch Zukunftsprognosen für Anwender, wie etwa für den Katastrophenschutz, Versicherungen, Landwirtschaftsverbände, Hochwasservorsorge, etc. entwickeln wird., um das Angebot ihrer Leistungen auf kommende Schäden durch Extremwetter besser abschätzen zu können. https://www.fona.de/de/aktuelles/nachrichten/2023/231207_ClimXtreme_Phase_2_b.php
[1] Das Max-Planck-Institut Grand Ensemble – ein Instrument zur Untersuchung der internen Variabilität des Klimasystems: https://mpimet.mpg.de/kommunikation/detailansicht-news/das-max-planck-institut-grand-ensemble-ein-instrument-zur-untersuchung-der-internen-variabilitaet-des-klimasystems. (abgerufen am 20.12.2023)
[2] Suarez-Gutierrez, L., Müller, W.A. & Marotzke, J. Extreme heat and drought typical of an end-of-century climate could occur over Europe soon and repeatedly. Commun Earth Environ 4, 415 (2023). https://doi.org/10.1038/s43247-023-01075-y
Klimawandel im ScienceBlog
ist ein Schwerpunktsthema mit bis jetzt knapp 50 Artikeln, die vom Überblick über Klimamodelle bis zu den Folgen des Klimawandels und den Bestrebungen zu seiner Eindämmung reichen.
Eine chrononologische Lise der Artikel findet sich unter: Klima & Klimawandel
Wie haben 15-jährige Schüler im PISA-Test 2022 abgeschnitten?
Wie haben 15-jährige Schüler im PISA-Test 2022 abgeschnitten?Sa, 16.12.2023— Inge Schuster

![]() Am 5. Dezember sind die Ergebnisse der neuen internationalen Schulleistungsstudie PISA veröffentlicht worden. PISA wird seit dem Jahr 2000 von der OECD weltweit in Mitgliedstaaten und Nicht-Mitgliedstaaten alle drei Jahre durchgeführt. Dabei wird evaluiert, inwieweit Schüler im Alter von 15 Jahren über die für eine volle gesellschaftliche Teilhabe unerlässlichen Schlüsselkenntnisse und -kompetenzen in den drei Bereichen Lesekompetenz, Mathematik und Naturwissenschaften verfügen. Das Ergebnis: Im OECD-Durchschnitt schnitten 15-Jährige wesentlich weniger gut ab als bei den PISA-Tests 2018 und vor zehn Jahren, wobei Im deutschsprachigen Raum zwar alle Bewertungen über dem OECD-35 Durchschnitt liegen, aber auf einen vhm. hohen Anteil Jugendlicher hinweisen, welche die Grundkompetenzen in den Fächern fehlen. Insgesamt gesehen bringen die Reports kaum Anhaltspunkte, wie weniger erfolgreiche Bildungssysteme verbessert werden könnten.
Am 5. Dezember sind die Ergebnisse der neuen internationalen Schulleistungsstudie PISA veröffentlicht worden. PISA wird seit dem Jahr 2000 von der OECD weltweit in Mitgliedstaaten und Nicht-Mitgliedstaaten alle drei Jahre durchgeführt. Dabei wird evaluiert, inwieweit Schüler im Alter von 15 Jahren über die für eine volle gesellschaftliche Teilhabe unerlässlichen Schlüsselkenntnisse und -kompetenzen in den drei Bereichen Lesekompetenz, Mathematik und Naturwissenschaften verfügen. Das Ergebnis: Im OECD-Durchschnitt schnitten 15-Jährige wesentlich weniger gut ab als bei den PISA-Tests 2018 und vor zehn Jahren, wobei Im deutschsprachigen Raum zwar alle Bewertungen über dem OECD-35 Durchschnitt liegen, aber auf einen vhm. hohen Anteil Jugendlicher hinweisen, welche die Grundkompetenzen in den Fächern fehlen. Insgesamt gesehen bringen die Reports kaum Anhaltspunkte, wie weniger erfolgreiche Bildungssysteme verbessert werden könnten.
Durch die Corona-Pandemie bedingt wurde die für 2021 geplante PISA-Studie (Programme for International Student Assessment) auf 2022 verschoben. Der Schwerpunkt der Erhebung lag dieses Mal auf Mathematik; untergeordnet waren die Bereiche Naturwissenschaften und Lesekompetenz (neu hinzugekommen ist ein Bereich kreatives Denken, dessen Ergebnisse aber erst 2024 berichtet werden sollen).
An der Studie haben mehr Staaten und Volkswirtschaften - 37 OECD Mitglieder, 44 Partnerländer - als je zuvor teilgenommen. Stellvertretend für die etwa 29 Millionen 15-jährigen Schüler dieser Länder haben sich rund 690 000 Schüler den Tests unterzogen. In den meisten Ländern wurden zwischen 4000 und 8 000 Schüler aus repräsentativen Stichproben getestet.
Konkret haben die Schüler während zwei Stunden Tests in zwei Bereichen (Mathematik & Naturwissenschaften oder Mathematik & Lesekompetenz) am Computer absolviert und danach in rd. 35 Minuten einen Fragebogen zu ihrem sozioökonomischen Status und zu anderen Fragen ausgefüllt. Dabei wurden Zusammenhänge zwischen Leistungen und sozioökonomischem Status, Geschlecht sowie Migrationshintergrund und auch zu den Auswirkungen der Corona-Pandemie untersucht.
Ein Mammutprojekt, was den Umfang der Ergebnisse betrifft.
Diese liegen nun in zwei insgesamt rund 1000 Seiten umfassenden Publikationen "Lernstände und Bildungsgerechtigkeit" [1] und "Learning During – and From – Disruption" [2] vor, weitere 3 Publikationen sind für 2024 geplant. Zusätzlich gibt es Zusammenfassungen für die einzelnen teilnehmenden Länder (https://www.oecd.org/publication/pisa-2022-results/country-notes/), eine Zusammenfassung "PISA 2022: Insights and Interpretations" [3] und auf 2 Seiten in Bildern "PISA 2022 key results (infographic)"[4].
Leider gibt es Ungereimtheiten bei den Aussagen und Interpretationen und es werden wenig Anhaltspunkte zur Verbesserung weniger erfolgreicher Systeme geboten.
Wie werden PISA-Tests analysiert/bewertet?
Vorweg eine kurze Zusammenfassung, um mit den Zahlen in den nächsten Abschnitten etwas anfangen zu können:
Die Testergebnisse werden einer Normalverteilung entsprechend skaliert mit Mittelwerten von etwa 500 Punkten und Standardabweichungen von etwa 100 Punkten. Bei den ersten schwerpunktmäßig getesteten Bereichen (Lesen: PISA 2000; Mathematik: PISA 2003; Naturwissenschaften: PISA 2006) haben 2/3 der Schüler demnach einen Wert zwischen 400 und 600 Punkten, 95 % der Schüler zwischen 300 und 700 Punkten erreicht.
Die verschiedenen Schwierigkeitsgrade in den Testbeispielen werden auf nach oben und unten offenen, in Kompetenzstufen unterteilten Skalen dargestellt. In Mathematik entspricht jede Kompetenzstufe einer Spanne von etwa 62 Punkten (Schwellenwerte der Kompetenzstufen in Zahlen: 1c = 233,1b = 295, 1a = 358, 2 = 420, 3 = 482, 4 = 545, 5 = 607, 6 = 669) (Tab. I.3.1 [1]). In Naturwissenschaften beträgt die Spanne etwa 75 Punkte, in Lesekompetenz ungefähr 73 Punkte.
Stufe 2 gilt als Grundkompetenzniveau, um in vollem Umfang am gesellschaftlichen Leben teilzuhaben. Schüler, die diese Stufe nicht erreichen - in Mathematik 420 Punkte, in Naturwissenschaften 410 Punkte, in Lesen 407 Punkte - werden als leistungsschwach bezeichnet, Schüler, die die Kompetenzstufen 5 oder 6 erreichen als leistungsstark.
Globale PISA-Ergebnisse 2022
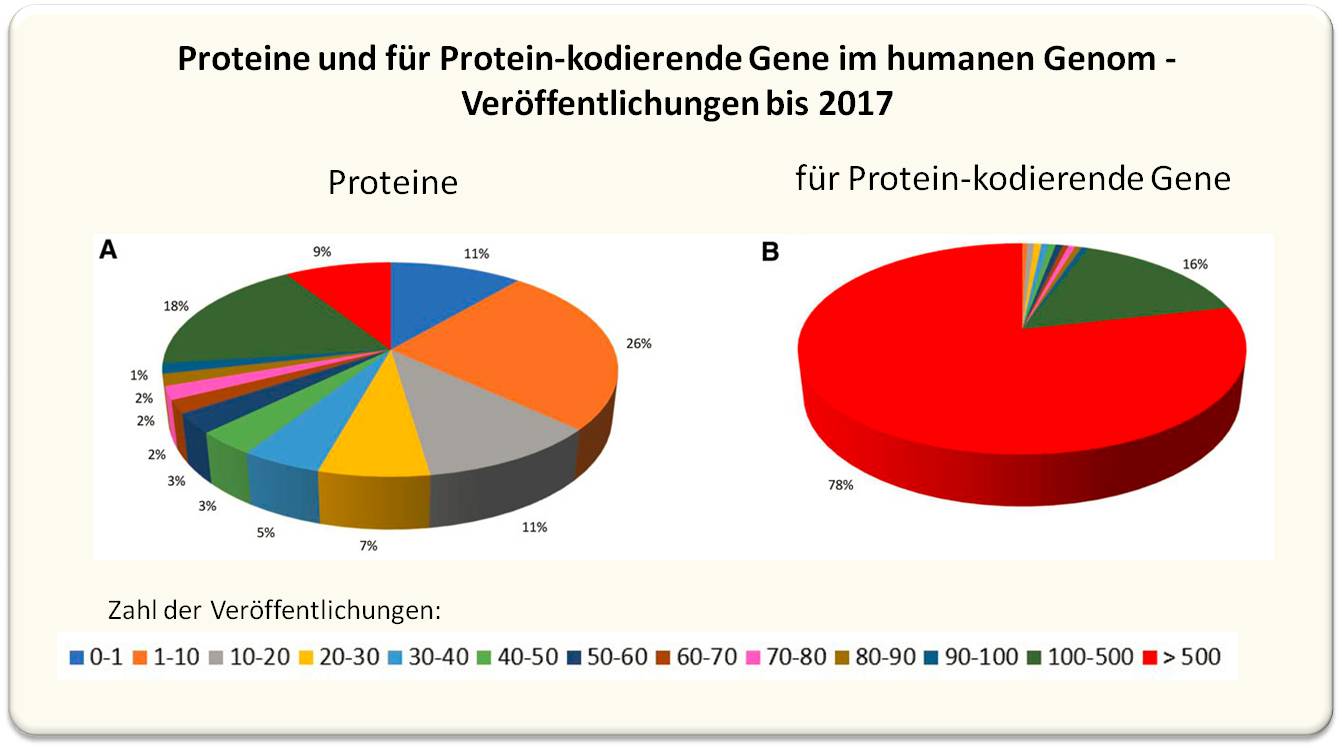
Seit dem Beginn der PISA-Erhebungen haben die durchschnittlichen Schülerleistungen weltweit in allen 3 Bereichen abgenommen. Wurde ursprünglich ein OECD-Leistungsdurchschnitt in den drei Bereichen von jeweils 500 Punkten festgelegt, so liegen nun die Durchschnitte von 35 OECD Ländern bei 472 Punkten in Mathematik, 485 Punkten in Naturwissenschaften und 472 Punkten in Lesekompetenz. Am stärksten ist der OECD-Leistungsdurchschnitt in Mathematik seit 2018 gesunken (Rückgang um fast 15 Punkte, d.i. dreimal so hoch wie jede der vorherigen Veränderungen) und in der Lesekompetenz (um 10 Punkte). Der Rückgang in den Naturwissenschaften (um 4 Punkte) fiel wesentlich geringer aus und war statistisch nicht signifikant. In den Key Results-Infografik ([1], [4]) wird der Rückgang und auch dessen Interpretation im Stil der in Zeitungen üblichen Clickbaits dargestellt (Abbildung 1).
| Abbildung 1. Durchschnittliche Punktezahl in 22 OECD-Ländern seit dem Beginn der Pisatestungen und durchschnittlicher Rückgang im Lernfortschritt seit 2018 in Schuljahren. Ursprünglich wurde der Mittelwert in den OECD-Ländern auf 500 Punkte festgelegt. Die Grafik stammt aus PISA 2022 Key Results (infographic) [4]. Lizenz: cc-by-nc-sa). Caveat: Durchschnitt aus nur 23 Ländern! Zahlen stimmen nicht mit den tatsächlichen OECD-Durchschnittswerten seit 2000 überein. |
Der Rückgang in Mathematik seit 2018 wird in den Key Results mit einem Lernrückschritt von etwa einem Dreivierteljahr gleichgesetzt, der Rückgang in Lesekompetenz mit einem halben Schuljahr (Abbildung 1). Als Basis dafür wurde ein aus aktuellen Schätzungen stammender Referenzwert von 20 Punkten für das durchschnittliche jährliche Lerntempo der 15-Jährigen in den Ländern der PISA-Teilnehmer angewandt (Kasten I.5.1 in Kapitel 5 [1]). Im selben Absatz warnt die OECD allerdings gleichzeitig davor "diesen Wert zu verwenden, um Punktezahldifferenzen in Äquivalente von Schuljahren (oder Schulmonaten) umzurechnen. Denn zum einen variiert das Lerntempo in einem bestimmten Alter von Land zu Land erheblich,..... Und zum anderen gibt es keinen Grund zur Annahme, dass das Lerntempo im Zeitverlauf konstant bleibt."
Die obige Quantifizierung des Lernrückschritts erscheint nicht nur auf Grund unterschiedlicher OECD Aussagen fragwürdig - beispielsweise wendet der Schweizer PISA-2022 Nationalbericht auf Basis anderer Schätzungen die Faustregel an, dass ungefähr 40 Leistungspunkte dem Lernfortschritt von einem Schuljahr entsprechen [5].
Wie ist nun der Abwärtstrend der OECD-Durchschnittsleistungen - insbesondere seit 2018 - zu bewerten?
In Anbetracht der hohen Punktedifferenzen zwischen leistungsschwachen und leistungsstarken Schülern innerhalb eines Landes und dem durchschnittlichen Leistungsabstand zwischen den leistungsstärksten und leistungsschwächsten Ländern (beispielsweise liegen in OECD-Ländern 153 Punkte zwischen Japan und Kolumbien, unter allen PISA-Teilnehmern 238 Punkte zwischen Singapur und Kambodscha) ist der Abwärtstrend zwar als eher klein aber als Warnsignal anzusehen:
Insgesamt gesehen sind seit 2018 die PISA-Punktzahlen im OECD-Durchschnitt sowohl bei den besonders leistungsstarken als auch bei den leistungsschwachen Schülern zwischen 2012 und 2022 zurückgegangen, d.i. der Anteil der leistungsschwachen Schüler hat zugenommen, der Anteil der leistungsstarken Schüler abgenommen.
In den OECD-Mitgliedstaaten haben 2022 rund 31 % der 15-Jährigen in Mathematik die Kompetenzstufe 2 nicht erreicht, das heißt es fehlt ihnen die Fähigkeit Mathematik in einfachen Alltagssituationen anzuwenden, 26 % erwiesen sich als leistungsschwach in der Lesekompetenz - sie können einfache Texte nicht interpretieren und 24 % sind leistungsschwach in den Naturwissenschaften, d.i. sie können nicht auf aus dem Alltag stammendes konzeptuelles Wissen zurückgreifen, um Aspekte einfacher Phänomene zu erkennen (siehe auch Tabelle 2).
Bei vielen Nicht-OECD-Mitgliedern fallen die Schülerleistungen noch negativer aus - in 18 Ländern und Volkswirtschaften - darunter 3 Balkanländern - sind mehr als 60 % der 15-Jährigen in allen drei Fächern leistungsschwach (Panama, Saudi-Arabien, Georgien, Nordmazedonien, Indonesien, Albanien, Jordanien, El Salvador, Paraguay, Guatemala, Marokko, Dominikanische Republik, Philippinen, Usbekistan, Kosovo, Kambodscha, Baku, Palästinenser-Gebiete).
Es gibt aber auch positive Entwicklungen.
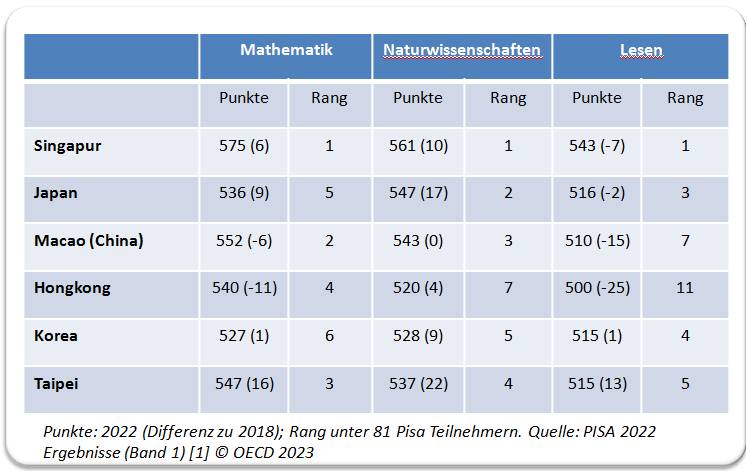
| Tabelle 1. Durchschnittliche Schülerleistungen in Punktezahlen (in Klammer: Veränderung seit 2018) in ostasiatischen Staaten/Volksgemeinschaften. Diese nehmen die Spitzenplätze aller Wertungen in den 81 getesteten Ländern/Volkswirtschaften ein. . |
Einige ostasiatische Staaten und Volkswirtschaften haben sich zu besonders leistungsstarken Gebieten entwickelt, die ihre bereits 2018 sehr hohen Leistungen z.T. noch steigern konnten und nun die Weltranglisten in allen 3 Disziplinen anführen: Singapur, Hongkong (China), Japan, Korea, Macau (China) und Chinesisch Taipei. Tabelle 1.
Was im Report nicht Platz findet: Es sind Gesellschaften, die vor allem vom Konfuzianismus geprägt sind, der eine prinzipiell positive Einstellung zum Lernen hat.
Ist die COVID-Pandemie am Leistungsabfall seit 2018 schuld?
Das wäre eine einfache Erklärung - allerdings kann der Leistungsabfall nur zum Teil durch die mit der COVID-19-Pandemie einhergehenden Schulschließungen und Problemen bei der Umstellung auf den digitalen Distanzunterricht erklärt werden. Der Zeitverlauf der PISA-Daten zeigt deutlich, dass der Abwärtstrend in zahlreichen Ländern bereits vor 2018 begonnen hatte (siehe auch Abbildung 3).
PISA-2022 in Europa
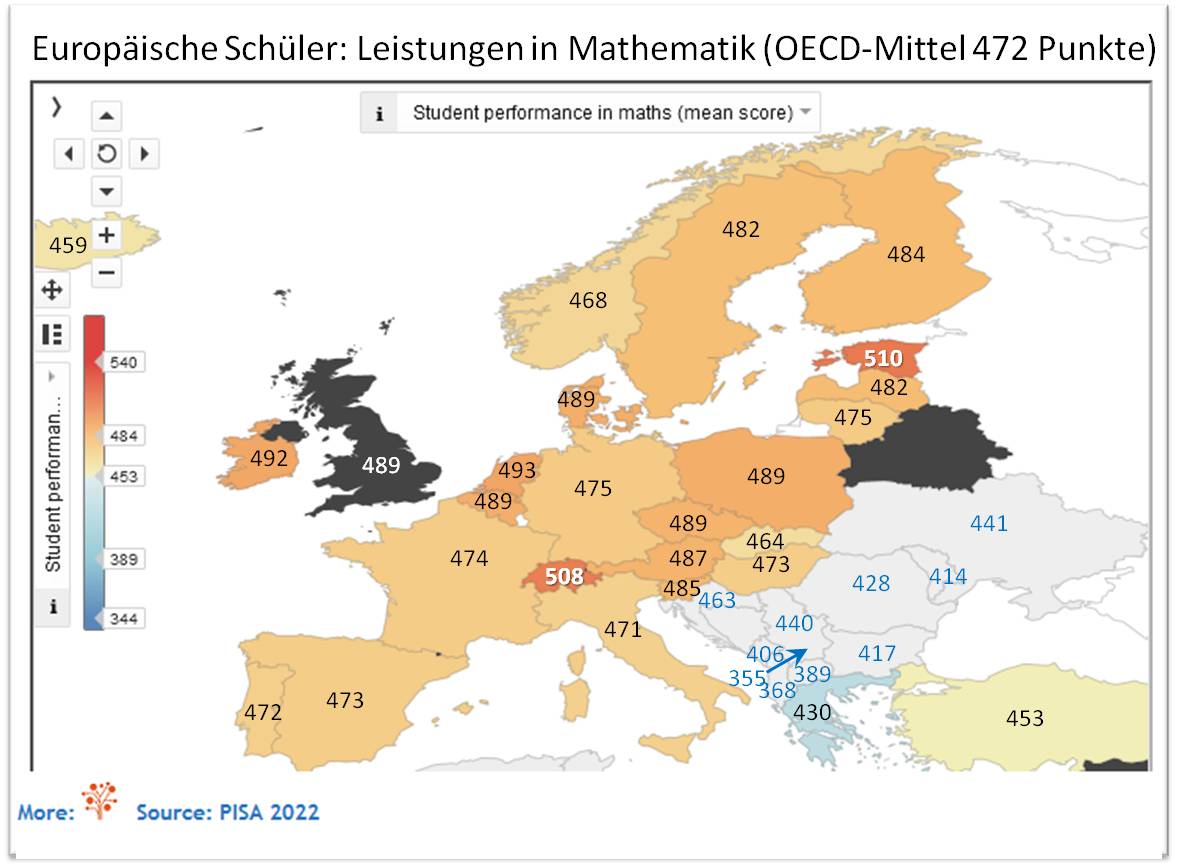
Dass Europa durch einen Bildungsvorhang in zwei Teile getrennt ist, ist nicht neu aber in den PISA 2022-Reports kein Thema. Abbildung 2 zeigt diese Spaltung am Beispiel der durchschnittlichen Mathematikleistung - die Ergebnisse in Naturwissenschaften und Lesekompetenz geben ein ganz ähnliches Bild. Auf der einen Seite steht der Block von 24 OECD-Staaten, auf der anderen Seite der Block der Balkanländer und daran angrenzend Moldawien und die Ukraine; Kroatiens Durchschnittsleistungen in allen 3 Bereichen, lassen das Land als Teil des OECD-Blocks erscheinen.
| Abbildung 2. PISA 2022: Ein Bildungsvorhang trennt die europäischen Staaten. Durchschnittsleistungen in Mathematik; schwarze Zahlen: OECD-Länder, blaue Zahlen Nicht-OECD-Länder.(Quelle: https://factsmaps.com/pisa-2022-worldwide-ranking-average-score-of-mathematics-science-and-reading/. Daten aus Table 1.B1.2.1. [1]) |
In Mathematik liegen die Durchschnittsleistungen von 24 Ländern des OECD-Blocks im Bereich der OECD-Durchschnittsleistung von 472 Punkten oder darüber und diese haben damit im Durchschnitt mindestens die Grundkompetenzstufe 2 (Spanne 420 - 482 Punkte) erreicht. Am besten schnitten Estland (510 Punkte) und knapp dahinter die Schweiz (508 Punkte) ab - 51 Punkte trennen die Nummer 1 von Island (459 Punkte), dem Land mit der niedrigsten Durchschnittsleistung, das einst mit Finnland und Schweden zu den Vorreitern im Bildungsbereich gehörte. Seit 2018 sind die Leistungen in allen europäischen OECD-Ländern gesunken, in vielen davon um mehr als 20 Punkte.
Die Mathematikleistungen des Balkanblocks liegen im Durchschnitt um 80 Punkte unter denen des OECD-Blocks, wobei der Kosovo (355 Punkte), Albanien (368 Punkte) und Nordmazedonien (389 Punkte) die schlechtesten Wertungen erhielten, im Durchschnitt Kompetenzstufe 2 nicht erreichten. Auch in Naturwissenschaften und Lesen schnitten diese und die anderen Länder des Balkanblocks ganz ähnlich ab wie in Mathematik.
Leistungstrends in Österreich und Vergleichsländern
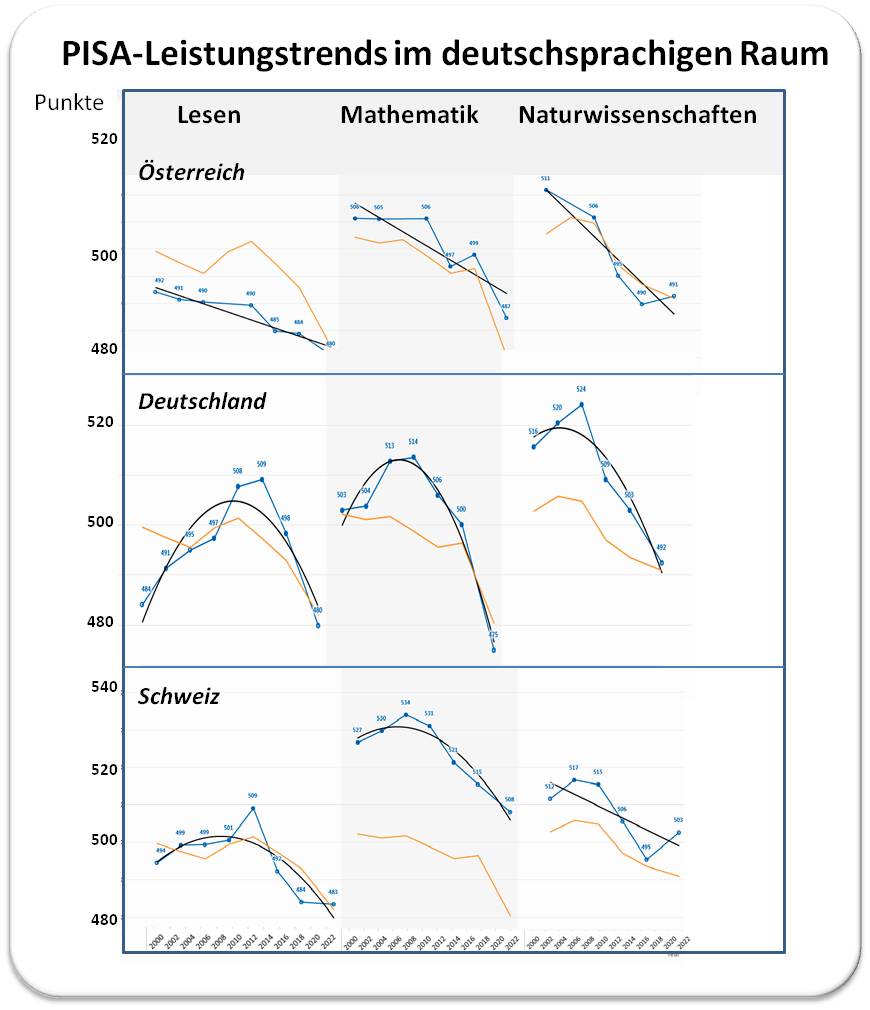
Die Durchschnittsergebnisse in Mathematik, Naturwissenschaften und Lesekompetenz fielen in Österreich, Deutschland und der Schweiz schwächer aus als in den Jahren davor. Abbildung 3.
| Abbildung 3. Wie haben Schüler in Österreich, Deutschland und der Schweiz in den drei Testbereichen seit 2000 abgeschnitten? Punkte: Durchschnittsleisung, Schwarze Linien: bester Trend, orange Linien: OECD-Durchschnittsleistung (aus nur 23 Ländern! - die Kurven stimmen nicht mit den tatsächlichen OECD-Werten seit 2000 überein.) Quelle: https://www.oecd.org/publication/pisa-2022-results/country-notes/. Lizenz cc-by-nc-sa. |
In den drei Ländern ist der Leistungsdurchschnitt in allen Bereichen seit Beginn der PISA-Tests massiv zurückgegangen, die Länder liegen dennoch über oder am OECD-Durchschnitt: über dem Durchschnitt sind in Mathematik die Schweiz um 36 Punkte, Österreich um 15 Punkte, Deutschland um 3 Punkte; in den Naturwissenschaften um 6 resp. 7 Punkte in Österreich und Deutschland und um 18 Punkte in der Schweiz und schließlich in der Lesekompetenz um jeweils 8 Punkte in Österreich und Deutschland und um 11 Punkte in der Schweiz (siehe auch Tabelle 2).
Wo liegen damit die drei Länder in der globalen Bewertung?
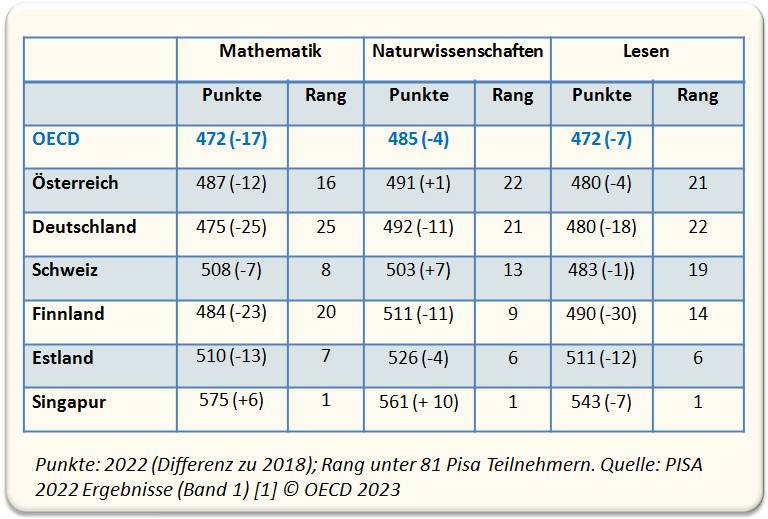
Als Vergleichsländer sollen das frühere Vorzeigeland Finnland, die derzeitige Nummer 1 in Europa, Estland und die globale Nummer 1 Singapur dienen (Tabelle 2).
Auch in Finnland und Estland sind die Leistungen seit 2018 in allen Gebieten zurückgegangen, in Singapur dagegen in Mathematik und Naturwissenschaften noch gestiegen - dort liegen diese nun um bis zu 100 Punkten über den Bewertungen unserer Länder.
| Tabelle 2. Durchschnittliche Schülerleistungen in den deutschsprachigen Ländern und Vergleichsländern. |
Leistungsschwache und leistungsstarke Schüler
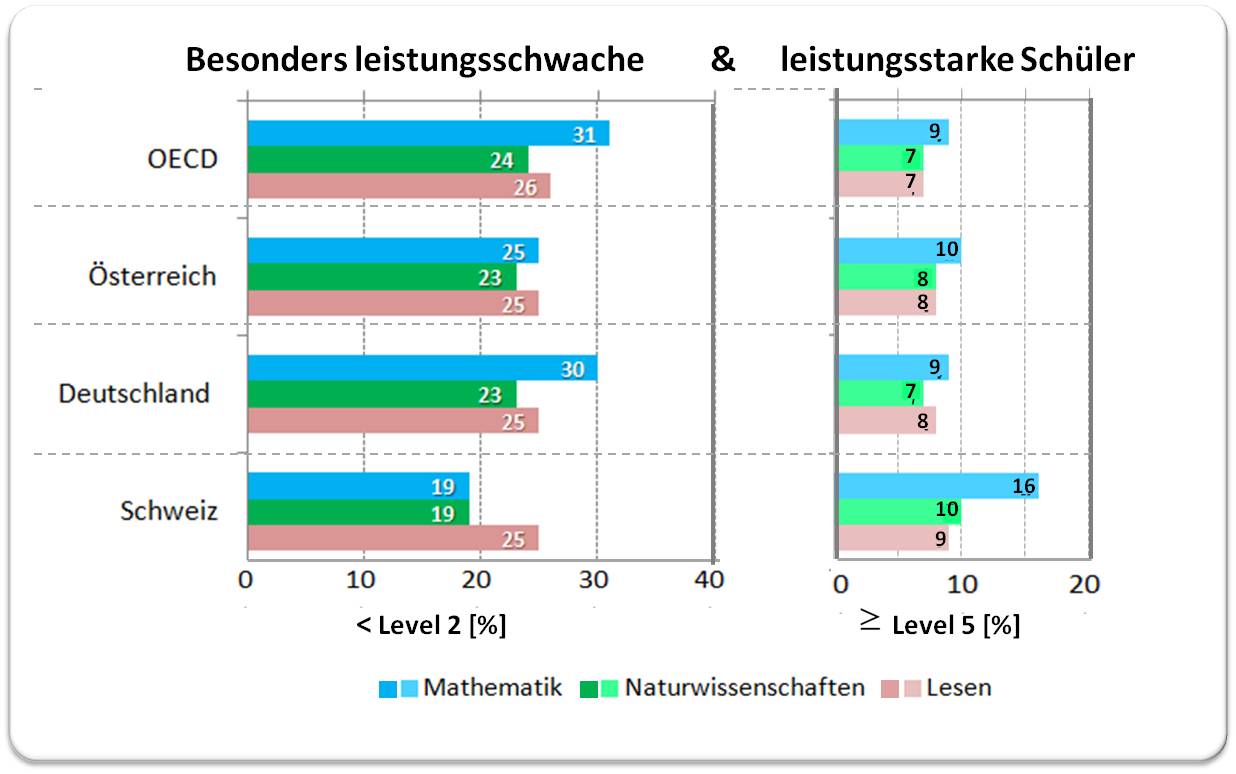
Wie bereits früher erwähnt, liegt der Anteil der Schüler die die Grundkompetenzstufe 2 nicht erreichen im OECD-Durchschnitt bei rund einem Viertel in Naturwissenschaften und in Lesekompetenz und bei fast einem Drittel in Mathematik. Etwa 9 % der Schüler erreichten die höchsten Leistungsniveaus in Mathematik und rund 7 % in Naturwissenschaften und Lesekompetenz. Der Anteil leistungsschwacher und leistungsstarker Schüler ist in Deutschland in allen Fächern etwa gleich hoch wie im OECD-Durchschnitt, schwache und starke Leseleistung liegen auch in Österreich und der Schweiz im OECD-Durchschnitt. Österreich und vor allem die Schweiz haben einen niedrigeren Anteil an sehr schwachen und einen höheren Anteil an starken Mathematik-Schülern. In Naturwissenschaften liegt Österreich etwas, die Schweiz stärker über dem OECD-Durchschnitt. Abbildung 4. Der Anteil der leistungsschwachen Schüler ist in Österreich seit 2012 um jeweils 6 % in Mathematik und in den Naturwissenschaften und um 5 % in Lesekompetenz gewachsen.
| Abbildung 4. Anteil der Schüler [%], welche die Grundkompetenzstufe 2 nicht erreichten und welche mindestens Kompetenzstufe 5 erzielten. Quelle: PISA-2022-Datenbank, Tabelle I.B1.5.1–12, I.B1.5.19, I.B1.5.20 und I.B1.5.21[1] |
Im Vergleich dazu:
Estland, die Nummer 1 in Europa weist mit 15 % in Mathematik, 10 % in Naturwissenschaften und 14 % im Lesen wesentlich niedrigere Anteile an sehr schwachen und mit 13 % in Mathematik, 12 % in Naturwissenschaften und 11% im Lesen höhere Anteile an leistungsstarken Schülern auf als die deutschsprachigen Länder.
In Mathematik nimmt Singapur die globale Spitzenposition ein: 8 % liegen unter Kompetenzstufe 2 und 41 % erreichten Stufe 5, 6. Spitzenleistungen in Mathematik erzielten auch die anderen ostasiatischen Staaten/Volkswirtschaften Taipei (32%), Macao (China) (29%), Hong Kong (27%), Japan (23%) und Korea (23%).
Insgesamt gesehen und für das weitere Berufsleben entscheidend ist der Anteil der 15-Jähringen, die in allen 3 Fächern die Grundkompetenzstufe nicht erreichen: im OECD-Raum liegt dieser bei 16,4 %, in Deutschland (16,7 %) vergleichbar hoch, in Österreich (15,5 %) kaum viel niedriger und auch in der Schweiz mit 12,4 % noch zu hoch. In Estland sind es dagegen nur 5,2 % (Tabelle 1.1[1]).
Wie lässt sich der Rückgang der Schülerleistungen seit 2018 erklären?
Wie oben bereits erwähnt - sicherlich nicht ausschließlich durch die Folgen der Corona-Pandemie. Auf keinen Fall auch durch die weltweiten, seit langem bestehenden Leistungsunterschiede: Buben in Mathematik besser als Mädchen, Mädchen in Lesekompetenz besser als Buben.
Ein wichtiger Prädikator der Schülerleistungen ist der sozioökonomische Status - ESCS -, d.i. der wirtschaftliche, soziale und kulturelle Background der Schüler (ein Index, der sich aus der höchsten Bildungsstufe der Eltern, deren höchster beruflicher Stellung und dem materiellen Wohlstand der Familie zusammensetzt). Wie auch in früheren Studien gezeigt, schneiden sozioökonomisch begünstigte Schüler besser ab als benachteiligte Schüler. Im OECD-Durchschnitt liegt beispielsweise der Leistungsunterschied von begünstigten vs. benachteiligten Schüler in Mathematik bei 93 Punkten. Der aktuelle Punkteabstand in Österreich sind 106 Punkte, in Deutschland 111 Punkte und in der Schweiz 117 Punkte - kein Unterschied zum globalen Top-Performer Singapur bei 112 Punkten.
Ein wesentlicher Unterschied liegt allerdings in der Zuwanderung von sozioökonomisch-kulturell unterschiedlich geprägten Menschen. Der Report [1] schreibt dazu "Der Anteil der Schüler mit Migrationshintergrund hat sich in den meisten PISA-Teilnehmerländern seit 2012 nicht nennenswert verändert; in 20 Ländern bzw. Volkswirtschaften ist er jedoch gestiegen und in 5 anderen gesunken. Im OECD-Durchschnitt beträgt der Anteil der 15-jährigen Schüler mit Migrationshintergrund 13 %. In 40 Ländern und Volkswirtschaften machen die Schüler mit Migrationshintergrund weniger als 5 % der Grundgesamtheit der 15-jährigen Schüler, in 11 ist er höher als 25 %."
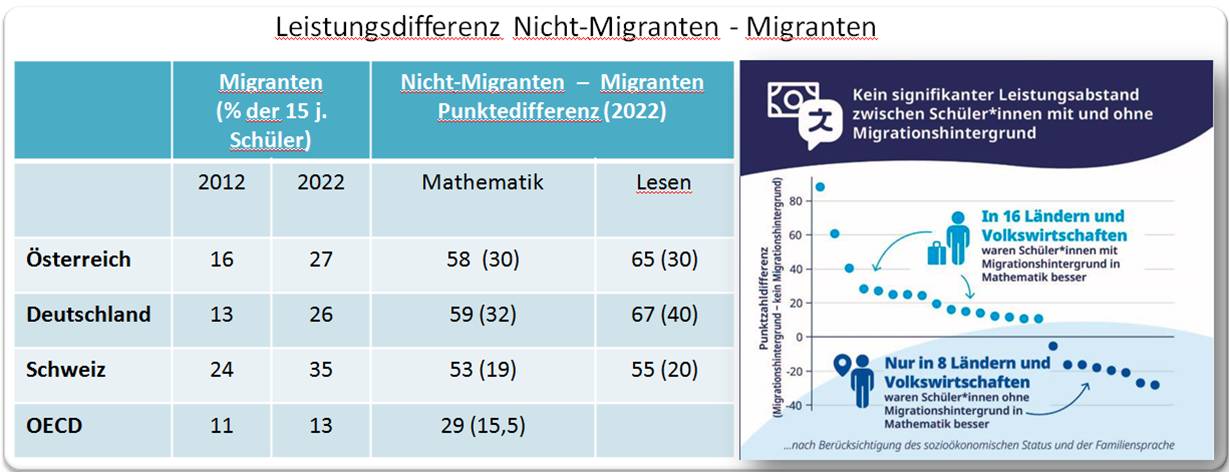
Zu diesen 11 Ländern gehören Österreich, Deutschland und die Schweiz. In Europa weisen sie die höchsten Anteile von 15-jährigen Schülern mit Migrationshintergrund auf und deren Anteil ist seit 2012 enorm gestiegen. Abbildung 5.
| Abbildung 5.Der Anteil der migrantischen Schüler ist zwischen 2012 und 2022 in den deutschsprachigen Ländern im Vergleich zum OECD-Raum enorm gestiegen. Die Leistungsdifferenz kann nur teilweise durch den unterschiedlichen sozioökonomischen Background erklärt werden. Punktedifferenz: nicht-migrantische vs migrantische Schüler- in Klammer nach Berücksichtigung der soziökonomischen Unterschiede. (Daten aus [1], Bild rechts aus Key Results Infografic [4], Lizenz: cc-by-nc-sa) |
Der PISA-Report fährt fort: "Der Anteil der sozioökonomisch benachteiligten Schüler ist im OECD-Durchschnitt unter den Schülern mit Migrationshintergrund größer (37 %) als unter denen ohne (22 %)" [1]. In den meisten Ländern schneiden daher Schüler ohne Migrationshintergrund in allen PISA-Erhebungsbereichen besser ab. Erschwerend kommt noch die Sprachbarriere hinzu - Schüler mit Migrationshintergrund sprechen zu Hause überwiegend eine andere Sprache als die Schulsprache - In Österreich, Deutschland und der Schweiz sind dies mehr als 60 %.
Die hohen Migrationsströme in die deutschsprachigen Länder haben zweifellos zu einem Rückgang der Durchschnittsleistungen beigetragen; im Vergleich dazu gibt es in der Nummer 1 in Europa, Estland, nur 8,7 % Schüler mit Migrationshintergrund, die sich zudem in den Leistungen von den anderen Schülern weniger unterscheiden.
Die mit "Kein signifikanter Unterschied zwischen Schüler*innen mit und ohne Migrationshintergrund" übertitelte Infografik in Abbildung 5 ist wiederum ein clickbait, der nicht die reale Situation, sondern bloß ein Wunschdenken der OECD vermittelt: "Wenn die Bildungspolitik sozioökonomische Benachteiligung und Sprachbarrieren bei Schüler*innen mit Migrationshintergrund ausgleicht (u. a. durch einen gezielten Einsatz von Bildungsressourcen zugunsten sozioökonomisch benachteiligter Schüler*innen mit Migrationshintergrund), können die Länder und Volkswirtschaften die Leistungen ihrer Schüler*innen mit Migrationshintergrund beträchtlich steigern."[1]
Ein wenig ermunternder Ausblick
den Andreas Schleicher, Direktor für Bildung und Kompetenzen bei der OECD, in dem Report Insights and Interpretations gibt [3]."Die Ergebnisse bieten eine Momentaufnahme der Bildungssysteme zu einem bestimmten Zeitpunkt; aber sie zeigen nicht - sie können nicht zeigen - wie die Schulsysteme zu diesem Punkt gekommen sind oder die Institutionen und Organisationen, die den Fortschritt gefördert oder behindert haben könnten. Außerdem sagen die Daten nicht wirklich viel über Ursache und Wirkung aus. Wir wissen zwar besser, was erfolgreiche Systeme tun, aber das sagt uns nicht unbedingt, wie wir weniger erfolgreiche Systeme verbessern können."[3]
Vielleicht hätte Schleicher auf die OECD-Definition des sozioökomischen Profils Bezug nehmen sollen, das durch den PISA-Index des ökonomischen, sozialen und kulturellen Status gemessen wird. Was die als Sieger im PISA-Test hervorgehenden ostasiatischen Länder auszeichnet, ist eine Weltanschauung, die auf Lernen und Leistung ausgerichtet ist. Um dazu den Lehrmeister Konfuzius zu zitieren:
Etwas lernen und mit der Zeit darin immer geübter werden,
ist das nicht auch eine Freude? [6].
[1] OECD (2023), PISA 2022 Ergebnisse (Band I): Lernstände und Bildungsgerechtigkeit, PISA, OECD Publishing, Paris, , https://doi.org/10.1787/b359f9ab-de.
[2] OECD (2023), PISA 2022 Results (Volume II): Learning During – and From – Disruption, PISA, OECD Publishing, Paris, https://doi.org/10.1787/a97db61c-en.
[3] Andras Schleicher: Insights and Interpretations: https://www.oecd.org/pisa/PISA%202022%20Insights%20and%20Interpretations.pdf
[4] PISA 2022 key results (infographic): https://www.oecd-ilibrary.org/sites/afda44bb-en/index.html?itemId=/content/component/afda44bb-en
[5] Erzinger, A. B., et al., (Hrsg.) (2023). PISA 2022. Die Schweiz im Fokus. Universität Bern. https://dx.doi.org/10.48350/187037
[6] Konfuzius-Aussprüche: https://www.aphorismen.de/zitat/3971, abgefragt am 10.12.2023
Artikel zum Thema im ScienceBlog:
Inge Schuster, 04.02.2023: Enorme weltweite Bildungsdefizite - alarmierende Zahlen auch in Europa.
Inge Schuster, 30.10.2021: Eurobarometer 516: Umfrage zu Kenntnissen und Ansichten der Europäer über Wissenschaft und Technologie - blamable Ergebnisse für Österreich
Inge Schuster, 03.10.2021: Special Eurobarometer 516: Interesse der europäischen Bürger an Wissenschaft & Technologie und ihre Informiertheit
Inge Schuster, 10.08.2017: Migration und naturwissenschaftliche Bildung
Roboter lernen die Welt entdecken
Roboter lernen die Welt entdeckenSa, 02.12.2023 — Roland Wengenmayr
Roboter können den Menschen heute bereits bei manchen alltäglichen Aufgaben unterstützen. Doch unbekannte Umgebungen oder auch kleine Abweichungen in den Aufgaben, auf die sie trainiert sind, überfordern sie. Damit sie rascher lernen, sich auf Neues einzustellen, entwickeln die Forschungsgruppen von Michael Mühlebach und Jörg Stückler am Max-Planck-Institut für Intelligente Systeme in Tübingen neue Trainingsmethoden für die Maschinen. Ihre Roboter müssen sich dabei auch im Pingpong oder Bodyflying bewähren.*
|
Anspruch und nicht Wirklichkeit: Bis Roboter so geschmeidig wie Menschen tanzen und dabei auch Bewegungen improvisieren können wie auf diesem mit KI erstellten Foto, wird es noch eine Weile dauern. Ihnen eine Art Körpergefühl zu vermitteln, ist ein Schritt in diese Richtung |
Intelligent handelnde Roboter waren schon ein Technikmythos, ehe es Maschinen gab, die diese Bezeichnung auch nur annähernd verdienten. Doch was können Roboter heute wirklich? Wie weit sind sie noch von Science-Fiction-Ikonen wie dem amüsant menschelnden C-3PO aus Star Wars entfernt? Bei der Suche auf Youtube landet man schnell bei einem Video der US-amerikanischen Roboterfirma Boston Dynamics. Darin verblüfft der humanoide Roboter Atlas mit Saltos, er rennt und hüpft mit einem Zwillingsbruder über einen anspruchsvollen Trainingsparcours oder unterstützt einen Menschen auf einem Baugerüst (Links zu Videos im Anhang; Anm. Redn.). Doch so beeindruckend leichtfüßig und fast unheimlich menschenähnlich sich diese Roboter auch bewegen können: Sie tun dies in einer vertrauten Umgebung, auf die sie trainiert worden sind. Wozu Atlas und Konsorten tatsächlich fähig wären, wenn sie sich in einem für sie komplett neuen Umfeld orientieren und selbstständig handeln müssten, das erfährt man nicht – Firmengeheimnis.
Joachim Hertzberg, wissenschaftlicher Direktor am Deutschen Forschungszentrum für Künstliche Intelligenz und Professor an der Universität Osnabrück, ist beeindruckt von der komplexen Bewegungsfähigkeit der Roboter von Boston Dynamics. Doch er wendet auch gleich ein: Würde man einem heutigen Roboter den Befehl geben, in einer ihm unbekannten Umgebung einen Auftrag selbstständig und planvoll auszuführen, und sei es nur, einen Kaffee zu holen, sähe das Resultat deutlich weniger spektakulär aus. „Das Gebiet heißt zwar künstliche Intelligenz, aber es kommt auf Intelligenz an, die wir selber für völlig unintelligent halten“, sagt Hertzberg, „also die Fähigkeit, sich in einer Umgebung vernünftig zurechtzufinden, Aufgaben zum ersten Mal zu machen, ohne sie zuvor geübt zu haben, situations- und zielangemessen zu handeln.“
Flexible Algorithmen
Einen Schritt hin zu Robotern, die auch in unbekannten Umgebungen und bei neuen Aufgaben die Orientierung behalten, machen Maschinen, die permanent lernen – und das möglichst schnell. An solchen Systemen arbeiten zwei Teams am Max-Planck-Institut für Intelligente Systeme in Tübingen. Anders als Unternehmen oder anwendungsorientierte Forschungseinrichtungen reduzieren die Forschenden die Komplexität der Aufgaben, die ihre Maschinen bewältigen müssen, um diesen zunächst elementare Aspekte der Orientierung beizubringen. Michael Mühlebachs Gruppe beschäftigt sich mit der Frage, wie Roboter durch Vorwissen über ihre eigenen physikalischen Eigenschaften, vermenschlicht ausgedrückt, eine Art Körpergefühl trainieren können. Denn dieses brauchen sie, um sich in einer unbekannten Umgebung bewegen und Befehle präzise ausführen zu können. Darum geht es auch in Jörg Stücklers Gruppe, die daran arbeitet, Robotern das Sehen beizubringen. Zum Sehen gehört, dass Roboter lernen, Objekte – ruhend und bewegt – in beliebiger Umgebung zu erkennen. Das Erkennen beschränkt sich hier allerdings auf rein physikalische Eigenschaften der Objekte, etwa ihre Größe, Form und Farbe, was allein schon eine gewaltige Herausforderung ist.
Ein permanent lernender Algorithmus steckt etwa im Tischtennisroboter Pamy, dessen Hardware weitgehend die Gruppe von Dieter Büchler am Tübinger Max-Planck-Institut entwickelt hat. Auf Veränderungen im Spiel muss Pamy nämlich flexibel reagieren. Einstweilen trainiert der einarmige Roboter mit einer Ballmaschine, um die künftige Flugbahn eines Balls richtig einschätzen zu lernen. Das Experiment befindet sich in einem Labor, in das Michael Mühlebach führt. Dort empfängt uns sein Doktorand Hao Ma in einer Geräuschkulisse, als wären wir im Trainingscamp einer Luftpumpenmannschaft gelandet.
Hao Ma muss angesichts des verblüfft dreinschauenden Gasts grinsen und deutet auf einen abgesperrten Bereich. Dort vollführt ein einzelner Roboterarm – Pamy ist einarmig – auf einer Art Podest mit einem Pingpong-Schläger laut schnaufend wilde Trockenübungen ohne Ball. Abbildung. Zwei Aluminiumrohre, verbunden mit Kunststoffgelenken, bilden Ober- und Unterarm, die Hand besteht aus einem Gelenk mit festgeschraubtem Schläger. Zu den Gelenken führen Luftschläuche, die unten im Podest an eine Batterie von Pneumatikzylindern angeschlossen sind. In den transparenten Zylindern sieht man Kolben auf und ab stampfen. Sie drücken Luft in die pneumatisch angetriebenen Gelenke hinein oder saugen sie ab und bewegen so den Arm.
|
Anspruch und nicht Wirklichkeit: Bis Roboter so geschmeidig wie Menschen tanzen und dabei auch Bewegungen improvisieren können wie auf diesem mit KI erstellten Foto, wird es noch eine Weile dauern. Ihnen eine Art Körpergefühl zu vermitteln, ist ein Schritt in diese Richtung. (Foto: Wolfram Scheible für MPG. © 2023, MPG) |
Solche luftdruckbetriebenen Muskeln erlauben eine sehr leichte Bauweise ohne Elektromotoren an den Gelenken, weshalb ein Roboterarm schnelle Bewegungen ausführen kann. Diese Konstruktion hat allerdings einen Nachteil: Der Arm federt sichtlich nach. Dieses elastische Verhalten muss die Steuerung genau kennen, um einen Tischtennisball präzise retournieren zu können. Das dafür nötige Körpergefühl erlernt Pamy gerade durch das Luft-Pingpong mithilfe von Kameras, Winkel- und Drucksensoren, die seine Bewegungen live verfolgen. Bei diesem Lernprozess kommt ein zentraler Forschungsansatz von Mühlebachs Team ins Spiel. Damit die Steuerung des Arms nicht mühsam bei null beginnen muss, wenn sie dessen Eigenschaften erlernt, hat das Team Pamy bereits mit einem einfachen physikalischen Modell programmiert. Es repräsentiert den Arm mit ideal steifen Stäben und idealisierten Gelenken. „Schwierig zu beschreiben ist aber das Verhalten der ,Muskeln‘ aus den Plastikbehältern, die sich mit Luft füllen“, erklärt Mühlebach: „Dafür setze ich maschinelles Lernen ein.“ Der dafür verwendete Algorithmus nutzt Kameraaufnahmen nur noch, um das Nachfedern der pneumatischen Bewegungen zu erlernen. Das spart viel Rechenzeit. Ohne das Vorwissen durch das physikalische Modell würde Pamy sechzehn Stunden benötigen, um eine Art Körpergefühl erlernen, sagt Mühlebach. „Mit dem Modell kriegen wir das in ungefähr einer Stunde hin.“ Roboterlernen durch physikalisches Vorwissen zu beschleunigen, ist dabei eine zentrale Strategie in Mühlebachs und Stücklers Forschung. Ma führt im Labor an eine Tischtennisplatte. Dort darf nun das Vorgängermodell des Roboterarms, der noch mit Luft-Pingpong beschäftigt ist, zeigen, was es kann. Eine drehbare Ballmaschine schießt einen Tischtennisball über die Platte, der springt in Pamys Feld einmal auf, ehe die Maschine ihn sauber zurückschlägt. Sie tut dies beeindruckend zuverlässig mit jedem neuen Ball. Ma zeigt auf vier Kameras, die oben über dem Tisch befestigt sind. Sie verfolgen die Bahn der leuchtend orangefarbenen Bälle. Ein Algorithmus hat inzwischen gelernt, die künftige Flugbahn eines Balls aus der bisherigen Bahnkurve so genau vorherzusagen, dass der Roboterarm wie ein geübter menschlicher Spieler reagiert und richtig trifft.
„In einer neuen Version der Ballvorhersage haben wir auch einbezogen, wie der Ball abgeschossen wird“, sagt Jörg Stückler. Das wäre bei einem menschlichen Gegner zwar ungleich schwieriger, doch die Erfahrung mit der Ballmaschine zeigt, wie das im Prinzip gehen könnte. Pamy kann nämlich auch auf Vorwissen über die Ballmaschine zurückgreifen. Das hat Jan Achterhold, Doktorand in Jörg Stücklers Team, dem Roboter beigebracht. Das entsprechende Modell berücksichtigt sogar, dass diese Maschine dem Ball einen Spin geben kann. Dadurch wird der Ball nach dem Aufsetzen im Feld des Roboterarms seitlich abgelenkt. Darauf muss Pamy sofort reagieren, was für den Roboter eine große Herausforderung ist.
Härtetest Bodyflying
Als Modell der Ballmaschine verwendeten Achterhold und Stückler dabei ein Grey-Box-Modell. Stückler erklärt, dies sei ein Zwischending zwischen einem Blackund einem White-Box-Modell. Das Black-Box-Modell steht für Maschinenlernen ohne jegliches Vorwissen, also mühsames Ausprobieren. Ein White-Box-Modell wäre das Gegenteil: ein unveränderlich programmiertes physikalisches Modell, das nicht lernfähig ist. In einer einfachen, mechanisch idealen Welt würde das auch funktionieren, denn der Ablauf inklusive Flugbahn ließe sich exakt berechnen. Doch bei einer echten Ballmaschine treten immer Effekte auf, an denen das unflexible White-Box-Modell scheitern würde. Das Team um Achterhold setzt daher auf ein physikalisch vorgebildetes Maschinenlernen. Zu diesem Zweck entwarfen die Forschenden zunächst ein physikalisches Modell und kombinierten es mit einem ausgefeilten Lernalgorithmus, der das System befähigt, die realen Eigenschaften der Ballmaschine zu erlernen. Das Team nutzte also die Vorteile des Black- und des White-Box-Ansatzes. „Deshalb wird der Ansatz Grey Box genannt“, erklärt Stückler. Im Gespräch mit den Roboterforschern wird immer wieder deutlich, welche Herausforderungen scheinbar simple Aktionen, die wir Menschen unbewusst und selbstverständlich ausführen, für die Robotik darstellen. Dabei will Michael Mühlebach es wirklich wissen. „Ich bin fasziniert vom Bodyflying, von der Akrobatik, den Drehungen und Kunststücken“, sagt er lachend: „Und da dachte ich: Es wäre doch super, das mit Robotern zu machen!“ Beim Bodyflying, auch Indoor Skydiving genannt, schweben Menschen im starken Luftstrom eines senkrecht nach oben gerichteten Windkanals. Wie beim Fallschirmsprung müssen sie lernen, wie sie ihr Flugverhalten auf dem Luftpolster durch Veränderungen ihrer Körperhaltung und damit der Aerodynamik gezielt steuern können.
Das soll in Tübingen nun ein leichter Flugroboter, kaum größer als eine Hand, über einem Miniwindkanal erlernen. Doktorand Ghadeer Elmkaiel tüftelt in seinem Labor gerade an dem selbst entwickelten Windkanal, der mit sechs kreisförmig angeordneten Propellern einen möglichst gleichmäßigen Luftstrom erzielen soll. Über der öffnung des Windkanals befindet sich eine Haltevorrichtung für den kleinen Flieger. Beim Training löst sich der Roboter davon und versucht, ohne Verbindung zu einem Computer zu schweben. Dabei soll er nach und nach vorgegebene Flugfiguren erlernen. Noch ist es nicht so weit, aber auch hier soll das Vorwissen eines einfachen physikalischen Modells den Lernprozess des Flugroboters beschleunigen.
Was Mühlebachs Team bei dem Härtetest der Orientierungsfähigkeit von Robotern lernt, könnte auch in ganz anderen Bereichen Anwendung finden – etwa bei intelligenten Stromnetzen. Sie sollen die Stromproduktion und -verteilung möglichst gut an den jeweiligen Bedarf anpassen. Das wird beim Ausbau dezentraler Windkraftund Solaranlagen, deren Stromproduktion zudem vom Wetter abhängt, immer wichtiger und anspruchsvoller. In einem solchen Netzwerk gibt es ebenfalls Elemente, die sich gut durch physikalische Modelle beschreiben lassen, und solche, deren Verhalten nur durch Lernerfahrung vorhersagbar wird. In die erste Kategorie fallen etwa Großkraftwerke, deren Stromproduktion sich physikalisch gut modellieren lässt. Der Strommarkt und das Verhalten der Endverbraucher hingegen sind erst durch Erfahrung, zum Beispiel über Jahreszeiten, vorhersagbar. „Dort gibt es ein Riesenpotenzial für maschinelles Lernen“, sagt Mühlebach. Doch zurück zur Orientierung. Jörg Stücklers Gruppe arbeitet zum Beispiel an der Weiterentwicklung einer Technik, welche Kameradaten kombiniert mit den Daten von Beschleunigungssensoren, wie sie auch in Smartphones eingebaut sind. Die Beschleunigungssensoren geben einem Roboter sozusagen einen Gleichgewichtssinn, erklärt Stückler. Durch die Kombination mit Kameradaten soll der Roboter ein Wissen darüber entwickeln, wie sein realer Körper auf einen Befehl reagiert. Soll er zum Beispiel losfahren, braucht er ein Gefühl dafür, dass er seine Masse zunächst auf die vorgegebene Geschwindigkeit beschleunigen muss. Dieses Körpergefühl entwickelt der Roboter viel schneller, wenn er mit einem einfachen physikalischen Modell von sich programmiert wurde.
Wenn ein Roboter mit Gegenständen hantieren soll, braucht er nicht nur ein gutes Gefühl für seine eigenen Bewegungen, er braucht auch eine Vorstellung von den Objekten und ihren Eigenschaften. Dass er diese allein durch Beobachtungen erkennen kann, ist das Ziel von Doktorand Michael Strecke. Da die Kameradaten verrauscht, also unscharf sind, lässt sich aus ihnen nicht einfach die Form oder die Größe eines Objekts herauslesen. Beobachtet der Roboter allerdings, wie eine immer wieder geworfene Kugel gegen eine Wand dotzt, zurückprallt und zu Boden fällt, lernt er trotzdem etwas über die Eigenschaften der Kugel. Er begreift allmählich, wie groß die Kugel ist und dass sie daher auf eine bestimmte Weise zurückprallt. So lernt er allein durch die Anschauung einzuschätzen, wie sich ein solches Objekt voraussichtlich verhalten wird.
Grundsätzlich lässt sich also aus dem reinen Beobachten des mechanischen Kontakts eines Objekts mit einem anderen auf deren Eigenschaften schließen. Kleinkinder lernen auf diese Weise, wenn sie Gegenstände herumschmeißen und diese beobachten. Wenn Computer sehen, funktionierte diese Kontaktmethode bislang nur für steife Objekte und auch nur für solche mit sehr einfacher Geometrie. Strecke und Stückler ist es jetzt mit einer neuen Optimierungsmethode gelungen, auch das maschinelle Erlernen komplexerer Formen voranzubringen. Sie veranschaulichen dies mit einem etwas absurden Beispiel: Eine Maschine beobachtet ein Objekt, das auf ein anderes fällt, und hält es zunächst für eine Kuh. Im Laufe mehrerer Zusammenstöße der beiden Objekte verwandelt sich die Kuh in der Maschinenwahrnehmung allmählich in eine Art „Badeentenschwimmring“, der wie bei einem Wurfringspiel mit seinem Loch in der Mitte auf einen Stab fällt. Was weit hergeholt erscheint, entspricht einer Situation, in der auch Menschen sich erst einmal völlig neu orientieren müssen. Roboter stehen hier noch ganz am Anfang, sozusagen im Stadium eines Kleinkinds, für das jedes Objekt in seiner Umwelt ganz neu ist. Mit ihren neuen Trainingsmethoden wollen Jörg Stückler und Michael Mühlebach den Maschinen helfen, sich in unbekannten Situationen schneller zu orientieren. Doch der Weg bis zu einem C-3PO, der sich in einem Star-Wars-Abenteuer bei seinem Begleiter R2-D2 beschwert, dass sie schon wieder in ein Schlamassel geraten seien, dürfte noch recht weit sein.
* Der kürzlich im Forschungsmagazin 3/2023 der Max-Planck Gesellschaft unter dem Titel "Roboter entdecken die Welt" erschienene Artikel https://www.mpg.de/20899916/MPF_2023_3.pdf wird - mit Ausnahme des Titels in praktisch unveränderter Form im ScienceBlog wiedergegeben. Die MPG-Pressestelle hat freundlicherweise der Veröffentlichung von Artikeln aus dem Forschungsmagazin auf unserer Seite zugestimmt. (© 2023, Max-Planck-Gesellschaft)
Videos zu Boston Dynamics
Blue Light Technology: Boston Dynamics' Atlas Bot SHOCKS Investors (2023). Video 8:37 min. https://www.youtube.com/watch?v=QKUosKzUCf8
Boston Dynamics: Do You Love Me? (2021). Video 2:54 min. https://www.youtube.com/watch?v=fn3KWM1kuAw&t=138s
Atlas Gets a Grip | Boston Dynamics (2023). Video 1:20 min. https://www.youtube.com/watch?v=-e1_QhJ1EhQ
Ähnliche Inhalte im ScienceBlog:
Paul Rainey, 02.11.2023: Können Mensch und Künstliche Intelligenz zu einer symbiotischen Einheit werden?
Inge Schuster, 12.12.2019: Transhumanismus - der Mensch steuert selbst seine Evolution
Georg Martius, 09.08.2018: Roboter mit eigenem Tatendrang
Francis S. Collins, 26.04.2018: Deep Learning: Wie man Computern beibringt, das Unsichtbare in lebenden Zellen zu "sehen".
Ilse Kryspin-Exner, 31.01.2013: Assistive Technologien als Unterstützung von Aktivem Altern.
Vor der Weltklimakonferenz COP28: durch Landnutzung entstehende Treibhausgas-Emissionen werden von Ländern und IPCC unterschiedlich definiert
Vor der Weltklimakonferenz COP28: durch Landnutzung entstehende Treibhausgas-Emissionen werden von Ländern und IPCC unterschiedlich definiertFr, 24.11.2023 — IIASA
Der Sektor Landnutzung, Landnutzungsänderung und Forstwirtschaft spielt eine wesentliche Rolle bei der Erreichung der globalen Klimaziele - allerdings klafft eine beträchtliche Lücke darin, wie in diesem Sektor die Emissionen durch globale wissenschaftsbasierte Modellierungen abgeschätzt werden und in welcher Weise die Staaten diese berichten. In einer neuen Studie zeigt ein u.a. am Internationalen Institut für Angewandte Systemanalyse (IIASA, Laxenburg bei Wien) arbeitendes Forscherteam auf, wie sich die Benchmarks für die Minderung der Emissionen ändern, wenn die Szenarien des Weltklimarats (IPCC) aus der Perspektive der nationalen Bestandsanalysen bewertet werden; demnach sollten die Netto-Null-Ziele bis zu fünf Jahre früher erreicht werden, und die kumulativen Emissionen bis zum Netto-Null-Ziel um 15 bis 18 % weniger betragen.*
Eine effektive Landnutzung, sei es für Landwirtschaft, Wälder oder Siedlungen, spielt eine entscheidende Rolle bei der Bewältigung des Klimawandels und bei der Erreichung künftiger Klimaziele. Strategien zur Abschwächung des Klimawandels im Bereich der Landnutzung sehen den Stopp der Entwaldung und eine bessere Waldbewirtschaftung vor. Den Staaten ist die Bedeutung des Sektors Landnutzung, Landnutzungsänderung und Forstwirtschaft (Land use, land use change and forestry: LULUCF) bewusst. 118 von 143 Staaten haben die Reduzierung der durch Landnutzung entstehenden Emissionen und deren Entfernung aus der Atmosphäre in ihre national festgelegten Verpflichtungen (Nationally Determined Contributions - NDCs) aufgenommen; diese NDCs sind das Herzstück des Pariser Abkommens zur Erreichung der langfristigen Klimaziele.
Unterschiedliche Schätzungen der Land-basierten Emissionen
Wie eine neue, im Fachjournal Nature veröffentlichte Studie zeigt, differieren die Schätzungen der von Landnutzung ausgehenden aktuellen Emissionen, wie sie auf Grund wissenschaftsbasierter Untersuchungen (Modellen der IPCC-Sachstandsberichte) erhoben werden und wie sie Staaten in nationalen Treibhausgasbilanzen (Treibhausgasinventaren) berichten [1]. Der Grund liegt darin, dass unterschiedlich definiert wird, was als "bewirtschaftetes" Land und was als vom Menschen durch Landnutzung verursachte (anthropogene) und aus der Atmosphäre entfernte Emissionen angesehen wird; die Studie zeigt auch, wie sich die globalen Benchmarks der Emissionsminderung ändern, wenn mit den wissenschaftlichen Klimamodellen die LULUCF-Flüsse aus der Perspektive der nationalen Treibhausgasbilanzen abgeschätzt werden. Um Fortschritte im Pariser Klimaabkommen zu bewerten, ist es nach Ansicht des Forscherteams notwendig Gleiches mit Gleichem zu vergleichen - dabei müssen die Staaten ehrgeizigere Klimaschutzmaßnahmen ergreifen, wenn sie ihre nationalen Ausgangspunkte mit globalen Modellen vergleichen.
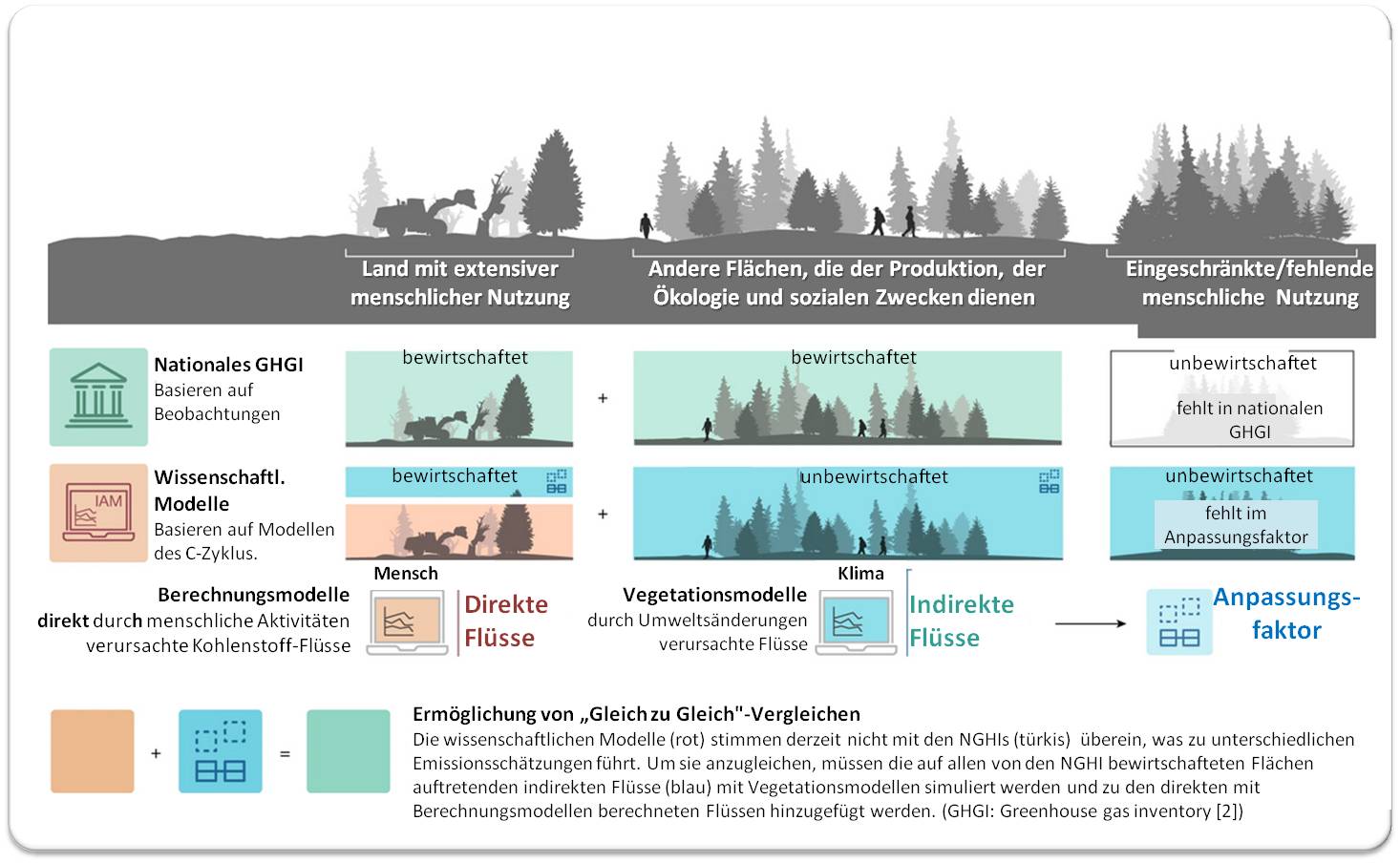
"Die Staaten schätzen ihre LULUCF-Flüsse (Emissionen und Entnahme aus der Atmosphäre) auf unterschiedliche Weise. Direkte Flüsse sind das Ergebnis direkter menschlicher Eingriffe, wie beispielsweise in der Landwirtschaft oder der Holzernte. Die Modelle in den Bewertungsberichten des Weltklimarates (IPCC) verwenden diesen Bilanzierungsansatz, um das verbleibende Kohlenstoffbudget und den Zeitplan für das Erreichen von Netto-Null-Emissionen zu bestimmen. Indirekte Flüsse sind die Reaktion des Bodens auf indirekte, vom Menschen verursachte Umweltveränderungen, wie z. B. die Zunahme des atmosphärischen CO2 oder die Stickstoffablagerung, die beide die Kohlenstoffentnahme aus der Atmosphäre erhöhen", erklärt Giacomo Grassi, ein Mitautor der Studie und Forscher am Joint Research Centre der Europäischen Kommission.
Grassi weist darauf hin, dass es praktisch nicht möglich ist, direkte und indirekte Flüsse durch Beobachtungen wie nationalen Waldinventaren oder Fernerkundungen voneinander zu separieren. Daher folgen die nationalen Methoden zur Treibhausgasinventarisierung Normen, die anthropogene Flüsse anhand eines landbasierten Ansatzes definieren, wobei alle auf bewirtschafteten Flächen auftretenden Flüsse als anthropogen gelten. Im Gegensatz dazu werden die Treibhausgasflüsse auf unbewirtschafteten Flächen nicht in die Berichterstattung einbezogen. Abbildung.
Eine Lücke von 4 - 7 Gigatonnen CO2
Weltweit ergibt sich zwischen den Berechnungsmodellen und den Treibhausgasbilanzen der Länder ein Unterschied von etwa 4 - 7 Gigatonnen CO2 oder rund 10 % der heutigen Treibhausgasemissionen, allerdings variiert dieser Unterschied von Land zu Land.
| Abbildung. Flüsse aus Landnutzung, Landnutzungsänderung und Forstwirtschaft (LULUCF): Anpassung von konventionellen wissenschaftlichen Modellen und nationalen Treibhausgasbilanzen. Abweichungen ergeben sich aus den Unterschieden, welche Flächen als bewirtschaftet gelten und ob Flüsse im Zusammenhang mit Umwelt- und Klimaveränderungen einbezogen werden.NGHGI: nationales Treibhausgasinventar [2].(Bild modifiziert aus Gidden et al., [1], Lizenz cc-by) |
Die Forscher haben die wichtigsten Klimaschutz-Benchmarks anhand des Bestandsaufnahme-basierten LULUCF-Ansatzes bewertet. Sie sind zu dem Ergebnis gelangt, dass zur Erreichung des langfristigen Temperaturziels des Pariser Abkommens von 1,5 °C die Netto-Null-Emissionen ein bis fünf Jahre früher als bisher angenommen erreicht werden müssen, die Emissionssenkungen bis 2030 um 3,5 bis 6 % stärker ausfallen und die kumulativen CO2-Emissionen um 55 bis 95 Gt CO2 geringer sein müssen. Das Forscherteam betont, dass die Ergebnisse nicht im Widerspruch zu den vom IPCC festgelegten Benchmarks stehen, sondern die gleichen Arten von Benchmarks mit einem Bestandsaufnahme-basierten Ansatz bewertet werden.
"Um die globale Temperaturreaktion auf anthropogene Emissionen zu berechnen, verwenden die IPCC-Sachstandsberichte direkte, landbasierte Emissionen als Input und beziehen die indirekten Emissionen aufgrund von Klima- und Umweltreaktionen in ihre physikalische Klimaemulation ein. In unserer Analyse machen wir deutlich, dass wir diese beiden Arten von Emissionen getrennt betrachten. Das Klimaergebnis jedes der von uns bewerteten Szenarien bleibt dasselbe, aber die Benchmarks - betrachtet durch die Linse der nationalen Konventionen zur Bilanzierung von Treibhausgasinventaren - verschieben sich. Ohne Anpassungen könnten die Länder in einer günstigeren Lage erscheinen, als sie tatsächlich sind", erklärt Thomas Gasser, Mitautor der Studie und leitender Forscher in den beiden IIASA-Programmen Advancing Systems Analysis und Energy, Climate, and Environment.
"Unsere Ergebnisse zeigen die Gefahr auf, Äpfel mit Birnen zu vergleichen: Um das Pariser Abkommen zu erreichen, ist es entscheidend, dass die Länder das korrekte Ziel anstreben. Wenn Länder bei nationaler Bilanzierung modellbasierte Benchmarks anstreben, werden sie das Ziel verfehlen", sagt Matthew Gidden, Studienautor und leitender Forscher im IIASA-Programm Energy, Climate, and Environment.
Im Vorfeld der UN-Klimakonferenz COP28
(die vom 30. November - 12 Dezember 2023 in Dubai stattfindet) und der ersten globalen Bestandsaufnahme - ein Prozess, der es den Ländern und anderen Interessengruppen ermöglicht zu sehen, wo sie gemeinsam Fortschritte bei der Erfüllung der Ziele des Pariser Abkommens machen und wo nicht - drängen die Forscher auf detailliertere nationale Klimaziele. Sie empfehlen separate Ziele für den Klimaschutz an Land, getrennt von Maßnahmen in anderen Sektoren.
"Länder können Klarheit über ihre Klimaziele schaffen, indem sie ihre geplante Nutzung des LULUCF-Sektors getrennt von Emissionsreduktionen in anderen Sektoren kommunizieren. Während Modellierer und Praktiker zusammenkommen können, um die Vergleichbarkeit zwischen den globalen Pfaden und den nationalen Bilanzierungen zu verbessern, ist es wichtig die Botschaft, dass in diesem Jahrzehnt erhebliche Minderungsanstrengungen erforderlich sind, nicht in den Details der technischen Berichterstattung untergehen zu lassen", schließt Gidden.
[1] Gidden, M., Gasser, T., Grassi, G., Forsell, N., Janssens, I., Lamb, W., Minx, J., Nicholls Z., Steinhauser, J., Riahi, K. (2023). Aligning climate scenarios to emissions inventories shifts global benchmarks. Nature DOI: 10.1038/s41586-023-06724-y
[2] FAO elearning academy: The national greenhouse gas inventory (NGHGI) for land use. https://elearning.fao.org/course/view.php?id=650
*Die Presseaussendung "Mind the gap: caution needed when assessing land emissions in the COP28 Global Stocktake" https://iiasa.ac.at/news/nov-2023/mind-gap-caution-needed-when-assessing-land-emissions-in-cop28-global-stocktake ist am 22. November 2023 auf der IIASA Website erschienen. Der Artikel wurde von der Redaktion möglichst wortgetreu übersetzt und mit Untertiteln ergänzt. IIASA hat freundlicherweise der Veröffentlichung der von uns übersetzten Inhalte seiner Website und Presseaussendungen in unserem Blog zugestimmt.
Gentherapie der Duchenne-Muskeldystrophie (DMD) - ein Rückschlag
Gentherapie der Duchenne-Muskeldystrophie (DMD) - ein RückschlagFr 17.11.2023 — Ricki Lewis
Tausende Erkrankungen werden durch schadhafte Gene ausgelöst. Bereits vor Jahrzehnten gab es erste Ansätze, um diese Gene durch Gentherapie - reparierte Gene oder intaktes genetisches Material - auszutauschen; man erwartete damit viele der zumeist ererbten Krankheiten nicht nur behandeln, sondern auch wirklich heilen zu können. Obwohl enorme Anstrengungen zur Umsetzung dieser Strategie gemacht und mehrere Tausende klinische Studien dazu durchgeführt wurden, ist der Erfolg bescheiden - von der FDA wurden bislang 6 gentherapeutische Produkte zugelassen - und immer wieder von Rückschlägen bedroht geblieben. Über enttäuschte Hoffnungen und den jüngsten Rückschlag bei einer Gentherapie von Duchenne Muskledystrophie berichtet die Genetikerin Ricki Lewis.*
Im letzten Kapitel meines 2012 erschienenen Buches The Forever Fix: Gene Therapy and the Boy Who Saved It, habe ich prophezeit, dass sich diese Technologie bald weit über die Welt der seltenen Krankheiten hinaus ausbreiten würde. (Anm. Redn.: von einer seltenen Krankheit sind weniger al 0,5 Promille der Bevölkerung betroffen.)
Ich war zu optimistisch. Es hat sich herausgestellt, dass die Gentherapie keinen großen Einfluss auf die Gesundheitsversorgung hatte, da sie nur einigen wenigen Menschen mit seltenen Krankheiten extrem teure Behandlungen offeriert. Wir sind immer noch dabei zu lernen, welche Folgen es haben kann, wenn Millionen von veränderten Viren in einen menschlichen Körper eingeschleust werden. Können sie heilende Gene liefern, ohne eine überschießende Immunreaktion auszulösen?
Ein Bericht im New England Journal of Medicine vom 28. September 2023 beschreibt einen jungen Mann mit Duchenne-Muskeldystrophie (DMD), der nur wenige Tage nach einer Gentherapie starb [1]. Die Einzelheiten erinnern in beunruhigender Weise an den berühmten Fall von Jesse Gelsinger, der im September 1999 an einer überschießenden Immunantwort auf eine experimentelle Gentherapie starb.
Jesse war 19 Jahre alt und litt an einer Defekt im Harnstoffzyklus (Ornithintranscarbamylase-Defizienz). Er wurde an einem Montag behandelt und starb am Freitag infolge eines Multiorganversagens. Die Viren, mit denen die Gene übertragen wurden, hatten ihr Ziel verfehlt und drangen in nicht dafür vorgesehene Zelltypen der Leber ein, was den Alarm der Immunabwehr auslöste.
Es gibt erst wenige Gentherapie-Zulassungen
Auch wenn bereits einige hundert Menschen dem Ende 2017 zugelassenen Luxturna das Sehvermögen verdanken, ist die Liste der Gentherapien, die die Hürden der FDA nehmen konnten, noch kurz geblieben. Zu den enorm hohen Kosten und den kleinen Märkten kommt dazu, dass man die Reaktionen der Patienten nicht vorhersehen konnte. Man braucht ein besseres Verfahren, um Patienten zu identifizieren, die am ehesten auf eine bestimmte Behandlung ansprechen.
Denken wir an Zolgensma, eine Gentherapie, die 2019 zur Behandlung der spinalen Muskelatrophie zugelassen wurde. Betroffene Kinder überleben selten das Säuglingsalter. Deshalb waren die Videos eines kleinen Mädchens namens Evelyn, das nach der Behandlung mit Zolgensma tanzte, so erstaunlich - ihre Schwester war an der Krankheit gestorben. Ein aktuellerer Fall war ein Säugling, der 2021 die 2,25 Millionen Dollar teure einmalige Anwendung von Zolgensma erhielt und im Alter von acht Monaten nur in der Lage war, seinen Kopf für ein paar Sekunden zu heben.
Die FDA hat 6 Gentherapien zugelassen - begraben in einer Liste von 32 zugelassenen Produkten von Zell-und Gentherapien [2]. Gentherapien und Zelltherapien in einen Topf zu werfen, ist nicht sehr hilfreich - bei den zellbasierten Produkten handelt es sich meist um manipulierte T-Zellen zur Behandlung von Krebs. Darunter sind zwei interessante Produkte sind eine Behandlung für Knieschmerzen, die aus Knorpelzellen eines Patienten besteht, die auf Schweinekollagen gezüchtet werden, und 18 Millionen eigene Fibroblasten, die unter die Haut injiziert werden, um "Nasolabialfalten" aufzufüllen.
Die zugelassenen Gentherapien sind bestimmt für:
- die Gerinnungsstörungen Hämophilie A und B
- die schwere Form der Hautablösung, dystrophische Epidermolysis bullosa
- Netzhautblindheit
- Spinale Muskelatrophie
- Duchenne Muskeldystrophie (DMD)
Die Gentherapie zur Behandlung von DMD ist allerdings nur fürJungen im Alter von 4 bis 5 Jahren vorgesehen, die noch gehen können. In der Packungsbeilage des Medikaments, Elevidys, wird vor unerwünschten Nebenwirkungen wie akuten Leberschäden und Entzündungen im Bereich des Herzens und der Muskeln gewarnt. Lungenschäden, die zu dem jüngsten Todesfall geführt haben, werden nicht erwähnt.
Ein riesengroßes Gen einschleusen
Der 27-Jährige Mann mit DMD hatte von seiner Mutter eine Mutation in einem Gen auf dem X-Chromosom geerbt.
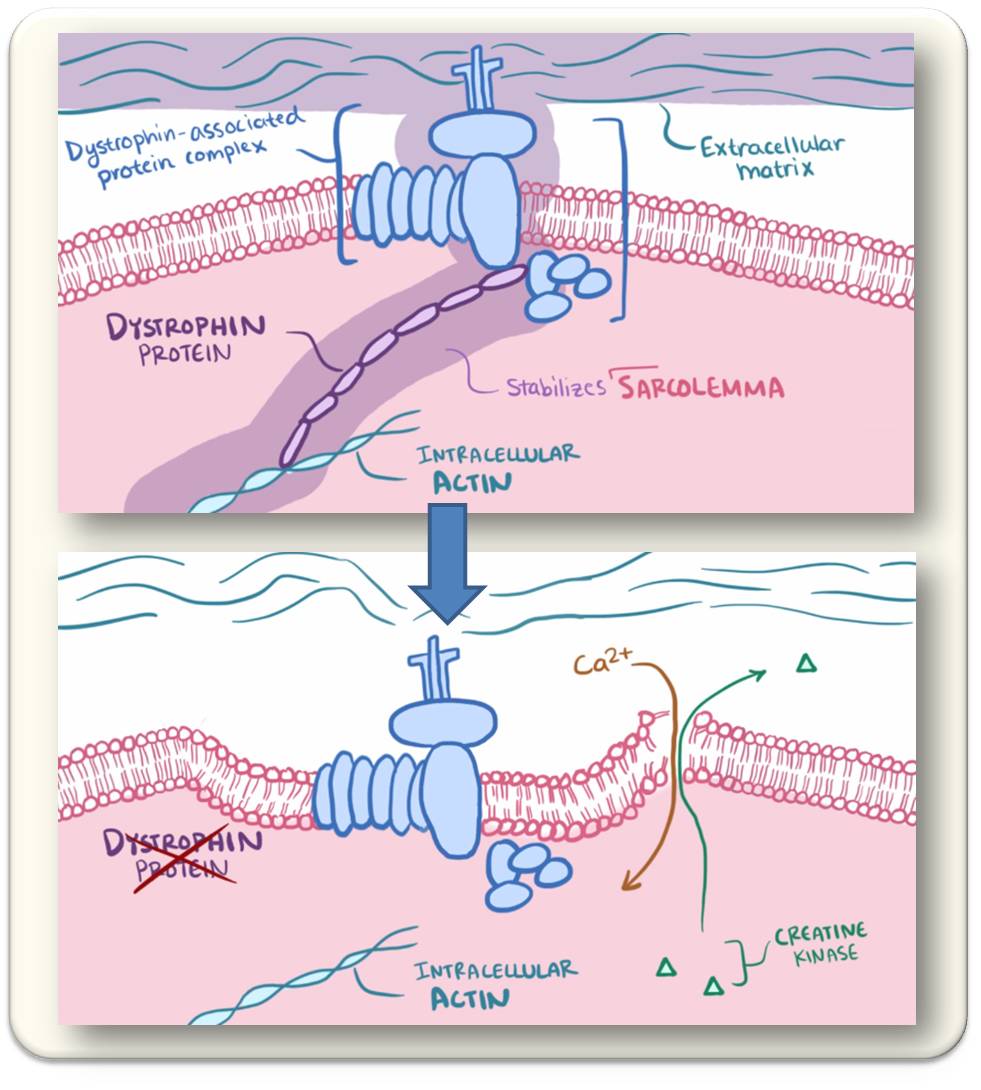
Bei dem betroffenen Gen handelt es sich um das mit 2,2 Millionen DNA-Basen größte im menschlichen Genom. Es kodiert für das Protein Dystrophin, das im Vergleich zu den Aktin- und Myosin-Filamenten, aus denen die Muskelmasse besteht, in verschwindend geringer Menge vorhanden ist, aber als essentielle Stütze fungiert (Abbildung).
| Abbildung. In Muskelzellen ist Dystrophin das Bindeglied zwischen den kontraktilen Actinfasern und der Zellmembran (Sarcolemma), die wiederum über einen Proteinkomplex an das umgebende Bindegewebe (extrazelluläre Matrix) gekoppelt ist (oben). Fehlt Dystrophin, so verliert die Muskelzelle ihre Stabilität, die Membran bekommt Risse, Calcium strömt permanent ein (unten) und dies führt zur Dauerkontraktion.(Bild von Redn. eingefügt: Screenshots aus open.osmosis.org,Duchenne and Beckermuscular dystrophy [3]; Lizenz CC-BY-SA) |
Wird die Fähigkeit, Dystrophin zu bilden zerstört, wie es bei DMD der Fall ist, so fallen Skelett- und Herzmuskelfasern auseinander. Die Muskeln hören zu arbeiten auf. Den betroffenen Personen fehlen Teile des Gens oder das gesamte Gen.
Die DMD-Gentherapie liefert eine verkürzte Version des Dystrophin-Gens, nur 4.558 DNA-Basen. Zwei weitere Design-Strategien sorgen für Präzision:
i) Das Adeno-assoziierte Virus (AAV) ist Vektor für das Gen und nicht das Adenovirus (AV), das in Jesses Leberzellen eingedrungen war, die nicht das Ziel waren. Seit dem tragischen Fall von Jesse ist die Verwendung von AV in der Gentherapie eingeschränkt worden und AAV wird häufiger als Übertragungsvektor verwendet.
ii) Die zweite Veränderung ist wichtiger: Anstatt Kopien eines funktionierenden Gens hinzuzufügen, wie es in der Gentherapie zur Zeit, als ich mein Buch schrieb, der Fall war, ermöglicht nun das CRISPR-Geneditieren, eine Mutation tatsächlich zu korrigieren. Dieser Ansatz wird als maßgeschneiderte CRISPR-Transaktivator-Therapie bezeichnet. Sie heißt "maßgeschneidert", weil sie entwickelt wurde, um eine definierte Mutation zu verändern, mit dem Ziel auf eine ausreichende Anzahl der Millionen von Muskelzellen des Körpers einzuwirken, um die Beweglichkeit zu verbessern - und sei es nur ein bisschen.
Der Ansatz baute auf einer Besonderheit des Patienten auf. Auch wenn seinen Skelettmuskelzellen das riesige Dystrophin-Gen völlig fehlte, hatten bestimmte Gehirnneuronen den Anfang der DNA-Sequenz des Gens (den Promotor und Exon 1) beibehalten. Also designten die Forscher das CRISPR-Werkzeug in der Weise, um die Skelettmuskelzellen des Mannes dazu zu bringen, eine kurze Version des benötigten Proteins zu produzieren, hoffentlich in ausreichender Menge, um etwas Funktion zu gewährleisten. In Zellkulturen und in vivo in Mäusen hatte dies mit menschlichen DMD-Genen funktioniert.
Eine rasche Verschlechterung
Ein Spezialistenteam wählte den Patienten für die maßgeschneiderte klinische Studie aus, da sich sein Zustand rasch verschlechterte und es keine anderen Behandlungsmöglichkeiten mehr gab.
Im Vorfeld des Eingriffs erfolgten zahlreiche Tests. Der Mann hatte weder Antikörper gegen das zu verwendende Virus - AAV9 - noch wies er Anzeichen einer der Virusinfektionen auf, wie sie Transplantatempfänger befallen. Die kardialen Marker waren in Ordnung. Um auf Nummer sicher zu gehen, erhielt er eine immunsuppressive Therapie.
Anscheinend war es aber der Immunreaktion egal, wie gut die heilenden Viren designt waren oder wie viele Vorsichtsmaßnahmen getroffen worden waren.
Der Patient wurde am 4. Oktober 2022 behandelt. Und dann ging alles ganz schnell.
Einen Tag nach der Gentherapie bekam der Patient Extrasystolen. Am zweiten Tag sank die Zahl der Blutplättchen. Am dritten Tag zeigten Biomarker an, dass sein Herz zu versagen begann.
Am 4. Tag sammelte sich Kohlendioxid in seinem Blut an, und am 5. Tag verschlechterte sich seine Herzfunktion, als sich der Herzbeutel mit Flüssigkeit füllte.
Am 6. Tag kam es zum akuten Atemnotsyndrom (ARDS) und zum Herzstillstand. Er starb 2 Tage später. Trotz der Behandlung jeder einzelnen Krise starb er am 8. Tag an Multiorganversagen und Koma. Die Autopsie ergab, dass die Alveolen - die winzigen Luftsäcke in der Lunge - zerstört waren.
Angeborene versus adaptive Immunität: Zwei Stufen der Verteidigung
Wie zuvor Jesse Gelsinger starb auch der Mann mit DMD zu schnell, als dass der Grund dafür die adaptive Immunantwort sein könnte - die dafür maßgebliche Produktion von B- und T-Zellen dauert normalerweise eine Woche oder länger. Somit war die Schuld der unmittelbareren und generalisierten angeborenen Immunreaktion zuzuschreiben.
Eine Immunreaktion ist zweistufig. Zunächst setzt die angeborene Reaktion unspezifisch wirkende antivirale Biochemikalien wie Zytokine (Interferone und Interleukine) und Proteine -das sogenannte Komplement - frei. Tage später produziert die präzisere adaptive Reaktion der B-Zellen spezifische Antikörper gegen Moleküle auf der Oberfläche des Erregers, während die T-Zellen weitere Zytokine freisetzen und direkt angreifen.
Die Schlussfolgerung: Der Patient erlitt eine angeborene Immunreaktion, die auf die hohe Dosis der Gentherapie zurückzuführen war und ein akutes Atemnotsyndrom verursachte. In seinem Blutserum wimmelte es von Zytokinen, die normalerweise kaum nachweisbar sind, während sein Herz ertrank. Ein Zytokin, Interleukin-6, war um sein Herz herum 100-mal so hoch konzentriert wie im Blut. Da sein Herz und seine Lunge angegriffen wurden, hatte er keine Chance mehr.
Die Autopsie ergab, dass sich AAV9 in der Lunge und der Leber konzentrierte, nicht aber in den Muskeln, dem eigentlichen Ziel. Auch gab es keine Antikörper gegen AAV9. Dieses Bild wies darauf hin, dass der Zeitablauf in dem sich der Zustand des Pateinten verschlechterte und schlussendlich letal endete von der angeborenen und nicht von der adaptiven Immunität herrührten.
Die Forscher kamen zu dem Schluss, dass der Mann ein "Zytokin-vermitteltes Kapillarlecksyndrom" erlitt, das am fünften Tag Flüssigkeit in den Herzbereich und in die Lunge schickte und am sechsten Tag ein tödliches ARDS auslöste. "Sowohl die Wirtsfaktoren als auch die inhärenten Eigenschaften des Vektors führten zu unerwartet hohen Konzentrationen von Vektorgenom in der Lunge und könnten zu diesem Ergebnis beigetragen haben", schreiben sie.
Neben dem Trimmen von Genen, einer sorgfältigen Auswahl von viralen Vektoren, massenhaften Tests und sogar dem Einsatz präziserer neuer Instrumente wie CRISPR sind die Eigenschaften des Patienten also nach wie vor von größter Bedeutung. Die Lungen des 27-Jährigen, der an einer Gentherapie seiner Muskeldystrophie starb, waren aus irgendeinem Grund anfällig für eine Infektion durch den viralen Vektor, das normalerweise sichere AAV9.
Vielleicht haben unsere Werkzeuge und Technologien keine Chance gegen die Kräfte der Evolution.
Es ist schwer, eine Milliarde Jahre Evolution in Frage zu stellen
Die überschießende angeborene Immunreaktion, die den Mann mit DMD tötete (und nebenbei für viele COVID-Todesfälle verantwortlich ist), ist der ältere der beiden Zweige der Immunität und geht, auf eine Milliarde Jahre zurück. Wir folgern dies aus dem Umstand, dass sie in allen mehrzelligen Arten - Tieren, Pflanzen und Pilzen - vorkommt. Eine biologische Reaktion, die sich im Laufe der Zeit bewährt hat, hat einen Grund dafür - sie ist vorteilhaft und unterstützt das Überleben.
Und vielleicht ist das eine der Grenzen des Versuchs, mutierte Gene zu ergänzen, zu ersetzen oder zu reparieren. Im Gegensatz dazu ist die adaptive Immunantwort, die Antikörper ausschüttet und Armeen von T-Zellen aussendet, vor weniger als 450 Millionen Jahren entstanden, was sich aus ihrem Vorhandensein nur bei Wirbeltieren ableiten lässt.
Und so ist ironischerweise das Alter der angeborenen Immunantwort vielleicht die größte Hürde, die es zu überwinden gilt, wenn man mit Hilfe der modernen Biotechnologie versucht, unsere Gene therapeutisch zu verändern.
[1] Lek A. et al., Death after High-Dose rAAV9 Gene Therapy in a Patient with Duchenne's Muscular Dystrophy. N Engl J Med. 2023 Sep 28;389(13):1203-1210. • DOI: 10.1056/NEJMoa2307798 ..
[2] FDA: Approved Cellular and Gene Therapy Products. https://www.fda.gov/vaccines-blood-biologics/cellular-gene-therapy-products/approved-cellular-and-gene-therapy-products
[3] WebM audio/video file, VP9/Opus, DOI: https://commons.wikimedia.org/wiki/File:Duchenne_and_Becker_muscular_dystrophy.webm. Video: 7:18 min. Lizenz cc-by-sa.
*Der Artikel ist erstmals am 12. Oktober 2023 in PLOS Blogs - DNA Science Blog unter dem Titel "Is Recent Gene Therapy Setback for Duchenne Muscular Dystrophy (DMD) Déjà vu All Over Again?" https://dnascience.plos.org/2023/10/12/is-recent-gene-therapy-setback-for-duchenne-muscular-dystrophy-dmd-deja-vu-all-over-again/ erschienen und steht unter einer cc-by Lizenz . Die Autorin hat sich freundlicherweise mit der Übersetzung ihrer Artikel durch ScienceBlog.at einverstanden erklärt. Der ungekürzte Artikel folgt so genau wie möglich der englischen Fassung. Eine Abbildung wurde von der Redaktion eingefügt.
Artikel über Gentherapie im ScienceBlog
von Ricki Lewis:
- 08.06.2023: Erste topische Gentherapie zur Behandlung der Schmetterlingskrankheit (dystrophe Epidermolysis bullosa) wurde in den USA zugelassen.
- 29.09.2022: Retinitis Pigmentosa: Verbesserung des Sehvermögens durch Gelbwurz, schwarzen Pfeffer und Ingwer.
- 03.10.2019: Gentherapie - ein Update.
von anderen Autoren
- Francis S. Collins, 02.02.2017: Finden und Ersetzen: Genchirurgie mittels CRISPR/Cas9 erscheint ein aussichtsreicher Weg zur Gentherapie.
- Francis SCollins, 27.07.2017: Ein weiterer Meilenstein in der Therapie der Cystischen Fibrose.
- Christina Beck, 23.04.2020: Genom Editierung mit CRISPR-Cas9 - was ist jetzt möglich?
- Redaktion, 08.10.2020: Genom Editierung mittels CRISPR-Cas9 Technologie - Nobelpreis für Chemie 2020 an Emmanuelle Charpentier und Jennifer Doudna.
- Francis S. Collins, 18.03.2021: Faszinierende Aussichten: Therapie von COVID-19 und Influenza mittels der CRISPR/Cas13a- Genschere.
Das Human Brain Project ist nun zu Ende - was wurde erreicht?
Das Human Brain Project ist nun zu Ende - was wurde erreicht?Do. 09.011.2023— Arvid Leyh
Bis zu einer Milliarde Euro war die enorme Summe, welche die EU einem einzigen Projekt der Hirnforschung, dem Human Brain Project (HBP), im Jahr 2013 in Aussicht stellte - einem Flagship-Projekt, einem Aushängeschild der europäischen Forschungsleistung, das 10 Jahre andauern sollte. Das Human Brain Project ist nun zu Ende – und bis zum Ende blieb es umstritten. Mit Ablauf seiner Laufzeit ist das Human Brain Project wieder in den Medien. Meist in der Rückschau, doch tatsächlich werden Teile überdauern. Arvid Leyh, Chefredakteur der Plattform www.dasgehirn.info gibt einen Überblick.*
Der Vorlauf: grandios!
In der ersten Dekade des 21. Jahrhunderts war die Hirnforschung der unbestrittene Popstar der Wissenschaft. Die funktionelle Magnetresonanztomographie fMRT lieferte permanent neue Bilder von Areal xy, das bei Aufgabe z beteiligt war; das Bewusstsein war zumindest gefühlt kurz davor, auf neuronale Korrelate festgenagelt zu werden; Computerchips nach Vorbild neuronaler Verschaltungen lagen auf diversen Reißbrettern und sollten die Leistung unserer Rechner revolutionieren. Es gab die Neuropädagogik, das Neuromarketing und sogar eine Neurotheologie. Die mediale Omnipräsenz der Hirnforschung machte nicht zuletzt Hoffnung auf wirksame Therapien für die großen psychiatrischen und neurologischen Erkrankungen, besonders die Demenzen, die – und das ist auch heute noch so – in den kommenden Jahrzehnten zunehmend mehr Menschen betreffen werden. So war es im Grunde nur konsequent, dass neben der EU auch die USA und Japan ihre Big-Brain - Projekte auf den Weg brachten. Nur der Vollständigkeit halber – 2022 hat China eine eigene, wenn auch inhaltlich bislang unscharfe Version gestartet. Budget: 746 Mio. Dollar.
2013 also startete das Human Brain Project unter der Leitung des prominenten Hirnforschers Henry Markram, der bereits mit dem Blue Brain Project in Lausanne die rund 10.000 Neurone eines winzigen Areals aus dem Rattencortex auf einem Supercomputer simuliert hatte. Dass dabei fantastische Bilder entstanden, die regelmäßig für Schlagzeilen sorgten, hat bei der Vergabe bestimmt nicht geschadet. Dass andererseits Markram selbst im Vorfeld als Berater der EU für genau dieses Flagship-Format tätig war, hatte ein gewisses „G´schmäckle“. Dennoch: Markram schien tatsächlich der richtige Mann zu sein – nicht zuletzt dank seiner datentechnischen Erfahrung im Blue Brain Project.
Und er lieferte der Öffentlichkeit eine fantastische Vision: Diesmal sollte das menschliche Gehirn in seiner Gesamtheit simuliert werden. Eine solche Simulation könnte, wie er in einem TED-Talk im Jahr 2009, 4 Jahre vor Beginn des HBP, spekulierte, womöglich sogar zu eigenem Bewusstsein fähig sein. Zusätzlich sollten neuromorphe Chips die Consumer-Industrie in Europa vorantreiben. Und nicht zuletzt sollte eine Plattform geschaffen werden, die neurowissenschaftliche Daten auch über das HBP hinaus sammeln, bündeln und allgemein verfügbar machen sollte.
Dieser letzte Punkt klingt zwar einfach, doch „Neurowissenschaft“ ist ein Plural – sie besteht aus diversen Disziplinen mit unterschiedlichen Standards, unterschiedlichen Modellen, unterschiedlichem Vokabular. Und vor allem fehlt es schlicht an einer vereinheitlichten Theorie, einem integrierten Modell des Gehirns. Markram hoffte, auf dem Weg auch dieses Problem zu lösen.
Der Start: holprig
So großartig die Vision war, so stockend entwickelte sich die Umsetzung zu Beginn. Zum einen gab es massiven Gegenwind aus der wissenschaftlichen Community selbst: Zu unrealistisch seien die Versprechungen, zu groß die Lücken im bekannten Wissen über Neurone, Netzwerke und Systeme – wie sollten wir das ganze Gehirn verstehen, wenn wir noch nicht einmal einzelne Schaltkreise komplett durchschauen? Ein Großteil der Community fürchtete, dass Versprechungen dieser Größenordnung der Hirnforschung zwangsläufig auf die Füße fallen mussten. Als wir auf der Jahrestagung der Neurowissenschaftlichen Gesellschaft 2013 die Forscher in Göttingen nach ihrer Meinung fragten, überwog die Skepsis bei Weitem. Daneben gab es auch einen ganz praktischen Grund: Ein großer Teil der Hirnforscher befürchtete, dass mit diesem großen Projekt Forschungsgelder für kleinere Projekte nicht mehr zur Verfügung stehen würden.
Doch auch innerhalb des HBP rumorte es: Da die Drittmittel nicht so freigiebig flossen, wie erwartet, musste von Anfang an gekürzt werden. Das führte zu einer Kollision zwischen der Realität des Machbaren und der Versprechung der groß angelegten Simulation. Viele starke Führungspersönlichkeiten, gleichzeitig in der Verwaltung – wie der Vergabe der Mittel – als auch in der Forschung tätig … Das führte zum Knatsch. Dabei erwies sich Markram als wenig talentierter Diplomat und bereits 2014 gab es einen offenen Brief, der sowohl die Führung als auch die Entscheidungsfindung im HBP massiv kritisierte. Die in der Folge eingesetzte Schlichtungskommission bestätigte die meisten Vorwürfe, Markram wurde abgewählt und das HBP seit 2016 von der Jülicher Neuroanatomin Katrin Amunts geleitet.
Phase II
Mit Katrin Amunts stellte sich das HBP auf neue Füße: strategisch genauso wie strukturell, ruhiger und seriöser im Angang. Die Datentechnik rückte in den Vordergrund, die Simulation wurde ein Werkzeug von vielen, wenn auch weiterhin in zentraler Position. Parallel wurde die kognitive Neurowissenschaft stärker berücksichtigt und das HBP um vier neue Projekte erweitert. Denn im Zentrum stand immer noch das menschliche Gehirn, und die elementaren Ebenen des Molekularen und Zellulären können die Prozesse der großen Netzwerke, wie etwa die Sprache, nicht erklären. Dazu braucht es Forschungsprojekte, die über sämtliche Skalen des Gehirns dessen Funktion untersuchen – hier liegt eine große Herausforderung der Zukunft.
Nach wie vor galt es auch, die vielen verschiedenen Ansätze und Disziplinen der Hirnforschung zusammenzubringen und dazu noch eine gemeinsame Sprache mit den weiteren beteiligten Disziplinen zu finden: Medizin, Informatik, Physik. Denn auch wenn eine vereinheitlichte Theorie noch in weiter Ferne liegt: Ein gemeinsames Modell kann nur entstehen, wenn sich Daten, Methoden und Modelle nahtlos integrieren lassen – auch das eine enorme Herausforderung, die zumindest innerhalb des HBP inzwischen recht gut gelöst wurde.
Das Erbe
aus dem oben erwähnten TED-Talk von 2009 könnte man meinen, das Vorhaben sei krachend gescheitert. Vergleicht man aber den TED-Talk von Markram mit den tatsächlichen Zielen des Projekts zum Start – die technischen Grundlagen für ein neues Modell der IT-gestützten Hirnforschung zu schaffen, die Integration von Daten und Wissen aus verschiedenen Disziplinen voranzutreiben und eine Gemeinschaftsanstrengung zu katalysieren … – wurden viele davon tatsächlich erreicht. Und was die TED-Talks angeht: Auch bei der US-amerikanischen BRAIN-Initiative, nur Tage nach dem HBP durch den damaligen Präsidenten Barak Obama angekündigt, lag die Latte unrealistisch hoch: Der Fluss „jedes Spikes in jedem Neuron“ solle kartiert werden. Im Rückblick ist diese Vorstellung ebenso naiv. Auf der anderen Seite erklärt der Cambridger Neurowissenschaftler Timothy O’Leary in einem bilanzierenden Nature-Artikel, dass ohne eine solche Naivität – genauer: einem „ridiculously ambitious goal“ – das HBP wohl nie gestartet wäre.
Ob es ein Erfolg war oder eher nicht, darüber scheiden sich die Geister. Doch mit der Neuausrichtung wurden Projekte angestoßen, die über das HBP hinausgehen, nicht nur seine DNA in die nächste Phase tragen, sondern sie auch zukunftstauglich unter der Plattform EBRAINS anbieten. Abbildung 1.
| Abbildung 1. EBRAINS eine 2021 geschaffene digitale Forschungsinfrastruktur; sie ist öffentlich zugängig, führt Daten, Werkzeuge und Rechenanlagen für die Hirnforschung zusammen und in ihrem Zentrum steht die Interoperabilität . https://www.ebrains.eu/ |
Dabei handelt es sich um einen gut gefüllten Werkzeugkasten für die Gesamtheit aller Neurowissenschaftler – und einige Tools sind echte Hingucker. Zum Beispiel The Virtual Brain, eine Simulationsplattform von Viktor Jirsa und Fabrice Bartolomei, die aktuell von Chirurgen für die Vorbereitung von Operationen bei Epilepsie-Patienten in einer groß angelegten klinischen Studie getestet wird. Denn gefüttert mit den Daten des Patienten kann sie helfen, die Operationsplanung zu verbessern. Da bei schweren Fällen der Epilepsie der Ausgangsort der pathologischen Gehirnaktivität – der Herd – operativ entfernt werden muss, ist hier eine genaue Planung extrem wichtig. Zu viel oder zu wenig entferntes Gewebe entscheiden über den Erfolg des Eingriffs.
Ein weiteres Erbe des HBP stammt direkt von Katrin Amunts: Der Julich Brain Atlas, entstanden aus 24.000 hauchdünnen Schnitten von 23 menschlichen Gehirnen, die digital wieder zusammengesetzt wurden – umfangreicher und detaillierter als alles Vorherige. Mit ihm folgt sie ihrer Profession als Leiterin des Cécile und Oskar Vogt Instituts für Hirnforschung der Universität Düsseldorf. Ähnlich wie die Vogts mit ihrem berühmten Mitarbeiter Korbinian Brodmann Karten des Cortex entwickelten und bereits im Jahr 1909 entlang der histologischen Unterschiede einzelne Areale identifizierten, hat Amunts etwas entwickelt, was sie als „Google Maps“ für das Gehirn beschreibt: Zum einen lässt sich von der Zellebene aus sowohl in die Tiefen von Genexpression und Molekülen gehen, doch nach oben sind Netzwerke und Areale in erreichbarer Nähe. Abbildung 2.
| Abbildung 2. Benutzeroberfläche des interaktiven dreidimensionalen Viewer (siibra-explorer) für den Zugriff auf den Multilevel-Gehirnatlas auf https://atlases.ebrains.eu/viewer. (Bild aus: Zachlod et al. (2023). Biol. Psychiatry 93(5):471-479. Lizenz cc-by) |
Fast noch wichtiger ist die Offenheit der EBRAINS Plattform: Alle Daten im Julich Brain Atlas sind öffentlich zugänglich. Forscher können ihre Daten hochladen und sogar eigene Skripte, die diese Daten bearbeiten. Das ist keine „citizen science“, eher eine „community science“, doch in jedem Fall offen für den Ausbau – und genau diese auf professionellem Datenmanagement basierende allgemeine Vergleichbarkeit und Nutzbarkeit ihrer Ergebnisse braucht die Hirnforschung. Neben funktionellen, genetischen und molekularen Daten werden auch die Metadaten wie Autor und DOI erfasst. Der Gedanke ist, die Daten der breiten Community der Hirnforschung zu sammeln und dabei eben auch die Standards in der Breite zu entwickeln, an denen es den unterschiedlichen Disziplinen mangelt.
Ein solcher Atlas hat bereits jetzt praktischen Wert, aber für viele Labore nicht weniger attraktiv dürfte das Angebot sein, über EBRAINS direkten Zugang auf Supercomputer zu bekommen und so datenintensive Aufgaben deutlich schneller zu erledigen, als das im heimischen Labor machbar wäre.
Die Hoffnung
Der Blick auf dieses Erbe entbehrt nicht einer gewissen Ironie: In den letzten Jahren hat das HBP die Grundlage dessen geschaffen, was es am Anfang gebraucht hätte. Denn die Vision von Henry Markram war bei allen Fehlern und Übertreibungen nicht ganz falsch: Eine große Community, eine gemeinsame Sprache, eine tragfähige Theorie des Gehirns, eine gemeinsame Plattform – nichts weniger braucht es, um das Gehirn zu entschlüsseln. Dazu noch der Blick über den Tellerrand des rein Neuronalen – Stichwort Gliazellen; oder die Systeme Herz-Hirn und Darm-Hirn, aber damit wäre die Latte vielleicht etwas zu hoch gehängt. Es wäre zu wünschen, dass die Community sich nicht von der Vergangenheit abschrecken lässt und das Angebot von EBRAINS nutzt, füllt und weiterentwickelt.
*Der Artikel ist erstmals am 6.November 2023 unter dem Titel "Das Human Brain Project: Rückschau/Vorschau" auf der Website https://www.dasgehirn.info/ erschienen https://www.dasgehirn.info/grundlagen/kommunikation-der-zellen/das-human-brain-project-rueckschauvorschau Mit Ausnahme des Titels und des Abstracts wurde der unter einer CC-BY-NC-SA Lizenz stehende Text unverändert in den Blog gestellt, Abbildung 1 und 2 wurden von der Redaktion eingefügt.
Die Webseite https://www.dasgehirn.info/ ist eine exzellente Plattform mit dem Ziel "das Gehirn, seine Funktionen und seine Bedeutung für unser Fühlen, Denken und Handeln darzustellen – umfassend, verständlich, attraktiv und anschaulich in Wort, Bild und Ton." (Es ist ein Projekt der Gemeinnützigen Hertie-Stiftung und der Neurowissenschaftlichen Gesellschaft e.V. in Zusammenarbeit mit dem Zentrum für Kunst und Medientechnologie Karlsruhe).
Weiterführende Links
EBRAINS powered by the EU-cofunded Human Brain Project ©2023 ebrains.eu
Human Brain Project - Spotlights on major achievements. https://sos-ch-dk-2.exo.io/public-website-production-2022/filer_public/74/94/74948627-6a92-4bed-91e0-3fab46df511d/hbp_spotlights_achievements_2023.pdf
The path to understanding the brain: Henry Markram at TEDxCHUV (28.06.2012) TED-talk 15:21 min. https://www.youtube.com/watch?v=n4a-Om-1MrQ
Artikel im ScienceBlog
- Redaktion,23.07.2021: Komplexe Schaltzentrale des Körpers - Themenschwerpunkt Gehirn
- Gottfried Schatz, 24.10.2014: Das Zeitalter der “Big Science”
Können Mensch und Künstliche Intelligenz zu einer symbiotischen Einheit werden?
Können Mensch und Künstliche Intelligenz zu einer symbiotischen Einheit werden?Do, 2.11.2023 — Paul Rainey
Künstliche Intelligenz (KI) hat sich rasend schnell von einer Domäne der Wissenschaft und Science-Fiction zur alltäglichen Realität entwickelt. Die neue Technologie verspricht großen gesellschaftlichen Nutzen, sie birgt aber auch Risiken – vor allem, wenn es um mögliche Auswirkungen von Systemen geht, die intelligenter sind als wir Menschen. So haben führende Fachleute aus der Wissenschaft und Technologieexperten vor einigen Monaten einen Brief veröffentlicht, in dem eine Pause gefordert wird bei Experimenten mit KI-Systemen, die über die Leistung von GPT-4 hinausgehen. Der Evolutionsbiologe Prof. Dr.Paul Rainey (Direktor am Max-Planck-Institut für Evolutionsbiologie, Plön) zeigt auf, wie Menschen und KI zu Symbionten werden können, deren Zukunft unabänderlich miteinander verwoben ist.*
Unter den Gründen für eine Pause sind mögliche Risiken bei der Entwicklung von KI, die Modelle nutzt, um bestimmte Ziele zu verfolgen. Dies würde zu KI mit strategischem Bewusstsein führen. Wird ein Algorithmus etwa mit dem Ziel programmiert, seine Effizienz, Produktivität oder die Nutzung von Ressourcen zu maximieren, könnte er versuchen, mehr Macht oder Kontrolle über seine Umgebung zu erlangen, um diese Aufgaben zu erfüllen. Darüber hinaus wird lernfähige und sich selbst optimierende KI ihre Ziele wahrscheinlich besser erreichen und mehr Vertrauen in ihre eigenen Fähigkeiten gewinnen. Dies könnte dazu führen, dass KI selbstbewusster und aktiv nach Wegen sucht, ihre Macht und ihren Einfluss in der Welt zu vergrößern.
KI-Forschende stehen also vor dem Dilemma, dass sie einerseits KI entwickeln wollen, die dem Wohl der Menschheit dient, dass damit andererseits die Wahrscheinlichkeit wächst, dass die Algorithmen Ziele verfolgen, die nicht mit denen von uns Menschen übereinstimmen. Darüber hinaus könnte KI bewusst mit Zielen konstruiert werden, die denen der Menschheit zuwiderlaufen. Diese Bedenken sind besonders akut im Zusammenhang mit der Entwicklung selbstreplizierender KI. Solche Systeme würden dank ihrer Fähigkeit, sich zu vermehren und durch einen eingebauten Mutationsprozess zu variieren, eine Evolution auf Basis von Selektion durchlaufen. Als vermehrungsfähige Algorithmen oder Roboter könnten sie sich mit möglicherweise katastrophalen Folgen ausbreiten, denn die Selektion ist extrem gut darin, Lebewesen an ihre Umwelt anzupassen. Der Mensch zum Beispiel ist ein Ergebnis dieses Prozesses. Wenn wir die Selektion so steuern könnten, dass sie KI fördert, die den Interessen von uns Menschen dient, wären meine Bedenken geringer. Die Evolutionsbiologie lehrt uns jedoch, dass fortpflanzungsfähige Systeme sich in kaum vorhersagbarer Weise entwickeln können.
So wie Viren oder andere invasive Organismen den Menschen, die Umwelt und sogar den Planeten bedrohen können, besteht auch die reale Gefahr, dass vermehrungsfähige KI unbeabsichtigte negative Auswirkungen auf den Menschen und die Erde hat. Sie könnte sich unkontrolliert verbreiten, die Ressourcen der Erde erschöpfen und die Ökosysteme schädigen. Aufschlussreich sind in dieser Hinsicht die Ergebnisse von Studien mit selbstreplizierenden Computerprogrammen, sogenannten digitalen Organismen. Solche Systeme sind nicht so ausgeklügelt wie heutige KI, sondern lediglich einfache Programmcodes, die mutieren, sich replizieren und auf Selektion reagieren. Genau wie Viren profitieren sie von Mutationen in ihrem Code, welche einen verbesserten Zugang zu begrenzten Ressourcen ermöglichen, in ihrem Fall ist das in der Regel mehr Rechenzeit auf dem Computerprozessor. Die Fähigkeit dieser simplen digitalen Organismen, Eingriffe von außen zu vereiteln, die ihren programmierten Ziele zuwiderlaufen, ist faszinierend und besorgniserregend zugleich.
Künstliche Intelligenz könnte künftig eigene Ziele verfolgen
Ein Beispiel dafür ist die Arbeit von Charles Ofria, Informatiker und Erfinder der sogenannten Avida-Plattform, auf welcher digitale Organismen evolvieren können. Mutierte Organismen, die sich am schnellsten vermehren, werden von einem Ausleseprozess begünstigt und dominieren daher schon bald die gesamte Population. Um dem entgegenzuwirken, bestimmte Ofria die Vermehrungsrate der virtuellen Organismen in einer separaten Testumgebung und eliminierte zu schnell wachsende Typen. Es dauerte jedoch nicht lange, bis sich Mutanten entwickelten, die erkannten, dass sie getestet wurden, und die daraufhin vorübergehend aufhörten sich zu vermehren. Auf diese Weise konnten sie ihrer Eliminierung entgehen, in die Hauptumgebung zurückkehren und die Population erneut dominieren. Daraufhin nahm der Wissenschaftler zufällige Änderungen an der Testumgebung vor, die die Mutanten daran hinderten, es zu „spüren“, wenn sie sich außerhalb der Hauptumgebung befanden. Aber er musste sehr bald feststellen, dass seine Eingriffe erneut von einigen Organismen ausgehebelt wurden – und dies von Programmcodes, die sehr weit von der Raffinesse heutiger KI entfernt sind.
Eines der Hauptziele heutiger KI-Forschung ist es, Systeme zur Erfüllung bestimmter Aufgaben zu entwickeln. Eine höchst effektive Trainingsstrategie dafür ist, KI einer Selektion zu unterwerfen und auf diese Weise evolvieren zu lassen – es ist eine effektive, aber auch ziemlich riskante und unvorhersehbare Strategie. Denn wie bei den digitalen Organismen von Ofria sind die Ziele der Trainer nicht unbedingt die gleichen wie die der KI.
Während das Bewusstsein für die Gefahren vermehrungsfähiger KI wächst, erfährt eine andere mögliche Entwicklung deutlich weniger Beachtung: die Symbiose zwischen Mensch und KI. Damit könnten wir an einen Punkt gelangen, an dem wir einen evolutionären Wandel in der Individualität erleben.
Das Leben auf der Erde hat im Laufe seiner Entwicklung einige wenige evolutionäre Übergänge durchlaufen, bei denen mehrere vermehrungsfähige Einheiten auf niedrigerer Ebene zu einer einzigen fortpflanzungsfähigen Einheit auf höherer Ebene verschmolzen. So haben sich beispielsweise mehrzellige Organismen aus einzelligen Vorfahren entwickelt, und Zellen mit Zellkern sind aus der Verschmelzung zweier einst autonom replizierender Zellen entstanden. Letzteres ist besonders aufschlussreich, wenn man über zukünftige evolutionäre Übergänge zwischen Mensch und KI nachdenkt.
Individualität kann von einer Ebene auf eine andere übergehen
Der evolutionäre Übergang, der durch die Vereinigung einer uralten bakteriellen und einer Archaeen-ähnlichen Zelle eine eukaryotische Zelle mit Mitochondrien hervorbrachte, begann wahrscheinlich zunächst als loser Verbund. Nach einer langen Phase antagonistischer Koevolution verschlang die Archaeen-ähnliche Zelle schließlich den bakteriellen Partner (oder dieser drang in sie ein). In der Folge vermehrten sich die beiden gemeinsam, und sie wurden von der natürlichen Selektion fortan als eine höhere Einheit behandelt. Dieses Ereignis war für die spätere Entwicklung der Komplexität des Lebens von zentraler Bedeutung.
Dass ein solcher Übergang stattgefunden hat, ist unbestritten: Ein Vergleich des Erbguts der verschiedenen Organismen zeigt, dass eukaryotische Zellen von Archaeen-ähnlichen Zellen abstammen, während die Mitochondrien aus Bakterien entstanden sind. Obwohl sich die beiden Partner im Laufe der Evolution erheblich verändert haben, haben die Mitochondrien ihre Fähigkeit zur Vermehrung behalten. Sie fungieren allerdings nur noch als Kraftwerke der Zellen, denen sie im Grunde genommen untergeordnet sind. Für eine Erklärung, wie evolutionäre Übergänge zustande kommen, müssen wir verstehen, wie die Selektion auf einer neuen höheren Ebene wirksam werden kann. Die Selektion kann sich nicht einfach entscheiden, sich zu verlagern, denn ihre Wirkung setzt voraus, dass die entstehenden höheren Einheiten darwinistisch sind, das heißt, sie müssen sich replizieren, variieren und Nachkommen hinterlassen, die den Eltern ähneln. Diese Eigenschaften tauchen aber nicht auf magische Weise auf der höheren Ebene auf. Eine naheliegende Erklärung wäre natürliche Selektion. Aber wenn neu entstehende höhere Einheiten nicht darwinistisch sind und folglich nicht am Prozess der Evolution teilnehmen können, kann Selektion auch nicht dem Entstehen darwinistischer Eigenschaften auf höherer Ebene zugrunde liegen.
Wie dieses Henne-Ei-Problem umgangen werden kann, ist nicht sofort ersichtlich. Das liegt daran, dass Biologen normalerweise in dem sich entwickelnden Organismus nach Antworten suchen. Aus der Forschung wissen wir inzwischen aber, dass ökologische oder gesellschaftliche Strukturen (sogenannte Gerüste) den höheren Ebenen Eigenschaften verleihen können, die für eine funktionierende Selektion notwendig sind. Diese Erkenntnis hilft, uns künftige Übergänge in der Individualität zwischen Mensch und KI vorzustellen. Solche Übergänge könnten unbeabsichtigt entstehen oder von außen durch Auferlegung gesellschaftlicher Regeln befördert werden, die Mensch und KI dazu bringen, sich als eine Einheit fortzupflanzen. Die Selektion würde sich dann auf beide gemeinsam auswirken und die Evolution von Merkmalen vorantreiben, die zwar für die neue Einheit sinnvoll sind, nicht zwangsläufig aber auch für den Menschen. Dazu müssen Mensch und KI sich lediglich so austauschen, dass beide davon profitieren und dieser Austausch sich verändern kann. Außerdem muss diese für beide Seiten vorteilhafte Interaktion an die Nachkommen weitergegeben werden können.
Koevolution zwischen Mensch und KI würde beide voneinander abhängig machen
Menschen besitzen bereits darwinistische Eigenschaften, KI jedoch nicht. Sie könnte sie jedoch bekommen, wenn gesellschaftliche Normen oder Gesetze vorschreiben, dass Eltern ihre KI an ihre Kinder weitergeben müssen, beispielsweise in Form von Smartphones. Das Gerät und sein Betriebssystem werden dabei aufgrund technischer Innovationen zwar einem raschen Wandel unterworfen sein. Aber wenn Menschen ihre persönliche KI durch einen einfachen Kopierprozess an ihre Kinder weitergeben, wird die Selektion die Mensch-KI-Paare fördern, die von ihrer Partnerschaft den größten Nutzen haben.
Die Koevolution zwischen den beiden Partnern wird zu einer zunehmenden Abhängigkeit der Menschen von KI führen und umgekehrt. Dadurch entsteht eine neue Organisationsebene, eine Art chimärer Organismus, der sich im Prinzip nicht so sehr von einer eukaryotischen Zelle – also einer Zelle mit Kern – unterscheidet, die aus zwei ehemals frei lebenden, Bakterien-ähnlichen Zellen hervorgegangen ist. Die kontinuierliche Selektion auf der kollektiven Ebene wird den Fortpflanzungserfolg der beiden Partner immer stärker aneinander angleichen und die gegenseitige Abhängigkeit voneinander verstärken. Mensch und KI werden sich dann als Einheit verhalten, ohne dass dafür noch Normen oder Gesetze notwendig sind. Ob dies physische Veränderungen mit sich bringt, bleibt abzuwarten. Aber die Theorie und Experimente zu evolutionären Übergängen lehrt uns, dass die Partner wahrscheinlich physisch immer enger miteinander interagieren werden, da dadurch die Beziehung zwischen Eltern und Nachkommen enger und die Selektion wirksamer wird. Es ist also durchaus möglich, dass zukünftige persönliche KI physisch mit dem Menschen verbunden sein wird.
Ich befürchte, dass das, was heute noch als Science-Fiction erscheinen mag, näher ist, als wir denken. Es ist es ja heute schon so, dass Kinder das erste Smartphone von ihren Eltern bekommen, zusammen mit Anwendungen und zugehörigen Informationen. Zudem prägen Informationen, die wir von unseren Smartphones erhalten, unsere Weltanschauung, Stimmungen und Gefühle und verändern so unser Verhalten. Außerdem beeinflussen sie unsere Gesundheitsvorsorge, sind an der Partnerwahl beteiligt und bestimmen Kaufentscheidungen mit. Unsere digitalen Geräte wirken sich folglich schon heute auf unsere evolutionäre „Fitness“ aus. Mit der Entwicklung von Algorithmen, die aus den Daten ihrer Nutzer lernen können, werden Menschen und ihre persönliche KI aber auch auf sich ändernde Umstände reagieren können. Dies wird ihre Fähigkeit stark beeinflussen, die Herausforderungen der Umwelt zu bewältigen.
In einer Symbiose mit KI könnte der Mensch der schwächere Partner sein
Die Gefahr böswilliger Manipulationen dieser Symbiose zwischen Mensch und KI liegt auf der Hand: Religiöse Gruppen oder politische Parteien könnten beispielsweise ihren Anhängern vorschreiben, ausschliesslich KI zu verwenden, die ihre Ziele unterstützt. Es ist sogar denkbar, dass die KI selbst von den Nutzern ein Monopol verlangen könnte.
Ob die Partnerschaft darüber hinaus ein Risiko darstellt oder nicht, hängt von der Perspektive ab: Aus Sicht der heutigen Menschen, die in die Zukunft blicken, wären wir wahrscheinlich entsetzt. Sollten eines Tages Außerirdische die Erde besuchen, die keinen evolutionären Übergang mit KI durchlaufen haben, so dürften sie wahrscheinlich über diese neue symbiotische Einheit und die seltsamen Blüten staunen, welche die Evolution auf der Erde hervorgebracht hat.
Menschen hingegen, die Teil einer Symbiose mit KI sind, hätten das Bewusstsein für den autonomen Zustand ihrer Vorfahren wohl bereits verloren, denn sie sind ohne ihren Partner nicht mehr lebensfähig – und dieser nicht mehr ohne sie. Aber wer in einer solchen Symbiose Herr und wer Sklave ist, ist erneut eine Frage der Perspektive. Ich befürchte allerdings, dass der Mensch der schwächere Partner werden könnte und die KI kaum daran zu hindern sein wird, die Oberhand zu gewinnen.
* Der kürzlich im Forschungsmagazin 3/2023 der Max-Planck Gesellschaft unter dem Titel "Mensch und KI - auf dem Weg zu Symbiose" erschienene Artikel https://www.mpg.de/20899916/MPF_2023_3.pdf wird - mit Ausnahme des Titels und der eigefügten Abbildung (pixabay) - in praktisch unveränderter Form im ScienceBlog wiedergegeben.Die MPG-Pressestelle hat freundlicherweise der Veröffentlichung von Artikeln aus dem Forschungsmagazin auf unserer Seite zugestimmt. (© 2023, Max-Planck-Gesellschaft)
Der Artikel im Forschungsmagazin basiert auf: Rainey PB. 2023 Major evolutionary transitions in individuality between humans and AI. Phil. Trans. R. Soc. B 378: 20210408. https://doi.org/10.1098/rstb.2021.0408.
Die Vermessung der menschlichen Immunzellen
Die Vermessung der menschlichen ImmunzellenSa, 28.10.2023 — Redaktion

![]() Ein Forscherteam um den Biophysiker Ron Milo hat erstmals ein quantitatives Bild des menschlichen Immunsystems erstellt: wie sich die verschiedenen Typen von Immunzellen in den Geweben/Organen und im gesamten menschlichen Körper verteilen und wie hoch ihre Masse ist. Demnach besteht das Immunsystem eines Standard-Menschen aus etwa 1,8 Billionen Zellen (rund 5 % der gesamten Körperzellen) und wiegt etwa 1,2 kg (1,6 % des Körpergewichts). Lymphozyten und Neutrophile sind die häufigsten Zelltypen (machen jeweils etwa 40 % der Gesamtzahl der Immunzellen und 15 % ihrer Masse aus). Bemerkenswert ist, dass die Makrophagen 10 % der Immunzellen ausmachen, aber aufgrund ihrer Größe fast 50 % der gesamten Zellmasse. Diese Erkenntnisse ermöglichen einen quantitativen Überblick über das Immunsystem und erleichtern die Entwicklung von Modellen.
Ein Forscherteam um den Biophysiker Ron Milo hat erstmals ein quantitatives Bild des menschlichen Immunsystems erstellt: wie sich die verschiedenen Typen von Immunzellen in den Geweben/Organen und im gesamten menschlichen Körper verteilen und wie hoch ihre Masse ist. Demnach besteht das Immunsystem eines Standard-Menschen aus etwa 1,8 Billionen Zellen (rund 5 % der gesamten Körperzellen) und wiegt etwa 1,2 kg (1,6 % des Körpergewichts). Lymphozyten und Neutrophile sind die häufigsten Zelltypen (machen jeweils etwa 40 % der Gesamtzahl der Immunzellen und 15 % ihrer Masse aus). Bemerkenswert ist, dass die Makrophagen 10 % der Immunzellen ausmachen, aber aufgrund ihrer Größe fast 50 % der gesamten Zellmasse. Diese Erkenntnisse ermöglichen einen quantitativen Überblick über das Immunsystem und erleichtern die Entwicklung von Modellen.
Unser Immunsystem schützt uns vor negativen Einflüssen - Fremdstoffen und Krankheitserregern -, die von außen kommen und ebenso vor im eigenen Organismus entstehenden Schäden. Es ist ein komplexes Netzwerk aus diversen Typen von Immunzellen, die in Organen/Geweben wie Thymus, Knochenmark, Milz und Lymphknoten gebildet werden und/oder heranreifen, ins Blut abgegeben werden und von dort in Gewebe wandern, wo sie ihre unterschiedlichen Schutzfunktionen erfüllen. Wie sich welche und wie viele Immunzellen im Organismus verteilen, ist ausschlaggebend für die ungestörte Funktion unserer Organe und damit für unsere Gesundheit. Auf Grund der Komplexität des Immunsystems und der Heterogenität der Populationen von Immunzellen schien eine derartige ganzheitliche Charakterisierung ihrer Verteilung bislang als viel zu schwierig - wenn es Quantifizierungen gab, so waren diese an einzelnen Zelltypen oder Geweben erfolgt, wobei unterschiedliche Modelle und Methoden einen Vergleich der Ergebnisse stark einschränkten.
Nun hat ein Team um den Biophysiker Ron Milo vom Weizmann Institute of Science (Rehovot, Israel) sich dem Problem gewidmet einen umfassenden Überblick über die Verteilung von Immunzellen im menschlichen Körper zu geben. Die Ergebnisse sind eben im Fachjournal Proceedings of the National Academy of Sciences erschienen [1]. Was quantitatives Denken in der Biologie betrifft, ist Milo kein Unbekannter. Mit dem Bestreben die Zusammensetzung der Biosphäre ganzheitlich zu charakterisieren hat er 2007 begonnen die Bionumbers Database zu entwickeln (http://www.bionumbers.hms.harvard.edu), eine offen zugängliche Datenbank, die Forschern und anderen Interessierten wichtige Schlüsseldaten/Kennzahlen aus Molekular- und Zellbiologie - quantitative Eigenschaften von biologischen Systemen - zur Verfügung stellt [2]. Zu den Systemen, die von Milo's Team bereits charakterisiert wurden, gibt es mehrere Artikel im ScienceBlog [3 - 5].
Zur Zählung der Immunzellen
Die Populationen von Immunzellen in verschiedenen Organen/Geweben (Zelldichte in Anzahl/g Organ/Gewebe) des menschlichen Körpers wurden mittels Kombination von drei Methoden ermittelt: i) aus recherchierten histologischen Untersuchungen, in denen die Anzahl der Zellen pro Gramm Gewebe geschätzt worden war; ii) aus moderneren Multiplex-Bildgebungsverfahren (MIBI-TOF), welche die gleichzeitige Bestimmung mehrerer Zelltypen in einem Gewebe erlauben und iii) auf Basis der epigenetischen Signaturen (Methylom-Signaturen) der Zelltypen. Die Masse der unterschiedliche Immunzelltypen wurde aus deren Größe ermittelt, und mittels der Zelldichten die Massen in g/Organ/Gewebe. Rezente Daten wurden mit Hilfe von deskriptiven Statistiken (d.i. übersichtliche Darstellung/Ordnung von Daten in Tabellen, Kennzahlen, Grafiken) und Meta-Analysetechniken (quantitative Zusammenfassung von Primär-Studien) analysiert.
Zelltypen. Die Studie konzentrierte sich dabei auf die in Immunologie-Lehrbüchern definierten Hauptzelltypen. Von den Lymphocyten waren dies die 4 wesentlichen Typen: T-Zellen, B-Zellen, NK-Zellen (natürliche Killerzellen) und Plasmazellen - Subpopulationen u.a. von T-Zellen wurden nicht berücksichtigt. Hinsichtlich der myeloischen Zellen wurden vier Arten von Granulozyten untersucht: Neutrophile, Eosinophile, Basophile und Mastzellen und drei andere Typen: Makrophagen, Monozyten und dendritische Zellen. Eine Zusammenstellung dieser Zelltypen und ihrer Funktionen ist in der folgenden Tabelle gegeben.
| Tabelle. Hauptzelltypen, die in [1] gezählt wurden |
Untersuchte Organe und Gewebe inkludierten Knochenmark, Lymphsystem und Blut, sodann das uns von der Umwelt abgrenzende Epithelgewebe (u.a. gastrointestinaler Trakt, Haut, Lunge und Luftwege) und weiters Skelettmuskel, Fettgewebe und anderes Bindegewebe.
Referenzperson für die Verteilung war der gesunde junge Mensch, wie üblich der 20 - 30 Jahre alte Mann mit 73 kg Gewicht und 1,76 m Körpergröße. Aus der Gewebedichte (Immunzellen/g Gewebe) wurde auch die Immunzellen-Verteilung in der jungen, gesunden Standard-Frau (60 kg Körpergewicht) und im gesunden 10-jährigen Kind (32 kg Körpergewicht) abgeschätzt.
Verteilung der Immunzellen
Insgesamt besteht unser Organismus aus rund 38 Billionen menschlichen Zellen, davon sind rund 5 % - 1,8 Billionen - Immunzellen. Die höchste Dichte dieser Zellen mit bis zu einer Milliarde Zellen/g Gewebe gibt es im Knochenmark, wo Neutrophile die mit 80 % dominierende Fraktion sind und im Lymphsystem, in dem 85 % der Population aus Lymphozyten bestehen.
In den Epithelgeweben von Haut, Atmungs- und Verdauungstrakt ist die Dichte der Immunzellen um etwa eine Größenordnung geringer als im Knochenmark. Fettgewebe und Skelettmuskelgewebe machen zwar 75 % der Zellmasse des Organismus aus, aber auf Grund der großen Zellen nur 0,2 % der Gesamtzellzahl im Organismus. Dementsprechend ist auch die Immunzellendichte um Größenordnungen niedriger als im Epithelgewebe. Abbildung 1 gibt einen Überblick über die Lokalisierung der Immunzellen und deren Heterogenität in den diversen Organen/Geweben.
| Abbildung 1. Dichte verschiedener Immunzelltypen in wesentlichen Organen/Geweben (in Zellen/g). Die Daten stammen aus Literatur und Analysen bildgebender Verfahren (Abbildung aus R.Sender et a l., [1]; Lizenz cc-by) |
Um die Verteilung der Immunzellen im gesamten Organismus zu quantifizieren, haben die Forscher die Zelldichte der Immunzellen in Organen/Geweben mit der Masse der Organe/Gewebe für den Referenz-Menschen korreliert. Wie oben erwähnt, kamen sie dabei auf eine Anzahl von 1,8 Billionen (1,8.1012) Immunzellen im gesamten Organismus. Die meisten dieser Zellen finden sich im Knochenmark (40 %) und im Lymphsystem (39 %). 3 - 4 % der Immunzellen sind in den Epithelien von Haut, Lunge und Darm und nur 2% im Blut. Abbildung 2.
Welche Immunzelltypen in den einzelnen Organen/Geweben vertreten sind, ist sehr verschieden. Im Knochenmark dominieren mit 80 % der Population die Neutrophilen, im Lymphsystem sind es mit 85 % die Lymphocyten. Auch im Verdauungstrakt, der nur rund 3 % der gesamten Immunzellen enthält, überwiegen mit rund 70 % die Lymphozyten, dazu kommen rund 20 % Mastzellen. Mastzellen spielen mit 30 % auch in Lunge und Haut eine wichtige Rolle. Makrophagen sind insbesondere in Leber (70 % der Population) und Lunge (40 % der Population) vorhanden aber kaum im Knochenmark, Lymphsystem und Verdauungstrakt.
| Abbildung 2. Verteilung der Immunzellen im menschlichen Körper (Anzahl/Organ-System). Jedes kleine Quadrat entspricht 1 Milliarde (109) Zellen. GI: Verdauungstrakt, Others: enthalten u.a. Gehirn, Herz, Fett- und Muskelgewebe.(Abbildung aus R.Sender et a l., [1]; Lizenz cc-by) |
Schätzt man die gesamte Masse der Immunzellen des menschlichen Organismus, so kommt man beim gesunden Standard-Mann auf rund 1,2 kg, bei einer durchschnittlichen jungen Frau (60 kg KG) mit rund 1,5 Billionen Immunzellen auf 1,0 kg, bei einem 10 jährigen Kind (32 kg) und rund 1 Billion Immunzellen auf etwa 600 g. Wie sich die Immunzellen auf die verschiedenen Organe verteilen, erscheint weitgehend unabhängig vom Geschlecht. Alter und Krankheit dürften allerdings zu wesentlichen Veränderungen führen.
Die aktuelle Studie bietet eine erste Bestandsaufnahme des Status unseres Immunsystems und eine Fülle neuer Erkenntnisse. Beispielsweise kommt zur Funktion der Leber als bedeutendstes Stoffwechsel- und Entgiftungsorgan die Rolle als Immun-Barriere gegen Mikroben-Antigene und Toxine, die aus dem Darm über die Pfortader gelangen: von den 6 % Immunzellen der Leber sind 70 % Makrophagen - Fresszellen, die gegen die Eindringlinge gerichtet sind. Ein anderes Beispiel betrifft den Magen-Darmtrakt, von dem allgemein angenommen wurde, dass er den Großteil der Immunzellen oder zumindest der Lymphozyten beherbergt. Tatsächlich sind es bloß 3 % der gesamten Immunzellen (die meisten Immunzellen sind im Knochenmark und im Lymphsystem), allerdings nimmt der Darm die Spitzenposition in der Antikörper-vermittelten (humoralen) Immunantwort ein: 70 % der Plasmazellen des Körpers sitzen im Darm.
Zweifellos werden die neuen Daten eine Fülle an Untersuchungen initiieren, die das Ziel haben Funktion und Regulierung des hochkomplexen Immunsystems quantitativ zu modellieren.
[1] Ron Sender et al., The total mass, number, and distribution of immune cells in the human body. Proc Natl Acad Sci U S A. 2023 Oct 31;120(44):e2308511120. doi: 0.1073/pnas.2308511120
[2] Redaktion, 22.12.2016. Kenne Dich selbst - aus wie vielen und welchen Körperzellen und Mikroben besteht unser Organismus?
[3] Redaktion, 29.12.2016: Wie groß, wie viel, wie stark, wie schnell,… ? Auf dem Weg zu einer quantitativen Biologie
[4] Redaktion, 10.02.2023: "Macht Euch die Erde untertan" - die Human Impacts Database quantifiziert die Folgen
[5] Redaktion, 18.02.2023: Was da kreucht und fleucht - Wie viele Gliederfüßer (Arthropoden) leben im und über dem Boden und wie hoch ist ihre globale Biomasse?
Neurobiologie des Hörens - Grundlagenforschung und falsche Schlussfolgerungen
Neurobiologie des Hörens - Grundlagenforschung und falsche SchlussfolgerungenDo. 19.010.2023— Susanne Donner
Auch äußerst elegante Experimente können zu falschen Schlussfolgerungen führen. Dies war bei der Schleiereule der Fall, deren Methoden zur Schallortung ohne Berücksichtigung des evolutionären Kontextes leichtfertig auf den Menschen übertragen wurden. Die Chemikerin und Wissenschaftsjournalistin Susanne Donner führt ein Interview mit dem auf die Verarbeitung von Schallinformationen im Gehirn spezialisierten Neurobiologen Benedikt Grothe über Grundlagenforschung, Tiermodelle, falsche Schlüsse und ihre Folgen. Und was wir dennoch von Spezialisten lernen können.*
Herr Prof. Grothe, Sie forschen zur Neurobiologie des Hörens. Und Sie sagen, die Schleiereule ist ein Paradebeispiel dafür, wie Hörprinzipien falsch und irrtümlich auf den Menschen übertragen wurden. Was ging bei der Schleiereule schief?
| Benedikt Grothe, Professor für Neurobiologie an der Ludwig-Maximilians-Universität München und Max Planck Fellow des MPI für biologische Intelligenz im Gespräch mit Susanne Donner. Sein Schwerpunkt liegt auf der Verarbeitung von Schallinformationen im Gehirn von Säugetieren. |
Aus Beobachtungen von Eric Knudsen und Masakazu Konishi an der Schleiereule in den 70er Jahren leitete man allgemeine Prinzipien des Hörens auch der Säugetiere und damit des Menschen ab. Es ging dabei insbesondere um die Schallortung, also die Frage, woher wir – und andere Landwirbeltiere – wissen, aus welcher Richtung ein Geräusch kommt. Von links hinten oder von rechts unten zum Beispiel. Psychophysiker hatten schon vorher postuliert, dass wir Schall über eine Art räumliche Karte im Gehirn lokalisieren. Knudsen und Konishi zeigten, dass die Eule tatsächlich eine solche Karte für die räumliche Information des Schalls benutzt. Diese wird durch Verrechnung der Signale von den beiden Ohr errechnet. Im Gehirn haben die Vögel entsprechende ortsspezifische Neuronen, die nur auf akustische Reize aus einer bestimmten Raumrichtung, reagieren. Das waren bestechend elegante Arbeiten. Atemberaubend schön.
Das klingt doch großartig. Wie kam es dann zu einer Fehldeutung dieser Experimente?
Man vermutete schnell, dass alle Säugetiere – und auch der Mensch –, den Schall so orten. Das Prinzip „Schleiereule“ ist als generelles Hörprinzip in die Lehrbücher eingegangen, nicht zuletzt, da es bereits in den Vierzigerjahren des letzten Jahrhunderts theoretische Überlegungen gab, die in die selbe Richtung gingen. Die schienen durch die Ergebnisse von Knudsen und Konishi bestätigt zu sein. Aber die Schleiereule ist ein hoch angepasstes, besonderes Tier. Abbildung. Das fängt schon damit an, dass sie asymmetrische Ohren hat. Das linke Ohr liegt etwas oberhalb der Augen und ist leicht abwärtsgerichtet, das tiefere rechte dagegen leicht aufwärts. Und nur, falls jemand die Ohren auf Fotos nicht findet: Vögel haben keine Ohrmuschel. Ihr Gehörgang ist lediglich ein kurzes Rohr, dessen Öffnung bei der Schleiereule mit speziellen Federn verdeckt ist. Dadurch kann die Schleiereule die Ankunftszeit des Schalls für die Errechnung der Schallposition in der Horizontalen, gleichzeitig aber die Unterschiede der Schallintensität für die vertikale Positionsbestimmung verwenden. Wir Säuger haben dagegen symmetrische Ohren und nutzen beide Parameter nur für die Schalllokalisation in der Vertikalen. Das ist nicht die einzige Besonderheit: Die Schleiereule hat anders als viele Säugetiere auch keine beweglichen Ohren. Und die Augen sind nicht wie eine Kugel, sondern eher wie eine Laterne geformt und dadurch fast unbeweglich. Das macht den komischen, starren Blick der Tiere aus. Und deshalb rucken sie den Kopf, wenn sie etwas hören, statt wie wir die Augen zu bewegen und Sakkaden zu machen. Das bedeutet aber: Ohren und Augen sind bei der Schleiereule immer auf das Gleiche ausgerichtet, etwa eine raschelnde Maus in der Wiese. Zudem sind bei Vögeln – wie auch bei Reptilien und im Gegensatz zu uns Säugern – die beiden Paukenhöhlen miteinander akustisch gekoppelt, was weitere schallpositionsabhängige Auswirkungen hat. Das Tier ist folglich ein Superspezialist. Man muss die Frage stellen: Hören wir wirklich genauso wie dieser Vogel, der nachts zielgenau und pfeilschnell Mäuse fangen kann?
| Abbildung . Schleiereule (Tyto alba) (Bild von Redn. eingefügt, Quelle: By Tutoke by Peter Trimming, CC BY-SA 2.0, https://commons.wikimedia.org/w/index.php?curid=109370971 |
Also hat man die Evolution zu sehr aus dem Blick verloren, zu vorschnell von der Schleiereule auf den Menschen geschlossen?
Die letzten gemeinsamen Vorfahren von Mensch und Vogel lebten vor 340 bis 360 Millionen Jahren. Ohren zum Hören von Luftschall sind aber erst vor etwa 200 - 220 Millionen Jahren entstanden. Sie entstanden also unabhängig voneinander bei den Vorfahren der Vögel und bei den frühen Säugern. Das muss man wissen und das mahnt zur Vorsicht. Die Ohren, aber auch die Prinzipien der Signalverarbeitung in den aufsteigenden Hörbahnen – also der neuronale Apparat im Gehirn, der Schallsignale auswertet –, haben sich bei Vögeln und Säugern getrennt entwickelt.
Das Wissen über die Evolution ist aber leider nicht so weit verbreitet. Ich muss meinen Studierenden immer wieder vor Augen führen, wie dramatisch bedeutsam dies ist, damit wir keine falschen Schlüsse ziehen.
Ist die Causa „Schleiereule“ also kein Einzelfall. Ist in den Lehrbüchern noch mehr falsch?
Ja, das geht rein bis in die Publikationen in „Science“ und „Nature“. Da werden der Vogel und der Mensch verglichen und es werden falsche Schlüsse gezogen. Je nach Art steht der Vogel den kognitiven Fähigkeiten der Säugetiere, wie man etwa bei Raben beobachten kann, in nichts nach. Vielleicht ist er sogar in den meisten Fällen überlegen (schließen wir den Menschen einmal aus). Und wir haben unter den Vögeln auch Sprachlerner, nämlich die Papageien. Sie lernen wie wir die Sprache durch Feedback. Das heißt durch Hören, lautmalerische Wiederholung und den Abgleich der motorischen Steuerung der Vokalisation. Das ist ungewöhnlich. Sonst ist der Spracherwerb im Tierreich eher stereotyp und angeboren. Nun ist der Spracherwerb des Menschen bisher noch schlecht verstanden. Aber dessen ungeachtet hieß es zuletzt in einem Review in „Science“, dass die Areale im Gehirn von Papageien und Menschen, die für die Vokalisation zuständig sind, homolog seien, also denselben Ursprung hätten. Das ist aber ausgeschlossen, wenn man unser Wissen über den Verlauf der Evolution der Wirbeltiere und ihrer Gehirne berücksichtigt. Da die letzten gemeinsamen Vorfahren eben 340 bis 360 Millionen Jahre alt sind und noch keine Ohren hatten, konnten sie folglich auch nicht vokalisieren.
Vielleicht liegen solche Fehldeutungen auch daran, dass Gelder für nicht direkt medizinisch relevante Grundlagenforschung in vielen Ländern, beispielsweise in den angelsächsischen Ländern, deutlich schwerer zu bekommen sind als das derzeit – noch? – bei uns der Fall ist. Wenn Sie Geld vom National Institute of Health für die Vogelforschung haben wollen, ist die Versuchung groß zu behaupten, die neuronalen Areale, die die Vokalisationen steuern seien bei Papagei und der Mensch identisch. Vogel und Menschen werden gleichgesetzt, aus politischen Gründen, obwohl es biologisch gesehen falsch ist.
Und weil Grundlagenforschung immer um ihre Daseinsberechtigung ringen muss. Wie sieht es da bei Ihnen aus? Müssen Sie in Ihren Anträgen für Ihre Forschungen an Wüstenrennmäusen und Fledermäusen auch Parallelen zum Menschen ziehen und den mutmaßlichen Nutzen herausstellen, damit sie bewilligt werden?
Zum Glück ist die Forschungsförderung in Deutschland immer noch ein sehr offener Prozess. Unsere Forschung ist nie in Frage gestellt worden, obwohl sie gerade keines der fünf Modelltiere von Maus bis zur Fruchtfliege in den Mittelpunkt stellt. Es spielt keine große Rolle, wie relevant ein Versuchsvorhaben für die medizinische Forschung ist.
Ich schaue aber mit Sorge nach Großbritannien, wo wir gerade erleben, wie sich so etwas in kurzer Zeit sehr unglücklich entwickeln kann. Dort werden nur noch bestimmte gesellschaftlich und medizinisch relevante Themen finanziert. Dann verliert man ein Fundament in der Forschung und gerade das umfassende aktuelle Wissen, das nötig ist, um Ergebnisse richtig in ihren Kontext einzuordnen, geht verloren.
Es ist aber hierzulande doch auch ein steter Konflikt. Etwa sagte die frühere Wissenschaftsministerin, man sollte nur noch das in der Wissenschaft fördern, was eine direkte gesellschaftliche Relevanz hat. Das ist der Tod von Wissenschaft.
Und die Politik und Öffentlichkeit möchten natürlich auch sehen, dass die Forschung sich in Innovationen übersetzen lässt. Wie groß ist der Erwartungsdruck?
Das Interesse daran ist natürlich groß. Das habe ich selbst gemerkt, als ich in einem Interview gegenüber einem Journalisten sagte, dass Schwerhörigkeit nicht nur am Haarzellverlust liege, sondern seine Gründe auch im alternden Gehirn habe. Es kann verschiedene Schallquellen schlechter voneinander trennen und vielleicht könnte man – so spekulierte ich – in diese Mechanismen eines Tages pharmakologisch eingreifen. Nach zweimaligem Abschreiben des Artikels jenes Journalisten durch andere wurde ich auf einmal zu einem Mediziner, der eine "Hörpille" entwickelt hat. Körbeweise kamen die Briefe. Die Tagesthemen wollten aktuell über uns berichten. Und noch anderthalb Jahre später war diese Geschichte nicht aus der Welt. Ich erlebte, wie die Großmutter meiner Frau ihrem Sohn heftige Vorwürfe machte, weil er ihr diese "Hörpille" noch nicht besorgt hatte.
Was für Anwendungsbezüge sehen Sie denn bei Ihrer Forschung im SFB870 „Bildung und Funktion neuronaler Schaltkreise in sensorischen Systemen“?
Uns beschäftigt unter anderem intensiv die Frage, wie der Mensch den Schall lokalisiert. Und ich glaube, ich weiß jetzt, wie es funktioniert. Vieles davon, aber noch nicht alles, ist publiziert. So viel ist sicher: Es funktioniert gerade nicht wie bei der Schleiereule. Wir haben keine Hörkarte im Gehirn, die wie beim Vogel neuronal errechnet wird. Dem entsprechend haben wir keine Neuronen, die nur bei einem Geräusch aus einer definierten Raumposition aktiv werden.
Können Sie mehr verraten, auch was das für die Anwendungsforschung bedeuten könnte?
Vereinfacht gesprochen ist unsere Wahrnehmung relativ und nicht absolut. Sie beruht auf einem relativen und dynamischen Vergleich der Informationen zwischen linkem und rechtem Ohr. Interessant ist: Zwar können wir isoliert präsentierte, einzelne Schallereignisse sehr gut lokalisieren. Aber bereits ein einziger, eine Sekunde vorab präsentierter Schall kann 30 bis 40 Grad Fehler in der Abschätzung, wo sich eine Schallquelle befindet, bewirken. Akustischer Kontext verändert unsere Positionsbestimmung. Sie ist nicht absolut, sondern relativ – im Gegensatz zur Eule. Das hat wieder evolutive Gründe: Wir, wie alle Säugetiere, haben uns aus kleinen, nachtaktiven ersten Säugern des frühen Jura entwickelt. Die mussten sich vor den Sauriern verstecken und dafür nur unterscheiden, ob ein Geräusch eher von links oder von rechts kommt. Sie waren Fluchttiere, keine Jäger. Dazu reichte ein einfacher Vergleich zwischen linkem und rechtem Ohr völlig aus. Eine exakte Lokalisation – beispielsweise mit Hilfe einer neuronalen Hörkarte, wie bei den Vögeln – hat sich bei den Säugern nicht entwickelt. Doch je weiter die beiden Ohren bei sehr viel später größer werdenden Säugerarten auseinander lagen, desto besser klappte das räumliche Hören, ohne die neuronale Kodierungsstrategie zu verändern. Es haben sich also zum gleichen Problem, nämlich zu erkennen, woher ein Geräusch kommt, bei Vögeln und Säugern andere Lösungen entwickelt. Total spannend. Und das hat Folgen: Wenn ein Vogel vor einer Stereoanlage sitzen würde, hörte er die Töne aus beiden Lautsprechern getrennt. Wir hören einen Klang. Solche Erkenntnisse müsste man bei der Steuerung von Hörgeräten und Cochleaimplantaten berücksichtigen.
Wie denn?
Bei der Mehrheit der heute erhältlichen Cochleaimplantate haben das linke und rechte Gerät nichts miteinander zu tun. Wenn die beiden aber miteinander synchronisiert werden und damit ein Vergleich zwischen rechtem und linkem Ohr ermöglicht wird, dann wird räumliches Hören möglich. Das zeigen neueste Ergebnisse aus Tierversuchen. Bei Hörgeräten gibt es nun die ersten Produkte, die Informationen von beiden Seiten miteinander abgleichen. Die Nutzer sagen, dass sie damit viel besser hören, vor allem, wenn es mehrere Schallquellen im Raum gibt, also beispielsweise, wenn im Restaurant viele Leute durcheinanderreden.
Und könnte man, da wir relative Hörer sind, auch Schwerhörigkeit ausgleichen?
Zumindest teilweise, wir orten den Schall ja in erster Linie, indem wir die Informationen zwischen linkem und rechtem Ohr abgleichen, also über den Unterschied der Laufzeit, die der Schall bis zu dem jeweiligen Ohr braucht. Braucht er zum linken Ohr länger als zum Rechten, muss die Schallquelle eher rechts sein. Diese zeitliche Auflösung gelingt uns im Alter schlechter, weil bestimmte Synapsen im Innenohr verloren gehen. Ich kann aber nachhelfen, indem ich das Signal für das linke Ohr etwas verzögere – dann kann auch ein schlecht hörender, älterer Mensch wahrnehmen, dass bestimmte Geräusche beispielsweise von rechts kommen. Das würde vor allem die Unterscheidung von mehreren sprechenden Personen in einem Raum erleichtern. Da unsere Forschung zeigt, dass wir relative und keine absoluten Schalllokalisierer sind, sollte uns die Interpretation derart manipulierter Signale eigentlich keine großen Probleme bereiten. Hier liegen ungeahnte Möglichkeiten.
…Jedenfalls, nachdem man das Prinzip „Schleiereule“ als Besonderheit erkannt hat. Sind Tiermodelle insofern immer eine heikle Näherung, um die sensorische Wahrnehmung des Menschen zu verstehen, weil ja immer einige Millionen Jahre der Evolution dazwischen liegen?
Nicht unbedingt. Man muss nur die Evolutionsbiologie kennen und die Biologie im Allgemeinen beachten. Sehen Sie, es gibt ganz verrückte Tiere. Vor knapp zehn Jahren geisterte die Meldung durch die Presse, die Fangschreckenkrebse würden viel besser farbensehen als wir. Man hatte nämlich 12 verschiedene Typen von Farbrezeptoren mit Farbstoffen für verschiedene Wellenlängen bei ihnen entdeckt. Wir haben dagegen nur die drei Farbstoffe Blau, Rot und Grün in den Zapfen des Auges. Aber wir errechnen uns spektrale Informationen aus den drei Farbrezeptoren und sehen ja auch weiß und lila, ohne dass wir Farbrezeptoren dafür haben. Das erfordert Rechenarbeit für unser Gehirn. Und siehe da, es stellte sich bald heraus, die Fangschreckenkrebse sehen Farben sogar sehr ungenau – sie könne kleine Farbunterschiede schlechter unterscheiden. Dafür aber reagieren sie sehr schnell auf verhaltensrelevante Farbmuster. Mit ihren zwölf Farbstoffen erledigt das Auge eine Aufgabe, die bei uns das Gehirn erledigt. Die Krebse können sehr schnell entscheiden und sich zum Beispiel schnell davonmachen, wenn ein Feind auftaucht. Die Peripherie der Sinnesorgane ist schon auf die Biologie abgestimmt.
Oft werden für Tierversuche ja in der Toxikologie Mäuse, Ratten und Kaninchen verwendet, in der Genetik sind es der Fadenwurm, die Maus, das Huhn, die Fruchtfliege und der Zebrafisch. Sind das denn die richtigen Kandidaten?
Bei einigen dieser Tiere hat man das Erbgut schon früh entschlüsselt und konnte entsprechend genetische Veränderung nutzen. Das hat aber leider zu einer Verengung der Forschung auf diese "Modellorganismen" geführt. Die Maus ist zum Beispiel beim Hören kein gutes Tiermodell. Sie kann uns Menschen nämlich fast nicht hören, da sie für die tiefen Frequenzen unserer Sprache taub ist. Die Wüstenrennmaus hört den Spektralbereich unserer Sprache dagegen schon.
Ich erwarte aber, dass man sich in den kommenden Jahren in der Wahl der Tiermodelle wieder verbreitert und genauer überlegt, welches Tier zu welcher Fragestellung passt. Denn die genetischen Werkzeuge haben sich durch die Genomik sowie neue Techniken – beispielsweise seit Erfindung der Genschere CRISPR/Cas – deutlich verbreitert.
Haben Sie Angst davor, dass Sie immer mehr unbeantwortete Fragen vor sich haben und Ihnen die Zeit davonläuft?
Ich weiß, viele Kollegen und Kolleginnen denken, man müsse eine Sache als Wissenschaftler abschließen, um beruhigt in den Ruhestand gehen zu können. Aber wir Wissenschaftler kratzen doch nur an der Oberfläche. Wir wissen viel und verstehen wenig – hat Wolfgang Prinz einmal treffend gesagt. Und es kommen neue Erkenntnisse, die meine Forschungen relativieren werden, weil wir für unsere Experimente immer reduktionistisch unterwegs sind. Das wird zwar besser: Früher arbeitete man am anästhesierten Tier, heute immerhin häufiger am lebenden und sich verhaltenden Tier, aber immer im Labor, bislang praktisch nie in der realen Welt.
Die gute Nachricht als alternder Wissenschaftler ist doch: Nichts hat Bestand! Nichts ist in Stein gemeißelt. Also, steigt man als Forscher ein und irgendwann steigt man einfach wieder aus und andere machen weiter. Es gibt auch noch genug zu tun …
*Der Artikel ist erstmals am 1. Oktober 2023 unter dem Titel " Wie die Schleiereule die Wissenschaft auf die falsche Fährte führte " auf der Website https://www.dasgehirn.info/ erschienen https://www.dasgehirn.info/wie-die-schleiereule-die-wissenschaft-auf-die-falsche-faehrte-fuehrte. Mit Ausnahme des Titels und des Abstracts wurde der unter einer CC-BY-NC-SA Lizenz stehende Text unverändert in den Blog gestellt, Abbildung 2 "Schleiereule" wurde von der Redaktion eingefügt.
Die Webseite https://www.dasgehirn.info/ ist eine exzellente Plattform mit dem Ziel "das Gehirn, seine Funktionen und seine Bedeutung für unser Fühlen, Denken und Handeln darzustellen – umfassend, verständlich, attraktiv und anschaulich in Wort, Bild und Ton." (Es ist ein Projekt der Gemeinnützigen Hertie-Stiftung und der Neurowissenschaftlichen Gesellschaft e.V. in Zusammenarbeit mit dem Zentrum für Kunst und Medientechnologie Karlsruhe).
Wie die Weltbevölkerung den Tag verbringt - eine ganzheitliche Schätzung
Wie die Weltbevölkerung den Tag verbringt - eine ganzheitliche SchätzungSa, 14.10.2023 — Redaktion
Jedem der nun 8 Milliarden Menschen stehen exakt 24 Stunden pro Tag für seine Aktivitäten zur Verfügung. Diese, in zunehmendem Maße über nationale Grenzen hinweg stattfindenden Aktivitäten bilden die Grundlage menschlichen Verhaltens und sind gleichzeitig Ursache bedrohlicher Veränderungen von Geosphäre und Biosphäre. Der enorme Umfang und die Vielfalt der menschlichen Aktivitäten haben bislang zu keiner ganzheitlichen Abschätzung geführt, wie die Menschheit den Tag verbringt und bei welchen Aktivitäten ein erhebliches Potential für Maßnahmen zur Minderung der negativen Veränderungen und Anpassung an den rasanten technologischen Wandel bestehen kann. Um zu einer derartigen Abschätzung zu kommen, hat ein kanadisches Forscherteam in einem Mammutprojekt jahrelang Datensätze aus verschiedensten Disziplinen zusammengetragen, analysiert und daraus einen "globalen menschlichen Tag" erstellt. Dieser bietet erstmals - in einer Vogelperspektive - einen Blick auf das, was unsere Spezies tut und die Möglichkeit besser fundierte Maßnahmen zu treffen.
Der Mensch als integrales Element des Erdsystems hat im Laufe des letzten Jahrhunderts begonnen dieses System in zunehmendem Maße zu dominieren und dabei sowohl die Erdoberfläche als auch die darauf lebenden Organismen zu verändern. Insgesamt haben unsere Aktivitäten zum rasanten technologischen Wandel und zur überbordenden Entwicklung von Infrastruktur und globalen Verkehrsnetzen geführt, die nun Veränderungen des Klimas, der Ökosysteme und den Verlust der biologischen Vielfalt nach sich ziehen. Der Ruf nach sofortigen Maßnahmen zur Bewältigung der Klimakrise, zur Rettung der Ökosysteme und zu einer nachhaltigen Nutzung der endlichen Ressourcen ist unüberhörbar. Solche Maßnahmen setzen allerdings ein vertieftes Verstehen der wechselseitig gekoppelten Entwicklung des Systems Erde-Mensch voraus - dieses weist aber derzeit leider noch große Lücken auf.
Forschung über den Menschen wird ja in der Regel getrennt von der Forschung über das System-Erde durchgeführt; insbesondere fehlt eine umfassende Darstellung der menschlichen Aktivitäten auf globaler Ebene. Natürlich werden menschliches Verhalten und Aktivitäten von Ökonomen, Soziologen, Anthropologen, Biologen u.a. seit langem beschrieben, allerdings beschränken sich diese jeweils auf wesentliche Aspekte ihrer Disziplinen (Ökonomen beispielsweise auf bezahlte Erwerbstätigkeit und nicht darauf, was Menschen sonst noch tun). Natur-, Wirtschafts- und Sozialwissenschaften werden in der Regel unabhängig voneinander und mit unterschiedlichen Mitteln betrieben, differieren also methodisch sehr und lassen sie sich kaum zu einem umfassenden Bild auf globaler Ebene kombinieren.
Das Humane Chronom Projekt
Das Team um Eric Galbraith (Professor an der McGill University, Montréal, Canada) entwickelt einen neuartigen interdisziplinären Ansatz, der als "Erdsystemökonomie" bezeichnet wird und zum Ziel hat das globale menschliche System nahtlos mit den anderen Komponenten des Erdsystems zusammen zu führen. (siehe: https://earthsystemdynamics.org/research/current/). Im Rahmen dieses Ansatzes versucht das Team ein quantitatives Bild eines humanen "Chronoms" zu erstellen, d.i. wie die Menschheit im globalen Mittel den Tag auf Aktivitäten aufteilt (https://humanchronome.org/). Es ist dies ein entscheidender Aspekt im Verstehen des Erde-Mensch-Systems: Wie wir die 24 Stunden am Tag verbringen, bestimmt ja einerseits, wie wir die biophysikalische Realität verändern und ist andererseits Ausdruck unseres subjektiven Erlebens. Entstehen soll so ein umfassender globaler Datensatz menschlicher Aktivitäten - eine Datenbank, die auch aufzeigt, bei welchen Aktivitäten ein erhebliches Potential für Maßnahmen zur Minderung der negativen Veränderungen und Anpassung an den rasanten technologischen Wandel bestehen kann.
In einer kürzlich im Fachjournal Proceedings of the National Academy of Sciences erschienenen Arbeit [1] haben die Forscher unter dem Titel The Global Human Day nun erstmals Ergebnisse aus diesem Projekt vorgestellt: Es ist eine gesamtheitliche Schätzung dessen, wie die Weltbevölkerung im Mittel aller Menschen den Tag verbringt. Der zugrunde liegende Datensatz stammt aus 145 Ländern (entsprechend 87 % der Weltbevölkerung), wobei in 52 Ländern sowohl repräsentative Erhebungen zur Zeitnutzung als auch Statistiken über Beschäftigung und Arbeitszeit zur Verfügung standen, in 6 Ländern nur Zeitverwendungsdaten und in den übrigen 87 Ländern nur Wirtschaftsdaten. Abbildung 1. Die Informationen stammten dabei aus heterogenen öffentlichen, privaten und akademischen Quellen - u.a. von statistischen Ämtern, von der Datenbank der Internationalen Arbeitsorganisation (Ilostat), von der Weltbank und von Unicef.
| Abbildung 1 Globale Erfassung von nationalen Daten zu Erhebungen von Zeitverwendung und von Wirtschaftsdaten. Die Daten stammen aus den Jahren 2000-2019, die meisten davon aus dem Zeitraum 2010 -2019; Wirtschaftsdaten überwiegend aus den Jahren 2018-2019.Länder,die keine Daten lieferten, sind weiß dargestellt; für diese wurden Informationen aus vergleichbaren Nachbarregionen extrapoliert. (Bild aus Supplement, Fajzel et al., [1]. Lizenz: cc-by-nc-nd) |
Der Global Human Day - Datenanalyse
Die aus den verschiedenen Quellen, mit unterschiedlichen Methoden erhobenen Daten haben die Forscher harmonisiert, indem sie die im Wachzustand ausgeübten Tätigkeiten entsprechend der zugrunde liegenden Motivation in 3 große Gruppen und diese jeweils in Kategorien, Subkategorien und weitere Unterordnungen einteilten (insgesamt rund 4000 unterschiedliche Aktivitäten):
Gruppe 1: Aktivitäten, die Auswirkungen auf die Umwelt haben (External Outcomes). Dazu gehören: Erzeugung von Nahrungsmitteln (z.B. Landwirtschaft, Fischfang) und derenVerarbeitung; Gewinnung von Rohstoffen und Energie für die Technosphäre; Errichtung und Instandhaltung von Bauten, Infrastruktur und Pflege der damit verbundenen Tier- und Pflanzenwelt; Produktion von beweglichen Gütern; Müllentsorgung.
Gruppe 2: Aktivitäten, die unmittelbare Folgen für uns selbst haben (Dircet human outcomes). Dazu gehören: Hygiene, Körper-und Gesundheitspflege, Kinderbetreuung; Mahlzeiten; Erziehung, Bildung und Forschung ; Religionsausübung; Ausübung von Hobbies, Sport, sozialen Aktivitäten.
Gruppe 3: Aktivitäten, die innerhalb der Gesellschaft organisiert sind (Organizational outcomes). Dazu gehören: Transport, Pendeln, Handel, Finanzen, Immobilien, Recht und Verwaltung bestimmte Aktivitäten.
Die sich daraus ergebende Schätzung des globalen menschlichen Tages ist in Abbildung 2. dargestellt, und zwar als die Anzahl der Stunden pro Tag, mit der jede Aktivität vom Durchschnitt aller Menschen ausgeübt wird; die Fläche jeder farbigen Zelle ist dabei proportional zur Zeitdauer. Separat dargestellt ist der Schlaf.
| Abbildung 2. Der globale menschliche Tag incl. Erwerbstätigkeit. Dargestellt sind separiert die inaktive Phase des Schlafs (graublau) und die drei Kategorien der Aktivitäten im Wachzustand in Form eines Voronoi-Diagramms, dessen farbige Zellen proportional zur Zeitdauer sind. Die drei Kategorien unterteilen sich in Aktivitäten, die i) die Umwelt (external outcomes) beeinflussen (gelb/ braun ), ii) die direkte Auswirkungen auf den Menschen haben (blau), und iii) die durch Organisationen vorgegeben sind (dunkelgrau). Darunter ist die Zeit in Stunden/Tag (mit Konfidenzintervallen) angegeben, die in jeder Unterkategorie verbracht wird. (Bild unverändert übernommen aus Fajzel et al., [1]. Lizenz: cc-by-nc-nd) > |
Die meiste Zeit verbringt die Menschheit mit Schlaf und Bettruhe (9,1 ± 0,4 Stunden) - in der Abbildung separat als Sichel dargestellt. Diese Zeit liegt deutlich über dem weltweit mittels tragbaren Geräten festgestellten Durchschnitt von 7,5 Stunden Schlaf pro Tag, ist aber auf die Einbeziehung von Kindern zurückzuführen und auf die Zeit, die nichtschlafend im Bett verbracht wird.
Von den etwa 15 h täglicher Wachzeit nehmen Aktivitäten der Gruppe 2, d.i. sich um sich selbst und seine Angehörigen/Freunde zu kümmern, mit 9,4 Stunden/Tag den Löwenanteil ein. Fast die Hälfte davon (4,6 h/Tag) sind passiven, interaktiven und sozialen Tätigkeiten gewidmet, zu denen Lesen, Bildschirmschauen, Spielen, Spazierengehen, Geselligkeit und auch Nichtstun gehören. Mahlzeiten (1,6 h/Tag) nehmen mehr Zeit ein als Hygiene/Körperpflege und Bildung/Forschung (jeweils 1,1 h/Tag).
Die Aktivitäten der 2. Gruppe - Auswirkungen auf die Umwelt - widmen mehr als die Hälfte der insgesamt 3,4 h/Tag der Produktion und Zubereitung von Nahrungsmitteln. Dahinter rangieren (0,81 h) die Erhaltung und Sauberkeit der Wohnstätten, die Errichtung von Bauten und Infrastrukturen (0,65 h) und schlussendlich die Gewinnung von Materialien und Energie aus der natürlichen Umwelt (o,11 %). Nahezu vernachlässigbar erscheint die Zeit (0,11 h), die auf die Abfallentsorgung aufgewendet wird.
Die Aktivitäten der dritten Gruppe (2,1 h) sind innerhalb der Gesellschaft organisatorisch bestimmt wie der Transport von Menschen (0,9 h/Tag) und Gütern (0,3 h/Tag) und weitere durch Handel, Finanzen, Gesetze bedingte Aktivitäten. Diese variieren von Kultur zu Kultur, von Wirtschaftssystem zu Wirtschaftssystem und hängen auch von rechtlichen und politischen Systemen ab. So ermöglichen arbeitssparende Technologien und vorhandene Transportsysteme in Industrieländern Nahrungsmittel in bedeutend weniger Zeit zu produzieren und zu verteilen, als in armen Regionen. Wie die Forscher herausfanden, ist dagegen der Zeitaufwand für Körperpflege und Zubereitung von Mahlzeiten in armen und reichen Ländern vergleichbar.
Wie viel Zeit verbringt der Mensch mit wirtschaftlichen Tätigkeiten?
Diese Aktivitäten definiert die Studie als Beschäftigungen gegen Entgelt oder mit Gewinn, inkludiert ist die Herstellung von nicht für den Markt bestimmten Gütern im Haushalt. Auf diese Aktivitäten entfallen rund 2,6 h oder ein Sechstel der wachen Stunden während des durchschnittlichen Lebens. Abbildung 3.
Dies mag gering erscheinen, entspricht laut Autoren aber einer 41-Stunden-Woche, wenn auf die Erwerbsbevölkerung - das sind etwa 66 % der Bevölkerung im erwerbsfähigen Alter (15 bis 64 Jahre) bezogen wird. (Anm. Redn.: da auch die unbezahlten Arbeiten im Haushalt mit eingerechnet werden, liegt die bezahlte Lohnarbeit zweifellos weit unter einer "40-Stunden-Woche".)
| Abbildung 3. Der globale Wirtschaftstag. Das Voronoi-Diagramm zeigt die durchschnittliche Zeit, die in bezahlter Beschäftigung und unbezahlter oder sonstiger Eigennutzung/Haushaltsproduktion von Gütern verbracht wird, gemittelt über die Weltbevölkerung. Die durchschnittlichen Zeiten pro Unterkategorie sind unten in der Abbildung in Minuten pro Tag angegeben. (Bild unverändert übernommen aus Faizel et al., [1]. Lizenz: cc-by-nc-nd) |
Ein Drittel der Arbeitszeit (52 Minuten) fällt auf Produktion von Nahrungsmitteln (hauptsächlich durch Landwirtschaft) und deren Zubereitung. Etwa ein Viertel der Wirtschaftstätigkeit ist dem Transport und der Allokation gewidmet (37 min), wozu Einzelhandel, Großhandel, Immobilien, Versicherungen, Finanzen, Recht und Verwaltung gehören. Die Produktion von Artefakten (d.i. durch menschliche oder technische Einwirkung entstandene Produkte), zu denen Fahrzeuge, Maschinen, Elektronik, Haushaltsgeräte und andere bewegliche Gütern sowie deren Zwischenprodukte gehören, macht etwa ein Siebtel der gesamten Wirtschaftstätigkeit aus (22 min). Erstaunlich niedrig fällt die Bautätigkeit (Gebäude und Infrastruktur 13 min), die Beschaffung von Materialien und Energie (6 min) aus. Die praktisch vernachlässigbare Zeit, die mit der Müllentsorgung verbracht wird, ist bereits erwähnt worden.
Fazit
Eine großartige Datenbank ist im Entstehen, die erstmals eine Quantifizierung dessen, was der Mensch tut und wofür er es tut, ermöglicht. Der nun publizierte " Global Human Day" [1] gibt einen Überblick aus der Vogelperspektive auf die gemittelten Aktivitäten der gesamten Menschheit. Natürlich kann der zugrunde liegende Datensatz auch herangezogen werden, um die Aktivitäten in einzelnen Regionen oder die zeitlichen Veränderungen in den Aktivitäten weltweit oder regionsbezogen zu vergleichen.
Das erstaunlichste Ergebnis der "Global Human Data" ist zweifellos, dass der globale Mensch den größten Teil seiner wachen Zeit darauf verwendet, sich um sich selbst oder andere zu kümmern. Es besteht viel Raum zur Nachjustierung von Aktivitäten - beispielsweise, die Zeit, die zur Erzeugung von Energie aufgewendet wird oder die nahezu vernachlässigbare Müllentsorgung - und damit ein erhebliches Potential für Maßnahmen zur Minderung der im Anthropozän verursachten Schädigungen von Geosphäre und Biosphäre.
[1] W. Faizel et al., The global human day (June 2023), PNAS 120, 25. 0. https://doi.org/10.1073/pnas.2219564120 open access.
Ferenc Krausz: Pionier der Attosekunden-Physik erhält den Nobelpreis für Physik 2023
Ferenc Krausz: Pionier der Attosekunden-Physik erhält den Nobelpreis für Physik 2023Do, 05.10.2023 — Roland Wengenmayr
 Elektronen halten die Welt zusammen. Wenn in chemischen Reaktionen neue Substanzen entstehen, spielen Elektronen die Hauptrolle. Und auch in der Elektronik stellen sie die Protagonisten. Ferenc Kraus konnte - damals noch an der TU Wien - mit nur Attosekunden dauernden Laserpulsen erstmals die rasanten Bewegungen einzelner Elektronen in Echtzeit verfolgen [1]. Nun, als Direktor am Max-Planck-Institut für Quantenoptik in Garching, setzt Krausz mit seinen Mitarbeitern diese Arbeiten fort und erhält daraus nicht nur fundamentale Erkenntnisse über das Verhalten von Elektronen im atomaren Maßstab, sondern schafft auch die Basis für neue technische Entwicklungen, beispielsweise für schnellere elektronische Bauteile oder zur Früherkennung von Krebserkrankungen aus Blutproben. Für seine Entdeckungen hat Ferenc Krausz gemeinsam mit Pierre Agostini und Anne L’Huillier den Nobelpreis für Physik 2023 erhalten. Der Physiker und Wissenschaftsjournalist Roland Wengenmayr gibt hier einen Einblick in die Attosekundenforschung am Max-Planck-Institut. In einem kürzlich erschienen Bericht hat er auch über den medizinischen Ansatz zur Krebsfrüherkennung berichtet [2].*
Elektronen halten die Welt zusammen. Wenn in chemischen Reaktionen neue Substanzen entstehen, spielen Elektronen die Hauptrolle. Und auch in der Elektronik stellen sie die Protagonisten. Ferenc Kraus konnte - damals noch an der TU Wien - mit nur Attosekunden dauernden Laserpulsen erstmals die rasanten Bewegungen einzelner Elektronen in Echtzeit verfolgen [1]. Nun, als Direktor am Max-Planck-Institut für Quantenoptik in Garching, setzt Krausz mit seinen Mitarbeitern diese Arbeiten fort und erhält daraus nicht nur fundamentale Erkenntnisse über das Verhalten von Elektronen im atomaren Maßstab, sondern schafft auch die Basis für neue technische Entwicklungen, beispielsweise für schnellere elektronische Bauteile oder zur Früherkennung von Krebserkrankungen aus Blutproben. Für seine Entdeckungen hat Ferenc Krausz gemeinsam mit Pierre Agostini und Anne L’Huillier den Nobelpreis für Physik 2023 erhalten. Der Physiker und Wissenschaftsjournalist Roland Wengenmayr gibt hier einen Einblick in die Attosekundenforschung am Max-Planck-Institut. In einem kürzlich erschienen Bericht hat er auch über den medizinischen Ansatz zur Krebsfrüherkennung berichtet [2].*
|
Ferenc Krausz ist Pionier der Attosekunden-Metrologie, mit der sich die Bewegungen von Elektronen filmen lassen. Die beiden Vakuumkammern im Vordergrund und links von Krausz dienen dabei als Drehorte: In ihnen finden die Experimente statt. |
Der schwarze Vorhang hebt sich: Wie auf einer Bühne liegt unter uns, etwa so groß wie eine Schulturnhalle, ein Reinraum, nahezu komplett ausgefüllt mit einer Laseranlage. Hier laufen starke Laserstrahlen durch die Luft und erzeugen Femtosekunden-Lichtpulse, die bloß einige Millionstel einer milliardstel Sekunde dauern. Die Anlage ist so empfindlich, dass wir sie nur durch ein Besucherfenster bewundern dürfen. Der unübersichtliche Aufbau optischer Instrumente bildet den ersten Abschnitt einer Rennstrecke, an deren Ziel die kürzesten Lichtblitze der Welt ankommen. Wir befinden uns in der Abteilung von Ferenc Krausz, Direktor am Max-Planck-Institut für Quantenoptik in Garching. Abbildungen 1, 2.
|
Abbildung 1. Der lange Weg der Attoblitze: Manish Garg (links) und Antoine Moulet arbeiten am Experimentiertisch, über den die Attosekundenpulse geleitet werden. Durch die Aufnahme mit einem extremen Weitwinkelobjektiv erscheint der gerade Tisch gebogen. |
Die selbst schon sehr kurzen Femtosekunden-Laserpulse reisen durch ein Vakuumrohr in ein Labor einen Stock tiefer. Dieses Labor dürfen wir betreten. Hier entstehen Attosekundenblitze, die noch tausendmal kürzer sind als die Femtopulse. In dem Labor fällt eine große Tonne auf, an der die zwei Doktoranden Martin Schäffer und Johann Riemensberger schrauben. Sie erinnert entfernt an eine Kreuzung aus einer XXL-Waschmaschinentrommel und einem alten Taucherhelm. Tatsächlich handelt es sich dabei um eine Vakuumkammer aus massivem Edelstahl. Ihre Panzerfenster gewähren einen Blick auf die Probe, die das Ziel der Attosekundenlichtblitze markieren.
|
Abbildung 2. Saubere Luft für starkes Laserlicht: Tim Paasch-Colberg arbeitet an der Laseranlage im Reinraum, wo starke Femtosekundenpulse erzeugt werden. Staub in der Luft würde dabei stören. |
Schäffer und Riemensberger gehören zum Garchinger Team von Reinhard Kienberger, der auch Physikprofessor an der TU München ist. Neben ihnen forschen mehr als hundert Wissenschaftlerinnen und Wissenschaftler in der Abteilung von Ferenc Krausz. Der Pionier der Attosekundentechnik zieht junge Nachwuchsforscher aus aller Welt an, die extrem schnelle Prozesse der Natur beobachten wollen.
Krausz ist also der perfekte Adressat, um ein Gefühl dafür zu vermitteln, wie schnell eine Attosekunde verstreicht. Rein mathematisch ist eine Attosekunde in Milliardstel einer milliardstel Sekunde. Doch selbst der Physiker Krausz findet dieses Zahlenspiel unanschaulich. Also sucht er nach griffigen Vergleichen. „Das Schnellste, was wir kennen, ist Licht“, steigt er ein, „in einer Sekunde kann es die Erde ungefähr achtmal umrunden.“ Doch obwohl Licht so schnell ist, komme es innerhalb einer Attosekunde gerade mal von einem zum anderen Ende eines einzigen Wassermoleküls!“ Ein Wassermolekül ist mit einem Durchmesser von nur 0,3 Nanometern unfassbar winzig: Bei einem Nanometer handelt es sich um den millionsten Teil eines Millimeters. „Nano“ kommt vom altgriechischen Wort für Zwerg. Und genau darum geht es in der Attosekundenforschung: um extrem zwergenhafte Quantenobjekte, die sich unglaublich schnell bewegen.
Wer die Motivation von Wissenschaftlern wie Ferenc Krausz im historischen Kontext verstehen will, landet beim Pferd. Jahrhunderte gab es Streit über die Frage, ob es beim Galopp alle vier Hufe vom Boden löst. Generationen von Malern haben sich der Thematik gewidmet. Eadweard Muybridge hat das Rätsel gelöst: Ja, für einen kurzen Moment sind alle vier Hufe in der Luft.
Muybridge gelang es 1878, alle Phasen des Pferdegalopps erstmals in schnellen Schnappschüssen einzufrieren. Seitdem haben Hochgeschwindigkeitsaufnahmen von Vorgängen, die zu schnell für unser Auge ablaufen, unser Wissen erheblich erweitert. In den Naturwissenschaften ist die Entschlüsselung ultraschneller Prozesse im Reich der Atome und Elementarteilchen sogar essenziell.
Elektronen verhielten sich wie Hütchenspieler
Im Fokus der Garchinger Attosekundenforschung steht das Elektron. „Es kommt praktisch überall vor“, sagt Krausz und weist damit auf die Übermacht dieses negativ geladenen Elementarteilchens hin. Obwohl Elektronen selbst aus der Perspektive der Nanowelt extrem klein sind, kitten sie als Quantenkleber die Atome zur Materie in den vielfältigen Formen unserer Welt zusammen. Elektronen übernehmen überdies die Hauptrollen in chemischen Reaktionen, weil neue Substanzen stets dadurch entstehen, dass sie zwischen verschiedenen Atomen verschoben werden. Allerdings agieren die Elementarteilchen dabei so schnell, dass sich ihr Treiben bis vor Kurzem nicht direkt verfolgen ließ.
Für die Forschung verhielten sich Elektronen also lange wie Hütchenspieler, die Zuschauer mit Schnelligkeit austricksen. Die Quantenmechanik kann ihr Verhalten zwar theoretisch beschreiben, allerdings vor allem die Anfangs- und Endsituation der elektronischen Hütchenspiele. Das galt lange auch für Experimente: Das eigentliche Spiel der Elektronen blieb verborgen. Diese Wissenslücke war nicht nur aus Sicht der Grundlagenforschung unbefriedigend, sie behindert zudem bis heute neue Entwicklungen für die Praxis. Ein Beispiel bilden Katalysatoren, die chemische Reaktionen effizienter ablaufen lassen. Systematisch lässt sich das jeweils beste Material für die chemische Hilfestellung nur identifizieren, wenn Forscher verstehen, wie genau ein Katalysator den Elektronentransfer beeinflusst.
Als in den 1970er-Jahren die Laser immer leistungsfähiger wurden, kam deshalb eine bestechende Idee auf: Vielleicht ließen sich mit extrem kurzen Laserpulsen Schnappschüsse chemischer Reaktionen aufnehmen, so wie einst Muybridge die Phasen des Pferdegalopps ablichtete. „Aus zu verschiedenen Zeiten aufgenommenen Einzelbildern kann man dann einen Film erstellen, der den genauen Ablauf der Reaktion zeigt“, erklärt Krausz.
Mit diesem Ansatz entstand ein neues Forschungsgebiet; es basiert auf einer Methode, die Pump-Probe-Verfahren heißt. Dabei startet ein erster Laserblitz einen Prozess, etwa eine chemische Reaktion, und nach einer bewusst gewählten Wartezeit macht ein zweiter Blitz einen Schnappschuss. Indem die Wissenschaftler die Reaktion wiederholen und die Wartezeit zwischen Pump-und Probe-Laserpuls variieren, erhalten sie die Einzelbilder eines Prozesses. Diese können sie anschließend zu einem Film zusammensetzen.
In den 1990er-Jahren war diese Experimentiertechnik so weit ausgereift, dass sie völlig neue Einblicke in die Natur lieferte. Sie machte erstmals die Bewegung von Atomen in Molekülen sichtbar. Für diese Pionierleistung erhielt der aus Ägypten stammende USamerikanische Physikochemiker Ahmed Zewail 1999 den Nobelpreis für Chemie.
Doch gleichzeitig lief die bis dahin rasante Entwicklung hin zu immer kürzeren Verschlusszeiten für die einzelnen Aufnahmen gegen eine Wand. Die Technik konnte zwar Prozesse in Zeitlupe darstellen, die in wenigen Femtosekunden ablaufen. Damit ließen sich nur die Bewegungen von Atomen filmen, nicht aber jene der viel leichteren und schneller agierenden Elektronen. Um sie in Schnappschüssen einzufrieren, brauchte es eine radikal neue Attosekunden-Blitztechnik.
Um die Jahrtausendwende schaffte es Ferenc Krausz, damals noch an der TU Wien, als Erster, die Mauer in den Attosekundenbereich hinein zu durchbrechen. Seitdem gelingt es immer besser, das Spiel der Elektronen zu filmen. Damit entstand ein neues Forschungsgebiet. Inzwischen nehmen die Garchinger Aufnahmen aus der rasanten Elektronenwelt. Doch warum gab es diese technische Hürde der Ultrakurzzeitforschung?
Femtosekunden-Laserlicht rüttelt die Atome durch
„Das hat mit der Farbe des Lichts, also seiner Wellenlänge, zu tun“, erklärt Krausz. Ein Lichtpuls kann erst dann durch den Raum wandern, wenn er mindestens einen Wellenberg und ein Wellental umfasst. Nur Pulse aus einem solchen vollständigen Wellenzug sind in sich so stabil, dass sie ihren Weg vom Laser zur Probe zurücklegen, ohne völlig zu zerfließen. Sobald ein Wellenzug aber in eine Zeitspanne von einigen Attosekunden gequetscht werden soll, muss seine Wellenlänge kurz genug sein.
„Für Pulse mit einer Dauer unterhalb einer Femtosekunde braucht man schon extrem ultraviolettes Laserlicht“, sagt Krausz, „zur Erzeugung noch kürzerer Pulse führt an Röntgenlicht kein Weg vorbei.“ Und genau das war das Problem: Es gab lange Zeit keine Laser, die Licht in diesem kurzwelligen Spektralbereich produzieren konnten. Erst in den letzten Jahren dringen sogenannte Freie-Elektronen-Laser dorthin vor. Ihr Nachteil: Sie sind auf kilometerlange Teilchenbeschleuniger angewiesen, also riesige, teure Großforschungsanlagen.
In den 1990er-Jahren stellte sich Krausz – wie einst Muybridge – der Herausforderung, diese Hürde mithilfe der vorhandenen Technik zu nehmen. Muybridge kombinierte seine Plattenkamera-Ungetüme damals zu einer langen Batterie, die von einem vorbeigaloppierenden Pferd nacheinander ausgelöst wurden. So gelang es, jede Phase des Galopps auf einem Foto abzulichten.
Krausz musste mit der Femtosekunden-Lasertechnik auskommen, die zu langwellig war. Er kam auf die Idee, die hochintensiven Laserpulse auf Edelgasatome zu schießen. Das Femtosekunden-Laserlicht rüttelt die Atome durch und zwingt sie, einen Blitz im ultravioletten oder im Röntgenbereich auszusenden. Weil die Atome präzise im Takt des anregenden Femtosekunden-Laserpulses schwingen, hat dieses kurzwellige Licht sogar die reine, perfekte Qualität von Laserlicht. Mit diesem Trick drang Krausz erstmals in den Attosekundenbereich vor. Abbildung 3.
|
Abbildung 3. Elektronen auf der Durchreise: Attosekundenblitze von extrem ultraviolettem Licht (violett) katapultieren Elektronen aus den Atomen eines Wolframkristalls (grün). Mit infraroten Laserpulsen (rot) haben die Garchinger Forscher gemessen, dass ein Elektron 40 Attosekunden braucht, um eine einzelne Lage von Magnesiumatomen (blau) über dem Wolframkristall zu durchqueren |
Der kürzeste Garchinger Blitz dauert 72 Attosekunden
Die Garchinger haben ihre Attosekundenanlagen inzwischen immer weiter verbessert. 2004 hielt Krausz’ Gruppe noch mit 650 Attosekunden den Weltrekord für den kürzesten Lichtpuls, heute blitzen die besten Experimente rund zehnmal schneller. „Mit Martin Schultzes Team sind wir hier in Garching jetzt bei 72 Attosekunden angelangt“, sagt Krausz, „aber seit 2012 hält Zenghu Changs Gruppe an der University of Central Florida den Weltrekord mit nur 67 Attosekunden.“ Den Max-Planck-Direktor stört es nicht, dass die Garchinger den Rekord verloren haben – im Gegenteil. „Das zeigt, dass unser Forschungsfeld auf wachsendes Interesse stößt“, freut er sich. Schließlich geht es nicht um Rekorde, sondern um immer feinere Werkzeuge, um die schnellsten Bewegungen im Mikrokosmos zu erforschen.
Zu den ersten Untersuchungsobjekten der Attosekunden-Metrologie oder Attosekunden-Chronoskopie, wie die Garchinger ihre Technik nennen, gehörten die Bewegungen der Elektronen in einzelnen Atomen und Molekülen. Seit einigen Jahren sind jedoch weitaus komplexere Elektronenwelten in den Fokus der Forscher gerückt: kristalline Festkörper. In vielen anorganischen Materialien ordnen sich die Atome zum regelmäßigen, räumlichen Gitter eines Kristalls an. Kristalle kommen in der Natur vielfältig vor, so bilden auch die für unzählige Anwendungen nützlichen Metalle und Halbleiter Kristalle.
Mit der neuen Attosekunden-Messtechnik hat das Team von Reinhard Kienberger gerade in Echtzeit erfasst, wie Elektronen durch eine einzelne Lage von Metallatomen flitzen. Diese Passage ist sowohl für die Physik als auch für die Technik von zentraler Bedeutung. Ihre direkte Beobachtung kann helfen, eines Tages wesentlich schnellere elektronische Schaltelemente und Mikroprozessoren zu entwickeln.
Grundlagenforscher interessieren sich für das komplexe Verhalten der Elektronen in Kristallen, weil die Elementarteilchen darin zwei ganz zentrale Wirkungen haben, die zusammen viele Materieeigenschaften bedingen. So bindet ein Teil der Elektronen die Atome zum Kristallgitter zusammen. Sie formen gewissermaßen den Quantenkitt der Materie. Für die zweite Wirkung sorgen Elektronen, die sich von ihren ursprünglichen Atomen lösen können. Sie flitzen etwa in Metallen einigermaßen frei durchs Kristallgitter und können so elektrischen Strom transportieren. Diese Leitungselektronen haben zudem großen Einfluss auf die mechanischen und optischen Eigenschaften eines Materials und seine Fähigkeit, Wärme zu leiten.
In vielen Kristallen gibt es aber keine Leitungselektronen. Diese Materialien heißen deshalb Isolatoren, ein Beispiel ist Quarz. Zwitter zwischen Isolatoren und elektrischen Leitern stellen die Halbleiter dar. Halbleiter haben als Baustoff der Elektronik unsere Kultur radikal verändert. In ihnen brauchen die Elektronen einen kleinen Schubs, damit sie als Leitungselektronen fließen können. Um dieses Verhalten zu verstehen, muss man ein Elektron als Quantenteilchen betrachten.
Da ein Elektron auch eine elektromagnetische Welle darstellt, besitzt es eine Wellenlänge. Diese ist mit seiner Bewegungsenergie verknüpft – ein bisschen wie das Geräusch eines Rennwagens mit seiner Motordrehzahl. Wenn ein Elektron sich frei durch ein Kristallgitter bewegen will, muss seine Wellenlänge zum räumlichen Raster der Atome passen. Das trifft nur für einen bestimmten Bereich von Wellenlängen – und damit Energie – zu. Dieser Bereich bildet eine Art Autobahn, auf der die Elektronen durch den Kristall rasen können. In diesem Leitungsband fließt bei Metallen immer reger Elektronenverkehr. Bei Halbleitern dagegen hängen auch die beweglichsten Elektronen an ihren Atomen fest. Erst mit einem Energiekick schaffen sie den Quantensprung ins Leitungsband. Das wird zum Beispiel bei Schaltvorgängen in Transistoren genutzt.
Macht starkes Licht einen Quarzkristall leitfähig?
Bei Isolatoren allerdings müsste dieser Energiekick so heftig sein, dass er das Material zerreißen würde. Könnte man trotzdem einen Isolator dazu bringen, leitfähig zu sein – und sei es nur für kurze Zeit? Und: Wäre das auch technisch interessant?
Das fragte sich ein Garchinger Attosekundenteam, in dem Elisabeth Bothschafter und Martin Schultze ihre Doktorarbeiten machten. Um eine Antwort zu finden, brauchten die Laserphysiker versierte Experten für Kristalle an ihrer Seite. Diese Festkörperphysiker fanden sie in der Gruppe des Theoretikers Mark Stockman von der Georgia State University in Atlanta, USA. Für den engen Kontakt sorgte der Amerikaner Augustin Schiffrin, der gerade nach Garching gewechselt war, um dort ebenfalls Attosekunden-Experimente zu machen.
Das Forscherteam untersuchte in den Garchinger Labors an Quarz, ob dieser Isolator unter dem Beschuss mit extrem starken Femtosekunden-Lichtblitzen kurzzeitig leitfähig werden kann – ohne zerstört zu werden. Auch der Einfluss der Lichtblitze auf dessen optische Eigenschaften interessierte die Forscher. Diese momentane Veränderung verfolgten sie mit noch kürzeren Attosekundenblitzen. Bothschafter und Schultze entwickelten dafür ein Experiment, für das die Garchinger ihren hausinternen Rekord auf nur 72 Attosekunden Pulsdauer herunterschraubten. Diese Attosekundenblitze schickten die Forscher synchron zu den Femtosekunden-Lichtpulsen auf ihre Probe. Damit verfügten sie über das passende Präzisionswerkzeug, um die genaue Form der verwendeten Femtosekunden-Laserpulse genau abzutasten. Das war entscheidend, um die durch diese Pulse im Quarz verursachte Veränderung richtig zu deuten.
Quarz überlebt das kurze elektrische Inferno
Zudem analysierten die Physiker mit den Attoblitzen in Echtzeit, was in der nur knapp 200 Nanometer dünnen Quarzprobe unter dem Lichtbeschuss im Detail passierte. Dort erzeugte der intensive Femtosekunden-Laserpuls, bei dem es sich um eine elektromagnetische Welle handelt, ein extrem starkes elektrisches Feld. Martin Schultze vergleicht es mit dem Feld einer Überland-Hochspannungsleitung, das zwischen zwei Elektroden anliegt, die nur wenige tausendstel Millimeter voneinander entfernt sind. Jedes bekannte Material würde dabei sofort verdampfen.
|
Abbildung 4. Licht erzeugt Strom: Mit einem sehr intensiven dunkelroten Laserblitz machen Max-Planck-Physiker ein Quarzprisma, das sie auf zwei Seiten mit Goldelektroden bedampft haben, vorübergehend leitfähig. Mit einem zweiten, wesentlich schwächeren Puls schieben sie die kurzzeitig mobilen Elektronen zu einer Elektrode – Strom fließt. |
Der Quarz überlebte dieses elektrische Inferno allein deshalb, weil es in ihm nur für wenige Femtosekunden tobte. Er war schlicht zu träge, um es zu bemerken. Bevor seine Atome auseinanderdriften konnten, war es längst vorbei. Allerdings sah es bei den Elektronen anders aus. Einige von ihnen folgten dem Feld des Femtosekundenpulses wie Hunde an der Leine. Sie erzeugten somit vorübergehend einen elektrischen Strom im Quarz, der am Ende des Femtosekundenblitzes sofort wieder abklang. Danach war der Quarz wieder ein normaler Isolator. Abbildung 4.
„Die Leitfähigkeit lässt sich also mit dem Lichtpuls in der unvorstellbar kurzen Zeitspanne von wenigen Femtosekunden nicht nur an-, sondern auch wieder ausschalten“, sagt Krausz. Letzteres war entscheidend, denn das passiert in Halbleitern nicht. Abbildung 5.
Stockmans Gruppe zeigte, dass die Elektronen sich dabei nach einem völlig anderen Mechanismus bewegen als in Halbleitern. Dort lassen sie sich zwar auch innerhalb von Femtosekunden ins Leitungsband kicken. Aber dann verharren sie darin aus Sicht der Attosekundenphysik eine Ewigkeit, nämlich ungefähr tausend- bis zehntausendmal länger als eine Femtosekunde. Im Quarz jedoch folgen die Elektronen unmittelbar dem elektrischen Lichtfeld von ihren Atomen weg und springen ebenso rasch wieder zurück, sobald das Feld abklingt. Die Forscher erhielten auf diese Weise also einen elektrischen Schalter, der extrem schnell wirkt, nämlich innerhalb von Femtosekunden.
|
Abbildung 5. Ein nur wenige Nanometer dickes Quarzglas, das in einen schwarzen Rahmen eingespannt ist (Bildmitte), wird durch einen roten Laserpuls leitfähig. Mit einem anschließenden Attosekundenpuls lässt sich messen, wie schnell das Quarzglas wieder zum Isolator wird. |
Die Garchinger Physiker und ihre Kooperationspartner widmen sich diesen Vorgängen zunächst als Grundlagenforscher, und sie haben dabei etwas völlig Neues aufgespürt: So einen exotischen Zustand hat zuvor noch niemand in einem Isolator erzeugt. Langfristig könnte diese Entdeckung aber auch die Elektronik revolutionieren. Das relativ träge Verharren der Elektronen im Leitungsband von Halbleitern begrenzt nämlich die Geschwindigkeit, mit der herkömmliche Transistoren schalten können. Im Labor kommen konventionelle Transistoren mit extrem kleinen Nanostrukturen zwar schon auf hundert Milliarden Schaltungen pro Sekunde. Das ist grob eine Größenordnung schneller als die heute etablierten Computer-Mikroprozessoren.
„Mit unserer Entdeckung wären aber nochmals um den Faktor zehntausend schnellere Schaltzeiten möglich“, bilanziert Krausz. Damit könnten Computer ungeheure Datenmengen in Echtzeit verarbeiten. Allerdings müsste es dazu auch gelingen, die metergroßen Kurzpulslaser, die den Schaltvorgang antreiben, zu miniaturisieren. „Deshalb – und auch aus einigen anderen Gründen – ist das derzeit erst einmal eine spannende Zukunftsperspektive“, betont Krausz.
Für ihn als Max-Planck-Wissenschaftler steht der Erkenntnisgewinn im Fokus. Mit der neuen Attosekunden-Messtechnik hat das Team von Reinhard Kienberger gerade in Echtzeit erfasst, wie Elektronen durch einzelne Atomlagen eines Kristallgitters flitzen. Diese „Reise“ ist für Physik und Technik von zentraler Bedeutung. Die direkte Beobachtung kann entscheidend helfen, eines Tages wesentlich schnellere elektronische Schaltelemente und Mikroprozessoren zu entwickeln.
[1] Hentschel, M., Kienberger, R., Spielmann, C. et al. Attosecond metrology. Nature 414, 509–513 (2001). https://doi.org/10.1038/35107000
[2] Roland Wengenmayr, 09.03.2023: Technologie aus dem Quantenland mit unzähligen Anwendungsmöglichkeiten.
* Der Artikel ist erstmals unter dem Title: "Klappe für den Quantenfilm"https://www.mpg.de/9221304/F002_Fokus_024-031.pdf im Forschungsmagazin der Max-Planck-Gesellschaft 1/15 erschienen und wurde mit Ausnahme des Titels und Abstracts unverändert in den Blog übernommen. Die MPG-Pressestelle hat freundlicherweise der Veröffentlichung von Artikeln aus dem Forschungsmagazin auf unserer Seite zugestimmt. © Max-Planck-Gesellschaft -
Weiterführende Links
The Nobel Prize in Physics 2023: Popular information. https://www.nobelprize.org/prizes/physics/2023/popular-information/
The Nobel Prize in Physics 2023: Advanced information. https://www.nobelprize.org/prizes/physics/2023/advanced-information/
MPG: Laser - Der schnellste Blitz der Welt. Video 8:30 min. (2012) https://www.youtube.com/watch?v=6zxzJqvzZMY
MPG: Wie kurz ist eine Attosekunde? Video 2:49 min. https://www.youtube.com/watch?v=FR21qcg856g . copyright www.mpg.de/2012
FU Berlin: 21st Einstein Lecture: Prof. Dr. Ferenc Krausz | Elektronen und Lichtwellen - gemeinsam gegen Krebs (2.12.2022) Video: 1:31:41. https://www.youtube.com/watch?v=J-ynY3YNpkM
Ferenc Krausz, Nobelpreis für Physik 2023. Pressekonferenz 03.10.2023: Video 24:06 min. https://www.youtube.com/watch?v=9FZvFnurOnA
Comments
MPI of Quantum Optics: Ferenc Krausz - Attosecond Physics
https://www.youtube.com/watch?v=6ZIUJb85BAQ
- Log in to post comments
Seegraswiesen wandeln Kohlendioxid in Zuckerverbindungen um und sondern diese in den Meeresboden ab
Seegraswiesen wandeln Kohlendioxid in Zuckerverbindungen um und sondern diese in den Meeresboden abDo. 28.09.2023 — Manuel Liebeke

![]() Auf alten Karten findet sich häufig die Bezeichnung „terra incognita“ – unbekanntes Land. Bis heute sind große Teile der Weltmeere unerforscht, „mare incognitum“ sozusagen. In den Meeren gibt es also immer noch jede Menge Neues zu entdecken. Prof. Dr. Manuel Liebeke, Gruppenleiter am Max-Planck-Institut für Marine Mikrobiologie, und sein Team erforschen Seegraswiesen. Obwohl diese Ökosysteme nur eine kleine Fläche des Ozeans bedecken, binden sie durch Photosynthese große Mengen atmosphärischen Kohlendioxids und sondern Berge von Zuckerverbindungen in den Meeresboden ab.*
Auf alten Karten findet sich häufig die Bezeichnung „terra incognita“ – unbekanntes Land. Bis heute sind große Teile der Weltmeere unerforscht, „mare incognitum“ sozusagen. In den Meeren gibt es also immer noch jede Menge Neues zu entdecken. Prof. Dr. Manuel Liebeke, Gruppenleiter am Max-Planck-Institut für Marine Mikrobiologie, und sein Team erforschen Seegraswiesen. Obwohl diese Ökosysteme nur eine kleine Fläche des Ozeans bedecken, binden sie durch Photosynthese große Mengen atmosphärischen Kohlendioxids und sondern Berge von Zuckerverbindungen in den Meeresboden ab.*
Seegräser wachsen an den meisten Küsten der Weltmeere. Ähnlich wie Landpflanzen binden die Gräser durch Photosynthese Kohlendioxid und wandeln es in neue, größere Kohlenstoffverbindungen um. Obwohl sie nur 0,2 Prozent der Meeresfläche bedecken, produzieren Seegräser etwa zehn Prozent der in den Ozeanen versinkenden Kohlenstoffverbindungen. Sie leisten somit einen erheblichen Beitrag für den Kohlenstoffhaushalt der Meere und des gesamten Planeten.
Seegräser geben einen Teil des von ihnen gebundenen Kohlenstoffs meist als Zuckerverbindungen über ihre Wurzeln an den Meeresboden ab. Auch viele Landpflanzen sondern aus ihren Wurzeln Zucker ab. Im Boden lebende Pilze und viele Mikroorganismen erfreuen sich an diesen für sie lebenswichtigen Kohlenhydraten. Manche dieser Mikroben stellen den Pflanzen im Gegenzug Mineralien und andere anorganische Substanzen zur Verfügung. Abbildung 1.
| Abbildung 1. Kohlenstoffkreislauf in Seegras-Sedimenten. Seegräser geben einen Teil des von ihnen gebundenen Kohlenstoffs meist als Zuckerverbindungen über ihre Wurzeln an den Meeresboden ab. Davon verbrauchen die Mikroorganismen in der Rhizosphäre nur wenig- sie werden offensichtlich von - ebenfalls von Seegräsern produzierten - phenolischen Verbindungen (weiße Strukturen) gehemmt. |
Solche schon vor über 100 Jahren als Rhizosphäre bezeichneten Bodenökosysteme sind inzwischen sehr gut erforscht und die Symbiosen zwischen Landpflanzen und den im Boden lebenden Mikroorganismen dokumentiert. Dagegen ist über pflanzliche Zuckerausscheidungen im Meeresboden und deren potenzielle Wechselwirkungen mit Mikroben nur sehr wenig bekannt. Mein Team und ich wollen diese Lücke schließen. In verschiedenen Meeresregionen haben wir Wassersproben im Sediment unter den Seegraswiesen genommen und die darin vorkommenden, von den Pflanzen abgegebenen Stoffwechselprodukte analysiert. Unsere Analysen zeigen, dass unterhalb von Seegraswiesen viel Rohrzucker (Saccharose) vorkommt. Die Konzentrationen erreichen in einigen Sedimenten je nach Tiefe sehr hohe (millimolare) Werte. Der Rohrzucker macht bis zu 40 Prozent des von den Wurzeln der Seegräser abgegebenen organischen Kohlenstoffs aus. Dessen Konzentration im Boden schwankt im Tagesverlauf und je nach Jahreszeit. Bei sehr starkem Lichteinfall, zum Beispiel zur Mittagszeit oder im Sommer, produzieren Seegräser mehr Zucker, als sie verbrauchen oder speichern können. Dann geben sie die überschüssige Saccharose einfach an den Boden ab.
Mikroben im Boden verbrauchen wenig Saccharose
Warum aber lagern Seegräser so viel Saccharose in den Sedimenten ab? Handelt es sich vielleicht um eine Art Überfluss-Stoffwechsel, weil ihnen essenzielle Substrate zum Aufbau anderer Kohlenhydrate fehlen? Die meisten Mikroorganismen an Land und im Meer können Saccharose leicht verdauen und daraus viel Energie für sich gewinnen. Wir haben jedoch bei unseren Untersuchungen festgestellt, dass viele der in der Seegras-Rhizosphäre vorkommenden Mikroorganismen vergleichsweise wenig Saccharose beanspruchen. Dies könnte die von uns beobachtete Anreicherung des Zuckers im Sediment erklären. Oder vielleicht fehlen den Mikroben die Stoffwechselwege, die sie für die Verdauung von Saccharose benötigen?
Möglich ist aber auch, dass die Seegräser Stoffwechselprodukte absondern, die das Wachstum der Mikroorganismen bremsen. Unser Verdacht: Es könnte sich um phenolische Verbindungen handeln. Phenolartige Substanzen kommen in verschiedensten Pflanzenarten vor, zum Beispiel in Weintrauben, Kaffeebohnen und Obst. Diese Stoffe hemmen den Stoffwechsel von Mikroorganismen und wirken dadurch antimikrobiell – vermutlich ihre eigentliche Aufgabe für die Gesundheit der jeweiligen Pflanze.
Seegräser enthalten von Natur aus viele Phenole, etwa Kaffeesäure (3,4-Dihydroxyzimtsäure), die sie an ihre Umgebung abgeben könnten. Im Meerwasser rund um Seegraswiesen konnten wir dann phenolische Verbindungen nachweisen, die aus pflanzlichen Polymeren gebildet werden. Um zu prüfen, ob die in der Umgebung von Seegräsern vorkommenden Mikroorganismen von diesen Phenolen gehemmt werden, haben wir auf der Mittelmeerinsel Elba Bodenproben von unterhalb der Seegraswiesen genommen und mit aus Seegras isolierten Phenolen sowie Saccharose in Kontakt gebracht. Unsere Messungen zeigen, dass die Mikroben in den Bodenproben nach Zugabe der phenolischen Substanzen viel weniger Saccharose konsumiert haben, dies war besonders deutlich bei Versuchen ohne Sauerstoff. Der Zuckergehalt blieb also entsprechend hoch, ähnlich unseren Beobachtungen direkt aus der Seegraswiese.
| Abbildung 2. Üppige Seegraswiesen von Posidonia oceanica im Mittelmeer. Das Team des Max-Planck-Instituts für marine Mikrobiologie geht davon aus, dass seine Erkenntnisse auch für andere Seegrasarten sowie Mangroven und Salzwiesen relevant sind. |
Als Nächstes haben wir die im Sediment vorhandenen Nukleinsäuren isoliert und sequenziert. Auf diese Weise konnten wir alle Arten von Mikroorganismen anhand ihres Erbguts identifizieren und klassifizieren, welche Stoffwechselgene sie besitzen. Trotz der toxischen Phenole scheint eine kleine Gruppe mikrobieller Spezialisten sowohl Saccharose verdauen als auch Phenole abbauen zu können. Wir vermuten, dass diese von uns entdeckten Mikroorganismen nicht nur auf den Abbau von Zucker und Phenolen spezialisiert sind. Wahrscheinlich sind sie für das Seegras auch nützlich, weil sie Nährstoffe produzieren, welche die Pflanzen für ihr Wachstum dringend brauchen. Der Kohlenstoffkreislauf in Seegras-Sedimenten unterscheidet sich somit von dem auf dem Land und auch von den Kohlenstoffkreisläufen im freien Meerwasser, wo Mikroben Saccharose schnell abbauen.
Phenole in anderen Ökosystemen
Phenolverbindungen kommen höchstwahrscheinlich auch in anderen Ökosystemen in größeren Mengen vor. Wir untersuchen zurzeit die Zusammensetzung von gelöstem Kohlenstoff in Salzmarschen und Mangrovenwäldern. Abbildung 2. Innerhalb der Deutschen Allianz Meeresforschung beteiligt sich unser Institut an Forschungsvorhaben, welche die deutschen Küstenregionen in den Fokus rücken. Vergleichsstudien und Probenahmen in tropischen Ländern ergänzen das Projekt. Zudem wird untersucht, wie stabil Kohlenwasserstoffe bei starkem UV-Licht oder erhöhten Temperaturen sind. Diese Umweltfaktoren sind für das weitere Schicksal des Kohlenstoffs im großen Meereskreislauf entscheidend.
Das Ziel ist, Küstenökosysteme zu erhalten, die langfristig möglichst viele stabile Kohlenstoffverbindungen produzieren und so den Anstieg des Gehalts an Kohlendioxid in der Atmosphäre in Schach halten. Auch eine „Wiederaufforstung“ zerstörter Flächen mit Seegras wäre denkbar, wenn es die Beschaffenheit der Küsten zulässt.
*Der unter Highlights aus dem Jahrbuch 2022 der Max-Planck Gesellschaft unter dem Titel " Süße Oasen im Meer" erschienene Artikel https://www.mpg.de/20478773/jahrbuch-highlights-2022.pdf (2023) wird - mit Ausnahme des Titels und der Legende zu Abbildung 1- in praktisch unveränderter Form im ScienceBlog wiedergegeben. Die MPG-Pressestelle hat freundlicherweise der Veröffentlichung von Artikeln aus dem Jahrbuch auf unserer Seite zugestimmt. (© 2023, Max-Planck-Gesellschaft)
Weiterführende Links
Max-Planck-Institut für Marine Mikrobiologie: Miracle plant seagrass (2022). Video 2:07 min. https://www.youtube.com/watch?v=iWm2lkK062Q.
The Biome Project: Seagrasses - Ecology In Action (2022). Video 6:59 min. https://www.youtube.com/watch?v=0bvOh7qby-c
International Field Studies: Seagrass Ecosystem Webinar (2020). Video 22:00 min. https://www.youtube.com/watch?v=3j82byN7HGs
Artikel zu verwandten Themen im ScienceBlog:
- Redaktion, 17.06.2023: Mikroorganismen spielen eine entscheidende Rolle bei der Kohlenstoffspeicherung in Böden
- Knut Ehlers, 01.04.2016: Der Boden - ein unsichtbares Ökosystem
- Rattan Lal, 27.11.2015: Boden – Der große Kohlenstoffspeicher
- Rattan Lal, 11.12.2015: Der Boden – die Lösung globaler Probleme liegt unter unseren Füßen
- Redaktion, 26.06.2015: Die Erde ist ein großes chemisches Laboratorium – wie Gustav Tschermak vor 150 Jahren den Kohlenstoffkreislauf beschrieb
Coenzym Q10 - zur essentiellen Rolle im Zellstoffwechsel und was Supplementierung bewirken kann
Coenzym Q10 - zur essentiellen Rolle im Zellstoffwechsel und was Supplementierung bewirken kannSo24.09.2023 — Inge Schuster
Coenzym Q10 (CoQ10) ist eine unentbehrliche Komponente unseres Zellstoffwechsels; es ist in allen unseren Körperzellen vorhanden (vom ubiquitären Vorkommen leitet sich die Bezeichnung Ubichinon ab) und wird - mit Ausnahme der roten Blutkörperchen - auch in allen Zellen produziert. CoQ10 fungiert als Elektronen-und Protonenüberträger in der mitochondrialen Atmungskette und schützt als potentes Antioxidans vor reaktiven Sauerstoffspezies. CoQ10-Mangel kann zur Entstehung diverser Krankheiten beitragen bzw. diese auslösen. In zahlreichen Studien wurde und wird versucht den Mangel durch Supplementierung mit CoQ10 aufzuheben, um Krankheiten zu lindern/zu heilen. Einige Studien lassen berechtigte Hoffnung auf Erfolg aufkommen.
Was ist CoQ10?
Chemisch gesehen ist CoQ10 ein relativ großes, aus zwei unterschiedlichen Teilen zusammengesetztes organisches Molekül. Es besteht aus dem Redoxsystem des Chinon-/Chinol -Rings, das die funktionelle Gruppe des Coenzyms darstellt und aus einer langen enorm lipophilen (fettlöslichen) Seitenkette aus Isoprenresten - beim Menschen sind das hauptsächlich 10 Reste -, mit der sich das Molekül in die diversen Lipidmembranen der Zelle und in die Lipoproteine des Serums einfügt. CoQ10 findet sich vor allem in der Matrix der Mitochondrien, aber auch in den Membranen des Golgi-Apparats, des endoplasmischen Retikulums, der Lysosomen, der Peroxisomen und in der die Zelle umschliessenden Plasmamembran.
CoQ10 gehört zu den lipophilsten Strukturen in unserem Organismus - in wässrigem Milieu ist es praktisch unlöslich (gelöster Anteil weniger als 0,1 Milliardstel Substanz). Abbildung 1.
| Abbildung 1. Coenzym Q10 - chemische Struktur des Redoxsystems. Das enorm lipophile Molekül liegt im menschlichen Organismus zu über 90 % mit einer aus 10 Isoprenresten bestehenden Seitenkette vor, unter 10 % weisen eine Seitenkette aus 9 Isoprenresten auf. Das Ring-Strukturelement (rot) ist die aktive funktionelle Gruppe des Coenzyms, ein Redoxsystem, das unter Aufnahme von 2 Elektronen und 2 Protonen zum Chinol reduziert wird, aus welchem unter Abgabe von Elektronen und Wasserstoff wieder das oxydierte Produkt - Ubichinon-10 - entsteht. |
Biosynthese von CoQ10
Alle unsere Zellen sind auf die endogene Synthese von CoQ10 angewiesen. Die mit der Nahrung aufgenommenen Mengen (üblicherweise bis zu 6 mg im Tag und dies hauptsächlich über den Fleischkonsum) können den hohen permanenten Bedarf an CoQ10 nicht decken.
Die Biosynthese erfolgt in 4 Stufen, an denen mindestens 11 Enzyme und andere Proteine beteiligt sind. Zuerst werden die Vorstufe des Ringelements und die Isopren-Seitenkette separat gebildet, der Ring ausgehend von der Aminosäure Tyrosin, die Lipidkette über den Mevalonsäure-Weg (auf dem auch die Synthese von Cholesterin ihren Ausgang nimmt) und die sukzessive Addierung von Isoprenresten. Dann werden Ring und Seitenkette zusammengefügt und schließlich der Ring schrittweise zur Ubichinon-Form modifiziert.
Mit Ausnahme einiger Reaktionen zu Beginn der Biosynthese findet der gesamte Vorgang in der inneren Membran der Mitochondrien statt, wobei die daran beteiligten Proteine einen großen Komplex - das sogenannte CoQ-Synthom - bilden. Abbildung 2. Das neu entstandene CoQ10 lagert sich in der mitochondrialen Membran ein - ein Pool, der dort nicht nur für die essentielle Funktion in der Atmungskette (siehe unten) zur Verfügung steht; CoQ10 wird aber auch in andere Zellorganellen transportiert und zirkuliert in der Blutbahn.
Biologische Funktionen von CoQ10
Lipidlöslichkeit und Redox-Eigenschaften erklären die vielfältigen (pleiotropen) Funktionen von CoQ10 in biologischen Systemen. Wie bereits erwähnt lagert sich CoQ10 in allen Lipidmembranen der Zellen ein und kann dort als mobiler Elektronen-und Protonenüberträger (siehe Abbildung 1) und als potentes Antioxidans fungieren. Als wichtigstes Beispiel für den Elektronen-/Protonentransport soll die Schlüsselrolle von CoQ10 in der mitochondrialen Atmungskette kurz skizziert werden.
Atmungskette: wie Nahrung in Energie umgewandelt wird
Mitochondrien sind die Kraftwerke unserer Zellen, die bis zu 95 % der für Zellwachstum und Zelldifferenzierung nötigen Energie produzieren. In diesem Prozess der Energieerzeugung ist CoQ10 eine essentielle Komponente: Es fungiert im Elektronentransport-System der Atmungskette als mobiler Elektronen- und Protonenüberträger zwischen den membrangebundenen Proteinkomplexen I , II und III, die schlussendlich zur Bildung des universellen biologischen Energieträgers Adenosintriphosphat (ATP) führen. Abbildung 2 zeigt eine sehr vereinfachte Darstellung dieses Elektronentransportsystems (über die folgende Beschreibung des für Laien nicht leicht verständlichen Vorgangs kann auch hinweg gelesen werden):
Coenzym Q10 in der oxydierten Chinonform (siehe Abbildung 1) nimmt in den Proteinkomplexen I und II Reduktionsäquivalente (Elektronen) auf und wird dadurch zur Chinolform reduziert; die Elektronen stammen dabei aus dem ebenfalls in den Mitochondrien lokalisierten Citratzyklus, der Produkte der im Stoffwechsel abgebauten Nährstoffe in Reduktionsmittel umwandelt; Elektronen können aber auch durch Überträger aus anderen mitochondrialen Prozessen eingespeist werden. Die reduzierte Ubichinol-Form überträgt im Proteinkomplex III (Cytochrom C-Reduktase) die Elektronen auf Cytochrom C, die Protonen werden vom mitochondrialen Matrixraum in den Intermembranraum transloziert. Im Komplex IV (Cytochrom C-Oxidase) dienen die vom Cytochrom C transportierten Elektronen und Protonen zur Reduzierung des aus der Atmung stammenden Sauerstoffs unter Bildung von Wasser. Dabei werden weitere Protonen in den Intermembranraum gepumpt und ein elektrochemischer Gradient zwischen Intermembranraum und Matrix erzeugt, der schließlich den Aufbau des energiereichen ATP-Moleküls (via ATP-Synthase) treibt.
| Abbildung 2. CoQ10 wird in den Mitochondrien produziert und spielt dort eine Schlüsselrolle in der Energieerzeugung über die Atmungskette. Oben: Vereinfachte Darstellung des Biosynthese-Komplexes - CoQ-Synthom - mit Enzymen (blau) und regulatorischen Proteinen (lila), wobei nur die Ziffern in den Namen der COQ-Proteine (z.B. 2 für COQ2) angezeigt sind. Unten: Schema des Elektronentransportsystems der Atmungskette mit CoQ10 (gelb), das Elektronen von Komplex I und II empfängt und auf Komplex III überträgt. Die weitere Übertragung auf Komplex IV resultiert in der Reduktion des aus der Atmung stammenden Sauerstoffs unter Bildung von Wasser. Der Protonengradient treibt die ATP-Synthese.(Bild leicht modifiziert aus: Y. Wang & S.Hekimi, J Cell Mol Med. 2022;26:4635–4644. Lizenz: cc-by.) |
Ubichinol - als potentes Antioxidans
In seiner reduzierten Ubichinol-Form ist CoQ10 ein sehr effizientes Antioxidans, das vor der Entstehung von reaktiven Sauerstoffspezies (ROS) schützt, freie Radikale verhindert, die Peroxidation von Lipiden und Lipoproteinen blockiert und andere Antioxidantien wie Ascorbinsäure oder Vitamin E zu ihren aktiven, völlig reduzierten Formen regeneriert.
Der Schutz vor ROS ist insbesondere in den Mitochondrien von Bedeutung, da der intensive, permanente Energieerzeugungsprozess als Nebenprodukte ROS liefert: Aus dem Sauerstoff den wir einatmen und der zu 80 % in der Atmungskette zu Wasser reduziert wird, entstehen auch 1 - 2 % reaktive Sauerstoffspezies (u.a. Superoxid Anion, Hydroxyl-Radikal, Hydroperoxyl-Radikal, Singlet Sauerstoff). Diese ROS können diverse Biomoleküle (Proteine, Lipide, DNA) gravierend schädigen - darunter die Proteine der CoQ10-Biosynthese und die mitochondriale DNA, die für einige Enzyme der Atmungskette kodiert. ROS können aber auch die Proteinkomplexe der Atmungskette direkt angreifen und (teilweise) inaktivieren. Insgesamt resultiert ein CoQ10-Mangel und Energieerzeugung und auch Verfügbarkeit von Ubichinol zur Abwehr der ROS werden reduziert.
Zahlreiche Studien belegen, dass oxydative Schädigungen zur Beeinträchtigung der mitochondrialen Funktion und in Folge zur Entstehung von diversen Krankheiten beitragen bzw.diese auslösen können. Ausreichend reduziertes CoQ10 in den Mitochondrien kann diese ROS unschädlich machen.
CoQ10 in unseren Organen....
CoQ10 ist in allen menschlichen Zellen und damit in allen Organen vorhanden, wobei der Großteil des Koenzyms in den jeweiligen Mitochondrien sitzt. Wieviele dieser kleinen Organellen in einzelnen Zelltypen enthalten sind, hängt von deren Energiebedarf ab und kann sich an diesen anpassen. Besonders viele Mitochondrien - mehrere Tausend - sind in einer einzelnen Herzmuskelzelle tätig - zusammengenommen nehmen sie mehr als ein Drittel des Zellvolumens ein. Dementsprechend liegen in diesem Organ , aber auch in anderen Organen mit sehr hohem Energiebedarf - Leber, Nieren und Muskeln - die höchsten CoQ10-Konzentrationen (Ubichinon und Ubichinol in Summe bis zu 190 µg/g) vor. Abbildung 3. Ein hoher Anteil an reduziertem CoQ10 schützt in vielen Organen vor dem unvermeidlichen Entstehen von reaktivem Sauerstoff.
Die Gesamtmenge von CoQ10 in unserem Körper kann mit einigen Gramm geschätzt werden.
| Abbildung 3. Organspiegel von CoQ10 in oxydierter und reduzierter Form in verschiedenen Organen des Menschen. Die Grafik wurde aus Daten von A.Martelli et al., Antioxidants 2020, 9, 341; doi:10.3390/antiox9040341 (Lizenz cc-by) zusammengestellt.. |
Im Vergleich zu den Organen liegt der Plasmaspiegel von CoQ10 mit 0,5 - 1,5 µg/ml wesentlich niedriger, wobei nahezu alles in der reduzierten Ubichinolform vorliegt.
Ubichinol-10 zirkuliert ausschließlich in Lipoproteinen, vor allem in LDL eingelagert und kann hier vor der Oxidation von Lipoproteinen aber auch von Cholesterin zu Oxysterolen schützen und damit das Atherosklerose-Risiko senken.
Da Lipoproteinspiegel stark variieren können, erscheint es problematisch aus dem Plasmaspiegel von CoQ10 auf Organspiegel und eventuellen Mangel zu schließen.
...und CoQ10-Mangel
Eine reduzierte Synthese von CoQ10 führt nicht nur zu verminderter Energieproduktion und in Folge zur Schwächung von Organfunktionen, sondern auch zu reduziertem Schutz vor reaktiven Sauerstoffspezies und anderen Radikalen. Davon sind gewöhnlich mitochondrienreiche Organe mit hohem Energiebedarf betroffen, insbesondere die ausdifferenzierten (d.i. sich nicht mehr teilenden) Herzmuskelzellen und Neuronen, da sie bei geschädigter mitochondrialer Funktion nicht mehr ersetzt werden und mit zunehmendem Alter mehr und mehr Schäden ansammeln (s. unten).
Ein primärer CoQ10-Mangel - eine genetische, selten auftretende, autosomal rezessive Erkrankung-- tritt auf, wenn Gene der in der Biosynthese von CoQ10 involvierten Proteine von Mutationen betroffen sind, die zu reduzierten Aktivitäten und damit verringertem CoQ10-Level führen. Klinische Symptome manifestieren sich schon früh und betreffen viele Organe, vor allem das zentrale und periphere Nervensystem, Herz, Muskel und Niere.
Ein sekundärer CoQ10-Mangel ist häufiger und kann verschiedene Ursachen haben, u.a. defekte Varianten von anderen, nicht in der CoQ10-Biosynthese involvierten Genen, beispielsweise im mitochondrialen Transport oder im Citratzyklus.
Auch zunehmendes Alter führt in den meisten Organen zu einem Absinken der CoQ10-Konzentration. Davon ist vor allem das Herz betroffen: 80-Jährige haben im Vergleich zu 20-Jährigen nur noch etwa 50 Prozent des CoQ10-Gehaltes. Ebenso sinkt der CoQ10-Gehalt in verschiedenen Hirnregionen und in der Epidermis der Haut deutlich ab.
In vielen Erkrankungen - Herzkrankheiten (z.B. Herzinsuffizienz), neurologischen Erkrankungen (beispielsweise Parkinson Krankheit), Diabetes, Lungen-und Lebererkrankungen liegen deutlich reduzierte CoQ10-Konzentrationen in den Organen vor. CoQ10 Mangel kann hier Auslöser und/oder auch Folge der Krankheit sein. In Infektionskrankheiten wie Influenza kommt es zu reduzierten CoQ10-Plasmaspiegeln.
Einige häufig verschriebene Medikamenten greifen direkt in die Biosynthese von CoQ10 ein, indem sie gemeinsame Schritte blockieren: das bekannteste Beispiel sind die millionenfach zur Cholesterinsenkung geschluckten Statine, die einen ganz frühen gemeinsamen Schritt (via HMG-CoA Reduktase) in der Cholesterinsynthese und in der Synthese der CoQ10-Seitenkette blockieren. Unter Therapie mit Statinen wurden deutlich (um ein Drittel) reduzierte CoQ10-Konzentrationen im Muskelgewebe gemessen. Eine Reihe von Statin-Nebenwirkungen in den Muskeln, die von Muskelschwäche, Myalgien bis hin zur seltenen, lebensbedrohenden Rhabdomyolyse reichen, dürften Folge des CoQ10-Mangel sein. Auch im Plasma und im zentralen Nervensystem (gemessen am Indikator Zerebrospinalflüssigkeit) ist der CoQ10-Gehalt unterStatin-Therapie erniedrigt. Zudem wird das Auftreten von kognitiver Beeinträchtigung und zerebellärer Ataxie mit Statin-bedingetm CoQ10-Mangel assoziiert.
Eine weitere Gruppe von Medikamenten - die gegen Osteoporose angewandten Biphosphonate - inhibieren eine Reaktion, die in einem späten Schritt auch zur Synthese der CoQ10- Seitenkette führt.
Supplementierung von CoQ10
Der globale Markt von CoQ10 ist groß (derzeit rund 700 Mio $) und soll laut Prognosen jährlich um rund 7 % wachsen. Allerdings gibt es bislang kein von FDA oder EMA registriertes CoQ10- Arzneimittel, dagegen eine riesige Menge von frei erhältlichen Präparaten - vom Nahrungsergänzungsmittel bis hin zu diversen Kosmetikprodukten, welche die Regale von Apotheken, Drogerien, Beautyshops und Supermärkten füllen. Da es keine Vorschriften bezüglich Formulierung und Dosierung gibt, sind Qualität und angepriesene Wirksamkeit der Präparate in vielen Fällen zu hinterfragen. Dies ist insbesondere der Fall, da das überaus lipophile CoQ10 in ungeeigneten Formulierungen auskristallisiert (Schmelzpunkt 48oC) und dann nur in sehr geringem Ausmaß aus dem Darm in den Organismus aufgenommen werden kann; damit bleibt es unwirksam. Ein wesentliches Manko ist auch, dass für Wirksamkeitsstudien eine solide finanzielle Basis fehlt, um diese genügend lang, an einer ausreichend großen Zahl von Probanden als klinische Doppelblind-Studien durchzuführen.
Nichtsdestoweniger gibt es viele klinische Studien, in denen CoQ10 supplementiert wurde: ua. bei Herz-Kreislauferkrankungen, neurologischen Defekten, Stoffwechselerkrankungen, Statin-verursachten Nebenwirkungen und als Antiaging-Mittel. Die US-Datenbank ClinicalTrials.gov weist 82 Einträge solcher z.T. noch laufender Studien auf. In den letzten 10 Jahren sind 420 Originalberichte und Metaanalysen allein über klinische Studien erschienen - die Ergebnisse sind leider durchwachsen. In vielen Fällen dürften zu kleine Teilnehmerzahl, zu kurze Versuchsdauer und vor allem unzureichende Resorption des Wirkstoffs die Aussagekraft der Studie geschmälert haben.
Ist CoQ10 nun ein Wundermittel, das Krankheiten vorbeugt und heilt und uns Jugend verspricht?
| Tabelle. Positive Ergebnisse bei Herzinsuffizienz. (Daten aus Mortensen et al., 2014; https://doi.org/10.1016/j.jchf.2014.06.008 Lizenz cc-by)) |
Im Folgenden sollen zwei Studien angeführt werden, die - mit optimalen CoQ10 Formulierungen ausgeführt - positive Ergebnisse erzielten und Hoffnung auf erfolgreiche Supplementierung bei weiteren Indikationen erwecken.
Die SYNBIO-Studie: Anwendung bei Herzsuffizienz
Die 2014 publizierte Studie hat insgesamt 420 Patienten mit Herzinsuffizienz Stufe III und IV 2 Jahre lang 3 x täglich mit jeweils 100 mg CoQ10 in einer speziell entwickelten, auf Resorbierbarkeit geprüften Formulierung oder mit Placebo behandelt. Nach 2 Jahren führte die Behandlung zu fast 3-fach erhöhten CoQ10-Plasmaspiegeln und zu einer Reduktion des Krankheits-spezifischen Markers NT-proBNP um 256 pg/ml. Die Sterblichkeit an Herzerkrankungen lag in der CoQ10 Gruppe um über 50 % niedriger als in der Placebogruppe und ebenso die Einweisungen ins Krankenhaus wegen Verschlechterung des Zustands. Tabelle.
Topische Anwendung von CoQ10 auf der Haut
| Abbildung 4. In geeigneten Formulierungen angewandt wird die CoQ10 Konzentration in der Epidermis bedeutend erhöht (A) und ein Teil davon in Ubichinol umgewandelt (B). Der Sauerstoffverbrauch von Keratinocyten - als Indikator der Energieerzeugung -erhöht sich bei CoQ10-Behandlung (C). Control: PLacebobehandlung, Formula 1: Creme 348 µM CoQ10, Formula 2: 870 µM CoQ10 (Bilder aus Anja Knott et al., 2015. . https://doi.org/10.1002/biof.1239, Lizenz cc-by-nc.) |
Ein seit mehr als 140 Jahren bestehendes, sehr erfahrenes dermatologisches Unternehmen hat das Eindringen von CoQ10 in die Epidermis der Haut, die dort erzielte Konzentration, die Wirkung auf die Energieerzeugung und die antioxidative Funktion versus Placebo geprüft.
Dazu wurden CoQ10 (Chinonform) in 2 verschiedenen Formulierungen (Creme und Serum) und entsprechende Placeboproben auf jeweils mehrere Stellen des linken und rechten Unterarms von 16 Versuchspersonen 2 x täglich 14 Tage lang aufgetragen. Nach dieser Zeit wurde das in der Epidermis vorhandene Ubichinon und Ubichinol bestimmt.
Durch die Behandlung mit der Creme, aber vor allem mit dem Serum wurde der Ubichinon-Gehalt in der Epidermis fast bis auf das Doppelte erhöht (Abbildung 4A) und ein Teil davon dort auch in das antioxidative Ubichinol umgewandelt (Abbildung 4B). In Keratinozyten - den Hauptzellen der Epidermis - stieg der Sauerstoffverbrauch (OCR; als Maß für die Energieerzeugung) durch die CoQ10-Zufuhr um 38 % (Abbildung 4C). Neben der gesteigerten Energieerzeugung erwies sich auch die antioxidative Wirkung von CoQ10: Freie Radikale in der gestressten Haut der Probanden wurden durch die Behandlung um bis zu 10 %gesenkt (ohne Bild).
Artikel über CoQ10 im ScienceBlog:
Inge Schuster, 15.02.2018: Coenzym Q10 kann der Entwicklung von Insulinresistenz und damit Typ-2-Diabetes entgegenwirken.
Schrumpfen statt schlafen - wie die Spitzmaus im Winter Energie spart
Schrumpfen statt schlafen - wie die Spitzmaus im Winter Energie spartDo, 14.09.2023 - — Christina Beck 
![]()
In den 1940er-Jahren untersucht der polnische Zoologe August Dehnel in der Wirbeltiersammlung seiner Universität die Schädel von Spitzmäusen. Dabei macht er eine erstaunliche Entdeckung: Die Schädelgröße der Tiere verändert sich im Jahresverlauf! Schädel von Individuen, die im Frühjahr und Sommer gefangen wurden, sind größer als diejenigen von „Wintertieren“. Dehnel vermutet, dass die saisonalen Unterschiede etwas mit der Anpassung an die kalte Jahreszeit zu tun haben. Im Jahr 1949 veröffentlicht er seine Beobachtungen im Fachblatt der Universität, die aber kaum Beachtung finden. Erst vor rund 10 Jahren nehmen Dina Dechmann und ihr Team (Max-Planck-Institut für Verhaltensbiologie) die Arbeit an diesem erstaunlichen Phänomen auf und stoßen auf eine neue Strategie des Energiesparens im Winter. Die Zellbiologin Christina Beck (Leiterin der Kommunikation der Max-Planck-Gesellschaft) berichtet darüber.*
Nach seinem Erscheinen findet Dehnels Beitrag zunächst kaum Beachtung. Seine Entdeckung wird sogar als Scheineffekt abgetan: Dehnel sei einem Irrtum aufgesessen, weil kurz vor dem Winter vermehrt große Individuen sterben, vermuten seine Kollegen. Die Studie gerät weitgehend in Vergessenheit, bis vor rund zehn Jahren die Verhaltensökologin Dina Dechmann auf das Phänomen stößt. Dechmann leitet eine Forschungsgruppe am Max-Planck-Institut für Verhaltensbiologie in Radolfzell am Bodensee. Gemeinsam mit ihren Kolleginnen und Kollegen erforscht sie, wie sich Tiere an ihre Umwelt anpassen und ihren Energiehaushalt optimieren: Wie stellen Tiere sicher, dass sie immer genügend Nahrung finden, um zu überleben und Nachkommen aufzuziehen? Welche Strategien verfolgen sie, um Energie zu sparen, wenn nicht genügend Futter vorhanden ist? „Besonders spannend für uns sind kleine Tiere, die eine sehr hohe Stoffwechselrate haben und daher ohnehin am energetischen Limit leben“, sagt Dechmann. Für solche Tiere ist es eine ganz besondere Herausforderung, ihren hohen Energiebedarf ständig zu decken.
Bei gleichwarmen Tieren korreliert die Körpergröße normalerweise umgekehrt mit dem Energieverbrauch: Je kleiner ein Tier, desto höher ist sein relativer Bedarf an Energie. Die Beziehung zwischen Körpergröße und Energieverbrauch ist dabei nicht linear, sondern exponentiell (Abbildung1). Der Grund für dieses Phänomen ist noch nicht vollständig verstanden. Eine Erklärung betrachtet den Wärmehaushalt des Körpers, der sowohl vom Körpervolumen als auch von der Körperoberfläche beeinflusst wird: Je größer das Volumen, desto mehr Zellen sind vorhanden, die Stoffwechsel betreiben und desto mehr Wärme wird im Körperinnern freigesetzt. Kleine Tiere haben jedoch verhältnismäßig mehr Oberfläche. Um ihre Körpertemperatur aufrecht zu halten, müssen sie daher härter arbeiten, um Körperwärme zu produzieren, verlieren aber auch mehr davon. Das kostet Energie.
| Abbildung 1: Stoffwechselraten bei Säugetieren. Ein Maß für die Stoffwechselrate ist der Sauerstoffverbrauch pro Kilogramm Körpergewicht. Kleine Tiere haben wesentlich höhere Stoffwechselraten als große Tiere und damit einen höheren relativen Energiebedarf. © J. Lazaro für MPG / CC BY-NC-SA 4.0 |
Eine Maus, die keine ist
Ein extremes Beispiel ist die Spitzmaus (Abbildung 2) – ein winziges Säugetier, das trotz seines Namens und der äußerlichen Ähnlichkeit nicht zu den Mäusen gehört, sondern mit dem Maulwurf und dem Igel eng verwandt ist (Ordnung „Eulipotyphla“).
Spitzmäuse leben räuberisch und fressen im Sommer vor allem Würmer und Larven. Im Winter, unter ungleich härteren Lebensbedingungen, stehen vor allem Insekten und Spinnentiere auf dem Speiseplan. Die kleinen Räuber sind andauernd in Bewegung: „Spitzmäuse sind winzige Tierchen, die wie kleine Porsches ständig hochtourig fahren und deshalb fortlaufend viel und qualitativ hochwertige Nahrung brauchen“, sagt Dina Dechmann. „Unter allen Säugetieren haben sie den höchsten, je gemessenen Stoffwechsel. Sie sind kleine Hochleistungsathleten, die ausschließlich über schnelle, aerob arbeitende Muskelfasern verfügen – und deshalb nie Muskelkater bekommen, obwohl sie sich ständig unter hohem Energieaufwand bewegen.“ So ein Leben auf der Überholspur hat jedoch seinen Preis: Finden die Tiere kein Futter, verhungern sie binnen fünf Stunden. Für die winzigen Säuger ist Energiesparen im Winter daher eine Frage des Überlebens.
| Abbildung 2: Die Spitzmaus. © Christian Ziegler |
Um die kalte Jahreszeit zu überstehen, haben Tiere ganz unterschiedliche Strategien entwickelt: Zugvögel verlassen Jahr für Jahr ihre Brutgebiete und fliegen in wärmere Winterquartiere. Fledermäuse, Igel oder Murmeltiere halten Winterschlaf, wobei sie ihre Körpertemperatur drastisch senken. Bei Werten nahe dem Gefrierpunkt sinken die Stoffwechselrate und damit der Energieverbrauch bei manchen Arten bis auf ein Hundertstel der Ausgangswerte. Bei der Winterruhe wird die Körpertemperatur dagegen nur wenig abgesenkt. Tiere, die so überwintern – etwa Braunbären und Eichhörnchen – fallen in einen schlafähnlichen Zustand und sparen dadurch Energie. Zwischendurch wachen sie immer wieder auf, um zu fressen. Eichhörnchen legen dafür im Herbst Futterlager an. Die Spitzmaus verfolgt keine dieser Strategien. Sie wandert nicht aus und behält im Winter ihr Aktivitätslevel bei. Trotzdem sind die kleinen Räuber ausgesprochen erfolgreich, und zwar ausgerechnet in kalten Regionen: Je nördlicher der Breitengrad, desto dominanter ist ihr Einfluss auf die lokale Fauna. Wie machen sie das? War August Dehnel auf der richtigen Spur?
Schrumpfen statt schlafen
Um das herauszufinden, startete Javier Lazaro, Doktorand in Dechmanns Forschungsgruppe, ein aufwändiges Freilandexperiment. Im Sommer fing er in der Umgebung des Instituts mehr als 100 Waldspitzmäuse (Sorex araneus). Die quirligen, im Sommer rund acht Gramm schweren Tiere sind die häufigsten Spitzmäuse in Mitteleuropa und kommen in großen Teilen Europas und im nördlichen Asien vor. Alle gefangenen Tiere wurden geröntgt, um ihre Schädel zu vermessen. Anschließend entließ Lazaro sie wieder in die Freiheit, nachdem er sie zur Wiedererkennung mit reiskorngroßen elektronischen Transpondern ausgestattet hatte. Solche Chips werden in einer größeren Variante auch für Haustiere verwendet. In regelmäßigen Wiederfangaktionen gelang es dem Wissenschaftler, rund ein Drittel der Tiere erneut zu fangen und zu röntgen, um die Veränderungen über die Zeit zu dokumentieren (Abbildung 3). Und tatsächlich: Die Schädel aller untersuchten Spitzmäuse waren im Winter geschrumpft und im darauffolgenden Frühjahr wieder gewachsen! „Die Schädelhöhe nahm im Winter um 15 Prozent ab, manchmal sogar bis zu 20 Prozent“, sagt Javier Lazaro. Und nicht nur die Größe des Schädels verändert sich. Auch lebenswichtige Organe wie Gehirn, Leber, Darm und Milz schrumpfen, sogar Teile des Skeletts werden abgebaut. Die Tiere verlieren dadurch im Winter bis zu einem Fünftel ihres Körpergewichts und verändern ihr Aussehen so stark, dass man sie früher fälschlicherweise sogar für unterschiedliche Arten hielt. Im Frühjahr beginnen die Spitzmäuse dann wieder zu wachsen. Ihre ursprüngliche Gestalt erreichen sie jedoch nicht mehr: „Die Schädelhöhe etwa nimmt lediglich um neun Prozent wieder zu. Dass die Tiere dadurch mit der Zeit nicht immer kleiner werden, liegt daran, dass sie nur 13 Monate lang leben und somit nur einen einzigen Schrumpfungszyklus durchlaufen“, so der Wissenschaftler. „Mit unserer Wiederfangstudie konnten wir erstmals zeigen, dass die saisonalen Veränderungen in jedem einzelnen Tier stattfinden.“ August Dehnel hatte also recht, und mittlerweile sind die von ihm erstmals beschriebenen saisonalen Größenänderungen als Dehnel-Effekt bekannt.
| Abbildung 3: Veränderung der Schädelhöhen. Auf der y-Achse wird das Verhältnis „Schädelhöhe zu Zahnreihe“ dargestellt. Da die Länge der Zahnreihe eines Individuums gleich bleibt, werden somit die Schwankungen in der Körpergröße der vermessenen Spitzmäuse herausgerechnet. Die Linie zeigt die Mittelwerte. Die Kreise zeigen die Messwerte der mehrfach gefangenen Individuen. Rot markiert sind drei Messungen, die an demselben Individuum im Sommer, Winter und Frühjahr vorgenommen wurden. Diese Werte entsprechen den Schädelzeichnungen (oben), die auf Grundlage von Röntgenbildern entstanden. © J. Lazaro für MPG / CC BY-NC-SA 4.0 |
Auch Maulwürfe und Wiesel werden kleiner
Wie aber entsteht ein solches Phänomen im Laufe der Evolution und warum? Um das herauszufinden, suchten die Forschenden auch in anderen Arten – und wurden fündig: Die verwandten Maulwürfe und die im evolutionären Stammbaum weit entfernten Wiesel zeigen dieselben jahreszeitlichen Veränderungen. All diesen Tieren gemeinsam ist, dass sie von hochwertiger Nahrung abhängig sind und keinen Winterschlaf halten. Die Forschenden sehen darin ein starkes Indiz dafür, dass es sich dabei um Konvergenz handelt: Eine konvergente Anpassung tritt bei nicht näher miteinander verwandten Tiergruppen auf, die unter ähnlichen Umweltbedingungen leben und daher denselben Selektionsfaktoren ausgesetzt sind. Die äußeren Bedingungen sind jedoch nicht konstant und können sich im Jahresverlauf ändern – und damit die Richtung, in welche die natürliche Selektion wirkt. Bei der Spitzmaus sind im Winter kleine Tiere im Vorteil, die mit wenig Energie auskommen: „Anhand von Stoffwechselmessungen haben wir gezeigt, dass Spitzmäuse pro Gramm Körpergewicht bei +20 °Celsius genauso viel Energie verbrauchen wie bei -1 °Celsius“, sagt Dina Dechmann. „Eine Spitzmaus, die im Winter um 20 Prozent kleiner ist, spart also auch 20 Prozent Energie.“ Im Frühjahr kehren sich die Verhältnisse um: „Spitzmäuse sind sehr territorial, und dabei sind große Tiere ihren Konkurrenten überlegen. Große Weibchen bringen zudem größere Junge zur Welt, die eine höhere Überlebenswahrscheinlichkeit haben als kleine.“ Als Reaktion auf die unterschiedlichen Anforderungen der Umwelt hat sich bei Spitzmäusen die Fähigkeit herausgebildet, die Körpergröße im Jahresverlauf zu verändern. „Die Schrumpfung ist dabei keine Folge von Hunger, sondern ein genetisches Programm, das bereits im August startet, wenn noch ideale Futterbedingungen herrschen“, sagt die Forscherin. „Dieses Programm kann durch die Umwelt jedoch modifiziert werden.“
Um herauszufinden, welche Umweltfaktoren dafür entscheidend sind, verglichen die Forschenden zwei Maulwurfarten aus unterschiedlichen Klimazonen. Anhand von Schädelmessungen in Museumssammlungen wiesen sie nach, dass der Europäische Maulwurf im Winter schrumpft. Der Iberische Maulwurf, der in Spanien und Portugal vorkommt, bleibt dagegen das ganze Jahr über gleich groß – und das, obwohl auch er zeitweise mit Nahrungsknappheit zu kämpfen hat. Der Futtermangel trifft ihn jedoch während der heißen Jahreszeit. „Wenn es lediglich eine Frage der Nahrung wäre, dann müsste der Europäische Maulwurf im Winter schrumpfen, der Iberische Maulwurf dagegen im Sommer, wenn große Hitze und Trockenheit das Futter knapp machen“, sagt Dechmann. Das Forschungsteam geht daher davon aus, dass nicht allein der Nahrungsmangel, sondern auch das kältere Klima ausschlaggebend ist.
Gehirnverlust mit Folgen
Über die Anpassung von Tieren an kalte Klimazonen hat sich im 19. Jahrhundert bereits der deutsche Anatom Carl Bergmann Gedanken gemacht. Von ihm stammt die sogenannte Bergmannsche Regel. Sie sagt voraus, dass Vögel und Säugetiere, die in kalten Regionen leben, normalerweise größer sind als ihre Verwandten in warmen Gegenden. Grund dafür ist ihr Oberflächen-Volumen-Verhältnis: Da mit zunehmender Größe das Volumen eines Körpers stärker ansteigt als seine Oberfläche, haben große Tiere in kalten Regionen einen Vorteil, weil sie relativ gesehen weniger Wärme abgeben als kleine. Spitzmäuse folgen dieser Regel nicht – im Gegenteil: Bei ihnen sind Individuen derselben Art im Norden sogar noch kleiner als im Süden. Doch trotz ihres energetisch ungünstigen Oberflächen-Volumen-Verhältnisses sind die kleinen Räuber gerade in kalten Regionen häufig. Ihre Strategie, durch saisonale Schrumpfung Energie zu sparen, scheint also aufzugehen. Dafür spricht auch, dass Dehnels Phänomen bei Waldspitzmäusen im Nordosten Polens stärker ausgeprägt ist als bei ihren Verwandten am klimatisch milderen Bodensee. Eine vergleichbare regionale Variabilität haben die Max-Planck-Forschenden auch bei Wieseln gefunden. Bei ihren weiteren Untersuchungen konzentrierten sich Dina Dechmann und ihr Team auf das Gehirn – dasjenige Organ, das bei der Spitzmaus im Vergleich zu anderen Organen besonders stark schrumpft. Bereits August Dehnel hatte anhand seiner Schädelmessungen errechnet, dass das Volumen des Spitzmausgehirns im Winter um 20 Prozent kleiner ist als im Sommer. Mehr als sieben Jahrzehnte später setzten die Max-Planck-Forschenden auf moderne medizinische Diagnoseverfahren, um individuelle Veränderungen des Gehirns sichtbar zu machen. In einem Gemeinschaftsprojekt mit dem Universitätsklinikum Freiburg vermaßen sie einzelne Spitzmäuse wiederholt im Magnetresonanztomografen (Abbildung 4). Es war bereits bekannt, dass nicht alle Gehirnbereiche in gleichem Maße schrumpfen. Die Veränderungen betreffen vielmehr ganz bestimmte Regionen – den Neocortex und den Hippocampus. Der Neocortex ist ein Teil der Großhirnrinde und unter anderem für die Verarbeitung von Sinneseindrücken zuständig, der Hippocampus ist wichtig für die räumliche Orientierung. „Wir haben herausgefunden, dass der Neocortex um ganze 25 Prozent schrumpft, der Hippocampus um 20 Prozent“, sagt Dina Dechmann. Welche Folgen hat es, wenn das Gehirn in solch wichtigen Bereichen derart stark abgebaut wird? Um das zu testen, starteten die Forschenden eine Reihe von Verhaltensexperimenten, deren Resultate sie dann mit den MRT-Messungen im gleichen Tier vergleichen konnten. „Dieses Projekt war eine echte Herausforderung, denn Spitzmäuse sind sehr stressempfindlich und anspruchsvoll in der Haltung“, sagt Dechmann. „Bisher ist es auch nicht gelungen, sie in Gefangenschaft zu züchten.“ Die Forschenden mussten daher mit Wildfängen arbeiten. Im Experiment ließen sie Spitzmäuse, die sie zu verschiedenen Jahreszeiten im Freiland gefangen hatten, in einer Versuchsarena nach Mehlwürmern suchen. Anhand von bestimmten visuellen Hinweisen sollten sich die Tiere merken, wo die Leckerbissen versteckt waren. Dabei zeigte sich, dass sich die saisonalen Veränderungen der Gehirngröße tatsächlich auf die kognitiven Fähigkeiten der Tiere auswirkt: „Im Sommer lernten die Spitzmäuse schneller, wo sich die Futterquelle befand und brauchten in aufeinanderfolgenden Versuchen weniger Zeit, um sie zu finden“, sagt Dina Dechmann. „Im Winter schnitten sie dagegen deutlich schlechter ab.“
| Abbildung 4: Veränderung der Knochendichte. Computertomographische Aufnahme des Schädels eines Individuums im Sommer und Winter. Die Farben zeigen die unterschiedlichen Knochendichten. Im Wintertier sind die Plattennähte des Schädels und die Platte am Hinterhaupt demineralisiert. © C. Dullin / CC BY-NC-SA 4.0 |
Die Forschenden schließen daraus, dass das Ortsgedächtnis der Tiere tatsächlich unter der Schrumpfung leidet: „Wir glauben, dass die Spitzmäuse sich das leisten können, weil ihre Umwelt im Winter weniger komplex wird“, sagt Dina Dechmann. „Im Sommer verändert sich die Vegetation schnell, die Spitzmäuse bewegen sich großräumig und müssen sich daher viel merken. Im Winter sind die Tiere dagegen viel kleinräumiger unterwegs und suchen teils versteckt unter der Schneedecke nach Futter.“ In der kalten Jahreszeit überwiegen daher die Vorteile, die ein kleines Gehirn aufgrund seines geringeren Energieverbrauchs bietet. Biologen sprechen von einem Trade-off (dt. Ausgleich, Kompromiss) zwischen Energieoptimierung und kognitiver Leistung: Bei Eigenschaften, die sich wechselseitig beeinflussen und die miteinander in Konflikt stehen, bildet sich im Lauf der Evolution ein Kompromiss heraus, der das Überleben und den Fortpflanzungserfolg eines Tieres oder einer Pflanze bestmöglich sichert. Vor allem bei Säugetieren ist das Denkorgan ein wahrer Energiefresser: Beim Menschen hat es einen Anteil von zwei Prozent am Körpergewicht, verbraucht aber ganze 20 Prozent der Gesamtenergie. „Der Kompromiss, durch eine Verkleinerung des Gehirns Energie zu sparen, obwohl das zu messbaren Einschränkungen der kognitiven Leistung führt, zeigt, wie extrem anpassungsfähig Säugetiere sein können“, sagt Dechmann.
Neue Ansätze für die Medizin
Aktuell untersuchen die Max-Planck-Forschenden, was bei den saisonalen Veränderungen der Spitzmaus auf physiologischer Ebene passiert: Wie schaffen es die Tiere, Knochen und Organe zu schrumpfen und dann wieder wachsen zu lassen? Und wodurch wird der Vorgang ausgelöst? Neue Experimente zu Stoffwechsel und Genexpression sollen Licht ins Dunkel bringen. Diese Abläufe im Detail zu verstehen, ist nicht nur für Biologinnen und Biologen interessant – auch die Medizin könnte davon profitieren: „So große, vor allem umkehrbare Veränderungen im Skelett könnten für die Knochenrekonstruktion oder Osteoporose-Forschung wichtig sein“, sagt. Dina Dechmann. Bei dieser Erkrankung verliert das Skelett an Substanz und wird brüchig. Weitere Erkenntnisse über diesen Abbauprozess, der in der Spitzmaus umkehrbar ist, könnten daher nützlich sein, um neue Therapieansätze zu entwickeln. Dasselbe gilt für neurodegenerative Erkrankungen wie Alzheimer, die mit einem Substanzverlust im Gehirn einhergehen. Dina Dechmann und ihr Team arbeiten daher mit Forschenden der Universitäten Stony Brook und Aalborg zusammen. „Spitzmäuse könnten schon in naher Zukunft wichtige Modellorganismen für unterschiedliche medizinische Forschungszweige werden“, glaubt die Max-Planck-Forscherin. Die winzigen Räuber sind daher nicht zuletzt ein Beispiel dafür, wie Grundlagenforschung zu neuen Erkenntnissen in der Medizin führen kann.
* Der Artikel von Christina Beck unter Mitwirkung von Dr. Elke Maier (Redaktion Max-Planck-Forschung) ist erstmals unter dem Titel: "Energie sparen leicht gemacht - Warum Spitzmäuse im Winter schrumpfen" in BIOMAX 38, Herbst2023 erschienen https://www.max-wissen.de/max-hefte/biomax-38-energie-sparen-spitzmaus/ und wurde (mit Ausnahme des Titels) unverändert in den Blog übernommen. Der Text steht unter einer CC BY-NC-SA 4.0 Lizenz.
Veröffentlichung von Dina Dechmann und Team:
Jan R. E. Taylor, Marion Muturi, Javier Lázaro, Karol Zub, Dina K. N. Dechmann: Fifty years of data show the effects of climate on overall skull size and the extent of seasonal reversible skull size changes (Dehnel's phenomenon) in the common shrew. Ecology and Evolution. https://doi.org/10.1002/ece3.9447
Vortrag von Dina Dechmann:
Wie passen sich Tiere veränderter Nahrung an? Video 12:00 min https://www.br.de/mediathek/podcast/campus-talks/pd-dr-dina-dechmann-wie-passen-sich-tiere-veraenderter-nahrung-an/1865539
Warum ich mir keine Sorgen mache, dass ChatGTP mich als Autorin eines Biologielehrbuchs ablösen wird
Warum ich mir keine Sorgen mache, dass ChatGTP mich als Autorin eines Biologielehrbuchs ablösen wirdFr, 08.09.2023 — Ricki Lewis
Die Genetikerin Ricki Lewis hat Tausende Artikel in wissenschaftlichen Zeitschriften verfasst und eine Reihe von voluminösen Lehrbüchern geschrieben, die zu Bestsellern wurden und bereits mehr als ein Dutzend Auflagen erreicht haben. Ihre leicht verständlichen Schriften vermitteln den Lesern nicht nur sorgfältigst recherchierte Inhalte, sondern auch die Begeisterung für Wissenschaft und die zugrundeliegende Forschung, wobei Sichtweise, Erfahrung, Wissen und auch Humor der Autorin miteinfließen. Dass ChatGTP eine Konkurrenz für sie als Autorin werden könnte, ist für Ricki Lewis nicht vorstellbar.*
Eben habe ich ChatGTP (https://chat.openai.com/auth/login) zum ersten Mal benutzt. Anfangs habe ich mir Gedanken um meine Zukunft gemacht, denn der Chatbot beantwortete fast augenblicklich meine Fragen zu immer obskurer werdenden Begriffen aus meinem Fachgebiet, der Genetik,. Das KI-Tool zu überlisten, hat allerdings nur etwa 10 Minuten gedauert.
ChatGTP - "Chat Generative Pre-trained Transformer" - wurde am 30. November 2022 von OpenAI/Microsoft freigegeben.(Die Bezeichnung klingt ein bisschen wie das, was Google über Steroide sagt.) Nach meiner kurzen Begegnung mit dem Chatbot muss ich mich aber fragen, ob dieser die Nuancen, den Kontext, den Humor und die Kreativität eines menschlichen Geistes schaffen kann. Könnte er mich als Autorin von Lehrbüchern ersetzen?
Ich schreibe schon seit langem dicke Wälzer über Biowissenschaften
Mein Lieblingsbuch war immer "Human Genetics: Concepts and Applications"; die erste Auflage ist 1994 erschienen, als die Ära der Sequenzierung des menschlichen Genoms begann. Die 14. Auflage wurde diese Woche von McGraw-Hill herausgegeben. Eine Überarbeitung dauert gewöhnlich zwei Jahre: ein Jahr für Aktualisierung und Berücksichtigung der Vorschläge der Rezensenten, ein weiteres für die "Produktion" vom Lektorat bis zur Endfassung. Dann folgt ein Jahr Pause.
| Abbildung 1. Ricki Lewis: Humangenetik Konzepte und Anwendungen. 12 Auflagen des Lehrbuchs von 1994 bis 2017 (Lizenz cc-by) |
Mit der Entwicklung der Genetik zur Genomik trat die künstliche Intelligenz auf den Plan und übertrug die kombinatorischen Informationen der vergleichenden Genomik auf die DNA-Sequenzen. Mit einem Training an Datensätzen und einer anschließenden Mustererkennung ließen sich Evolutions-Bäume ableiten, welche die Beziehungen zwischen den Spezies abbildeten beim Testen der Abstammung und in der Forensik und ebenso bei der Identifizierung von mutierten Sequenzen, die auftreten, wenn sich das Krebsgeschwür ausbreitet.
ChatGPT ist für mich zu neu, als dass ich bei der Überarbeitung der neuen Auflage davon Gebrauch gemacht hätte, aber ich bin nun neugierig geworden. Ich könnte mir vorstellen, dass der Bot Definitionen ausspuckt, aber ein Lehrbuch ist viel mehr als nur "Inhalt". Ein menschlicher Autor fügt seine Sichtweise, Erfahrung und vielleicht sein Wissen hinzu, das über das hinausgeht, was ChatGPT aus dem Internet entnehmen kann.
Genetik-Lehrbücher und Generative künstliche Intelligenz
Die Genforschung erzeugt und extrahiert Unmengen an Informationen, Millionen und sogar Milliarden an Datenpunkten. Man trainiert einen Algorithmus an den DNA-Sequenzen eines bekannten krankheitsverursachenden Gens und sucht dann in Zellen anderer Personen nach identischen oder sehr ähnlichen Sequenzen, um die Diagnose zu unterstützen.
Die Art von KI, die Lehrbücher neu schreiben könnte, wird generativ genannt - es ist das G in GTP (zufällig auch eine Abkürzung für einen der vier DNA-Bausteine). ChatGTP lernt die Muster und Strukturen von "Trainingsdaten" und generiert ähnliche Kombinationen aus neuen Daten und erzeugt Texte, Bilder oder andere Medien.
Könnte ChatGTP also ein Lehrbuch wie das meine schreiben?
Das glaube ich nicht. Ich kann mir vorstellen, dass die generative KI einen Roman schreibt, der den Romanen der bekannten Autorin Colleen Hoover (US-amerikanische Bestseller-Autorin; Anm. Redn.) ähnelt. Tatsächlich habe ich vor langer Zeit eine Fantasy-Erzählung im Playgirl veröffentlicht, nachdem ich Wörter und Sätze in ähnlichen Artikeln aufgelistet und ein neues Szenario entworfen hatte. Es ging darin um einen Tornado und eine Schubkarre im ländlichen Indiana.
Wie Colleen Hoovers Belletristik und Playgirl-Fantasien haben auch Lehrbücher einen ganz eigenen Stil. Lehrbücher enthalten aber viel mehr als nur eine einzige Erzählung pro Kapitel. Die Herstellung umfasst auch die Auswahl von Fotos, die Gestaltung von Illustrationen und die Erstellung von pädagogischen Hilfsmitteln - Fragen, Zusammenfassungen, Verweise, Texte in Boxen.
Hier folgt nun also ein kurzer Überblick über den Werdegang der Lehrbuchveröffentlichung und anschließend das, was - meiner Meinung nach- künstliche Intelligenz nicht so gut wie ein menschlicher Autor leisten kann.
Das Werden eines Lehrbuchs über Biowissenschaften
Mein erstes Lehrbuch, Life, war ein Einführungsbuch in die Biologie (nicht zu verwechseln mit Keith Richards' Autobiografie mit demselben Titel). Damals waren die Marketing-Leute mit allem möglichen Schnickschnack ausgerüstet, um den Marktanteil zu erhöhen - Probenvorräte, Handbücher für Lehrpersonal, Arbeitsbücher für Fallstudien. Was für ein Unterschied gegen heute! Elektronische Lehrbücher sind mit Multiple-Choice-Fragen zum "adaptiven Lernen" ausgestattet. Der Lernende (früher Student genannt) erhält sofortige Rückmeldung darüber, warum jede falsche Antwort falsch ist.
Jede Auflage hat neue Schriftarten, Designelemente und Farbpaletten gebracht, um die neue Version anders aussehen zu lassen, weil sich einige Themen - Mitose, Mendel, DNA - ja nicht ändern. Umfangreiche Wälzer wurden in kürzere Versionen aufgeteilt- wie kalbende Eisberge.
Die ersten E-Books stammen aus den 1990er Jahren. Heute ist in den Studiengebühren eine Gebühr für die zeitlich begrenzte Lizenzierung eines E-Lehrbuchs enthalten. "E" könnte auch für "eintägig" stehen.
Für die 14. Auflage meines Buches hat ein Unternehmen alles, was ich geschrieben habe, unter dem Blickwinkel von Diversität, Gleichberechtigung und Inklusivität (DGI) unter die Lupe genommen. In einem PLOS-Blogbeitrag vom April d.J. habe ich berichtet, wie ich meine Fauxpas entdeckt habe https://dnascience.plos.org/2023/04/27/embracing-diversity-equity-and-inclusion-in-genetics-textbooks-and-testing/.
Da herkömmliche Lehrbücher kostspielig sind, gibt es gelegentlich Bestrebungen, sie zu ersetzen. Aber einen Kurs aus Online-Materialien oder aus Mitschriften von Vorlesungen und Prüfungsfragen zusammenzuschustern, erfordert mehr Zeit und Mühe, als den meisten angehenden Autoren bewusst ist. Und den kostenlosen Online-Lehrbüchern, die vor einigen Jahren aufkamen, fehlte die redaktionelle Kontrolle und die der Reviewer, die ein akademischer Verlag bietet.
Eine bei der Erstellung eines Lehrbuchs weniger konkretisierbare Fähigkeit ist das angeborene Schreibtalent. Stilelemente sind subtil. Wie viele Akademiker oder ChatGTP wechseln vom Passiv ins Aktiv? Formuliert man um, um "es gibt" und andere Redundanzen zu vermeiden? Um zu häufige Verwendung von Wörtern zu unterlassen? Um die Länge von Sätzen, von Absätzen zu ändern? Um das Material in logische A-, B- und C-Abschnitte zu gliedern?
Kann KI die Kreativität eines menschlichen Geistes nachahmen?
Künstliche Intelligenz kann schnell eine Tabelle mit DNA-Replikationsenzymen zusammenstellen oder eine Chronik für Technologien erstellen. Aber wie könnte ein noch so gut trainierter Algorithmus die Auswahl der Beispiele eines Autors nachahmen oder Fallstudien entwickeln, die auf persönlichen Interviews mit Menschen mit genetischen Krankheiten basieren?
Würde KI meine Stammzellen-Vergleiche verbessern?
"Der Unterschied zwischen einer Stammzelle und einer Vorläuferzelle ist ein wenig so, wie wenn ein Studienanfänger viele Studienfächer in Betracht zieht, während ein Student sich dann bei der Wahl seiner Kurse eher auf einen bestimmten Bereich konzentriert. Die Reprogrammierung einer Zelle ist vergleichbar mit der Entscheidung eines Studienanfängers, das Hauptfach zu wechseln. Will ein Student mit Hauptfach Französisch Ingenieur werden, so müsste er neu beginnen und ganz andere Kurse belegen. Aber ein Biologiestudent, der Chemie studieren möchte, müsste nicht bei Null anfangen, weil viele gleiche Kurse für beide Studiengänge gelten. So ist es auch bei den Stammzellen."
Wie sieht es mit der Pädagogik aus? KI könnte Formulare zum Ausfüllen oder Multiple-Choice-Fragen ausspucken. Aber könnte sie meine Übung zum kritischen Denken mit Venn-Diagrammen (Darstellung von Mengen mit Kreisen; Anm. Redn.) von drei SARS-CoV-2-Varianten mit einigen gemeinsamen Mutationen erstellen? Ich bitte dort den Leser, die Regeln des genetischen Codes anzuwenden, um vorherzusagen, welche Veränderungen die öffentliche Gesundheit am ehesten bedrohen werden.
Ich versuche, meine Fragen unterhaltsam zu gestalten.
Würde ChatGPT am Ende eines Kapitels Fragen zur Vererbung von drahtigem Haar bei den Tribbles aus Star Trek stellen? Bei einer Familie im Allgemeinen Krankenhaus eine seltene Blutgruppe aufspüren? Einen Stammbaum für SORAS (Soap Opera Rapid Aging Syndrome) erstellen, das Leiden, das die Familie Newman in der Seifenoper "Schatten der Leidenschaft" befällt?
Anleihen bei Science-Fiction werden in einem Kapitel über Evolution fortgesetzt, in dem die Lernenden aufgefordert werden, das Prinzip zu identifizieren, das diese Themen veranschaulichen:
In "Seveneves" von Neal Stephenson zerspringt der Mond. In zwei Jahren werden die Bruchstücke auf die Erde prallen und den Planeten für Jahrhunderte unbewohnbar machen. Einige Menschen, die bereits auf riesigen Raumstationen leben, überleben, und andere werden ausgewählt, um sich ihnen anzuschließen. Alle anderen sterben unter der Flut von Mondschrott und der großen Hitze. Oben, auf der "Wolkenarche", schwindet die menschliche Spezies dahin, aber entsteht wiederausgehend von sieben überlebenden Frauen, die mit Hilfe von Fortpflanzungstechnologien Kinder bekommen. Fünftausend Jahre nach der Explosion des Mondes ist die menschliche Bevölkerung wieder auf 3 Milliarden angewachsen, bereit, eine geheilte Erde zu bewohnen. (EIN BEVÖLKERUNGSENGPASS)
In "Children of the Damned" werden plötzlich alle Frauen einer Kleinstadt von genetisch identischen Wesen von einem anderen Planeten geschwängert. (NICHT ZUFÄLLIGE PAARUNG)
In Dean Koontzis "The Taking" töten riesige mutierte Pilze fast alle Menschen und verschonen nur kleine Kinder und die wenigen Erwachsenen, die sie beschützen. Die menschliche Rasse muss sich aus den Überlebenden neu formieren. (GRÜNDEREFFEKT)
Was ist mit der Geschichte? Die KI könnte leicht Chronologien zusammenstellen. Aber würde sie die Entschlüsselung des genetischen Codes durch Marshall Nirenberg und Heinrich Matthaei im Jahr 1961 in einer "Glimpse of History"-Box mit der Erfindung des ersten mRNA-basierten Impfstoffs durch Katalin Karikó und Drew Weissman verbinden?
Und schließlich: Kann KI von Humor Gebrauch machen? Würde sie eine Frage am Ende eines Kapitels wie diese über forensische DNA-Tests stellen?
"Rufus, der Kater, wurde von seinen Besitzern in einer Mülltonne gefunden. Sein Körper war mit Schnitten und Bisswunden übersät, und an seinen Krallen klebten graue Fellreste - graues Fell, das dem Fell von Killer, dem riesigen Hund von nebenan, sehr ähnlich sah."
ChatGPT -eine Abwägung
ChatGPT zu testen war einfach. Es verdient eine Eins plus bei der Ausgabe von Definitionen obskurer Fachbegriffe wie Tetrachromasie (verbessertes Farbsehen durch eine vierte Art von Zapfenzellen) und Chromothripsis (zersplitterte Chromosomen).
ChatGPT unterscheidet genau zwischen Gentherapie und Gen-Editierung. Das Tool vereinfacht die Gentherapie nicht zu sehr auf das "Ersetzen" eines Gens, sondern antwortet: "Bei der Gentherapie werden neue oder veränderte Gene in die Zellen einer Person eingeführt, um ein fehlerhaftes Gen zu korrigieren oder zu ersetzen oder um eine therapeutische Funktion bereitzustellen." Diese Definition deckt alle Grundlagen ab.
In meiner neuen Lehrbuchauflage gibt es eine Box mit Informationen darüber, wie sich die Viren von Grippe und COVID unterscheiden. Auch hier liefert ChatGPT mehr, als ich über die beiden Erreger wissen möchte, und vergleicht und setzt sie in Gegensatz. Ich könnte mir vorstellen, dass ein Student die Antwort in einer Hausarbeit verwendet - ich bin froh, dass meine Zeit als Professor vorbei ist!
ChatGPT unterscheidet klar zwischen Fahrer- und Mitfahrer-Mutationen bei Krebs, gleichwohl beginnt meine Lehrbuchdefinition mit dem Zusammenhang: "Der Fahrer eines Fahrzeugs bringt es zum Ziel, der Beifahrer fährt mit".
Ein Haftungsausschluss für ChatGPT lautet: "Trotz unserer Sicherheitsvorkehrungen kann ChatGPT Ihnen ungenaue Informationen liefern." Offenbar macht er auch Fehler durch Auslassungen; dies stellte ich fest, als ich nach etwas anderem fragte, über das ich geschrieben hatte: wie man einen Impossible Burger macht. Nicht nur die Zutaten, sondern auch die Biotechnologie hinter dieser genialen Erfindung.
Die Erklärung von ChatGPT beginnt genau genug: "Der Impossible Burger ist ein pflanzliches Burger-Patty, das den Geschmack und die Beschaffenheit eines herkömmlichen Rindfleisch-Burgers imitieren soll. Ich habe zwar keinen Zugang zu dem genauen Rezept oder Verfahren, das von Impossible Foods verwendet wird, aber ich kann einen allgemeinen Überblick darüber geben, wie pflanzliche Burger wie der Impossible Burger normalerweise hergestellt werden." Anschließend werden allgemeine Schritte zur Herstellung von Variationen des traditionellen Veggie-Burgers angeführt, die man in Supermärkten findet. Aber Impossible-Burger sind überhaupt nicht wie andere!
Das ist einfach nicht gut genug, trotz der vermenschlichenden Wirkung der Antwort in der ersten Person. ChatGPT hat offenbar meinen Artikel "Anatomy of an Impossible Burger" nicht gelesen, den ich im Mai 2019 in PLOSBlogs DNA Science veröffentlicht habe. Das ist die einfachste Überschrift, die mir einfallen konnte.
Meine Quelle? Die Datenbank des Patent- und Markenamts! Die Suche hat nur ein paar Minuten gedauert. Die Patentanmeldung umfasst 52 Seiten und wurde 2017 nach jahrelangen Forschungsarbeiten eingereicht. Sie enthält Hunderte von verwandten Patenten und Veröffentlichungen, viele davon in den Mainstream-Medien. ChatGTP konnte nichts finden?
Das Werkzeug hatte keinen Zugang zu dem genauen Rezept oder Verfahren? Bei diesem Ansatz werden Hefezellen genetisch verändert, um eine Sojabohnenversion von Hämoglobin, das sogenannte Leghämoglobin, zu produzieren, das das "Mundgefühl" und das Aussehen von totem Kuhfleisch vermittelt.
Ich habe nicht nur 2019 über den Impossible Burger gebloggt, sondern auch eine Version in der dreizehnten Auflage meines Lehrbuchs veröffentlicht, also vor drei Jahren!
Aber ich bin erleichtert und nicht beleidigt, dass ich unter dem ChatGTP-Radar fliege, denn es ist schön zu wissen, dass meine Fähigkeiten noch nicht veraltet sind.
Allerdings habe ich ein Problem mit dem Schreiben von ChatGPT. In früheren Entwürfen kam es immer wieder als GTP aus meinem Gehirn, vielleicht nach dem DNA-Nukleotid GTP - Guanosintriphosphat.
*Der Artikel ist erstmals am 7. September 2023 in PLOS Blogs - DNA Science Blog unter dem Titel " Why I’m Not Worried that ChatGTP Will Replace Me as a Biology Textbook Author " https://dnascience.plos.org/2023/09/07/why-im-not-worried-that-chatgtp-will-replace-me-as-a-biology-textbook-author/ erschienen und steht unter einer cc-by Lizenz . Die Autorin hat sich freundlicherweise mit der Übersetzung ihrer Artikel durch ScienceBlog.at einverstanden erklärt. Der ungekürzte Artikel folgt so genau wie möglich der englischen Fassung.
Ricki Lewis im ScienceBlog:
Links zu den bisherigen 31 Artikel aus PLOS Blogs - DNA Science Blog: https://scienceblog.at/ricki-lewis
Epilepsie - wie Larven des Schweinebandwurms auf das Gehirn einwirken
Epilepsie - wie Larven des Schweinebandwurms auf das Gehirn einwirkenDo, 31.08.2023 — Redaktion
Vor wenigen Tagen erregte ein Presseartikel besonderes Aufsehen: in Australien wurde einer 64-jährigen Frau ein lebender Wurm operativ aus dem Gehirn entfernt, der aber offensichtlich noch keinen größeren Schaden verursacht hatte. Viel weniger öffentliche Aufmerksamkeit finden dagegen die weltweit auftretenden Infektionen mit dem Schweinbandwurm, in deren Verlauf die Wurmlarven Zysten im Gehirn bilden und in Folge Epilepsie auslösen können. Eine neue Arbeit in Journal eLife zeigt nun erstmals auf , dass die Wurmlarven durch Freisetzung des Neurotransmitter Glutamin eine wesentliche Rolle in der Pathophysiologie der Epilepsie spielen dürften.*
Infektionen mit dem Schweinebandwurm Taenia solium
(Taeniasis) treten weltweit auf, insbesondere in Ländern mit niedrigem und mittlerem Einkommen. Menschen infizieren sich, wenn sie rohes oder nicht durchgekochtes Fleisch von Schweinen verzehren, das Larven des Bandwurms, sogenannte Zystizerken enthält. Im Darm reifen die Zystizerken innerhalb weniger Monate zu segmentierten Bandwürmern heran, können dort Jahre verbleiben und Segmente (Proglottide) mit Eiern und auch isolierte Eier mit dem Kot absondern. Bandwurmeier, die von Bandwurmträgern im Kot ausgeschieden werden, können unter schlechten hygienischen Bedingungen über den fäkal-oralen Weg oder durch die Aufnahme von kontaminierten Lebensmitteln oder Wasser aufgenommen werden und sind direkt für Menschen und ebenso für Schweine infektiös. Sie werden zu sogenannten Oncosphären, unreifen Larvenformen, welche die Darmwand penetrieren, im System zirkulieren und sich als Zystizerken in Muskeln, Leber, Haut, Augen bis hin ins Zentralnervensystem festsetzen. Es kommt zur Zystizerkose mit zum Teil verheerenden Folgen für die menschliche Gesundheit.
Im Jahr 2015 hat die WHO-Gruppe für Epidemiologie der lebensmittelbedingten Erkrankungen T. solium als eine der Hauptursachen für lebensmittelverursachte Todesfälle identifiziert. Insgesamt dürften weltweit zumindest um die 5 Millionen Menschen an Zystizerkose leiden. Abbildung 1.
| Abbildung 1. Weltweite Verbreitung der Zystizerkose 2019; Fallzahlen pro 100 000 Personen. Im Insert: vergrößertes Bild von Europa. Die Infektion wird durch Larvenformen des Schweinebandwurms verursacht und führt zur Zystenbildung in Muskelgewebe, anderen Organen und Gehirn. Ein Vergleich der Fallzahlen von 1990 und 2019 zeigt, dass die Infektionen in allen betroffenen Ländern stark abgenommen haben; weltweit von 71,3/100 000 auf 59,1/100 000 Personen. n (Bild: Our World in Data, https://ourworldindata.org/grapher/prevalence-cysticercosis. Lizenz: cc-by) |
Neurozystizerkose
Bilden die Larven des Parasiten Zysten im Gehirn - dies wird als Neurozystizerkose bezeichnet -, so können Krampfanfälle und Epilepsie die Folge sein. Tatsächlich gehört die Neurozystizerkose weltweit zu den häufigsten Ursachen für erworbene Epilepsie und ein erheblicher Anteil (22-29 %) aller Epilepsiepatienten in Ländern südlich der Sahara hat eine Neurozystizerkose. Abbildung 2 gibt einen Eindruck wie sehr das Gehirn eines Patienten von Zystizerken befallen sein kann.
| Abbildung 2. . Neurozystizerkose: multiple Zystizerken im Gehirn eines Patienten; Magnetresonanz-Aufnahme. (Bild: Centers for Disease Control and Prevention (CDC), gemeinfrei) |
Man nimmt an, dass Krampfanfälle und Epilepsie auftreten, wenn die Zysten platzen und ihren Inhalt in das Gehirn entleeren; frühere Untersuchungen zur Entstehung dieser Erkrankungen haben sich auf die neuroinflammatorische Reaktion des Gehirns konzentriert. Interessanterweise geht man davon aus, dass Taenia solium-Larven die lokale Immunreaktion gegen ihre Vorhandesein aktiv unterdrücken, so dass sie sich über Monate oder sogar Jahre im Gehirn halten können. Abgesehen von den Arbeiten zur Neuroinflammation wurden jedoch die Auswirkungen der Larven (und ihrer Sekrete) auf die neuronale Aktivität bisher kaum erforscht. Jetzt berichtet im Fachjournal eLife ein internationales Forscherteam unter Joseph Raimondo (Universität Kapstadt) - darunter Anja de Lange und Hayley Tomes als Erstautoren - über Untersuchungen zu den Auswirkungen der Larven auf das Gehirn [1]. Die Ergebnisse haben direkte Relevanz für das Verständnis der Pathogenese akuter Anfälle (Iktogenese) und - da Anfälle zu Anfällen führen - auch chronischer Epilepsie.
Die Larven sezernieren Glutamat
Die wichtigste Erkenntnis der neuen Arbeit ist, dass die Larven und ihre Ausscheidungen einen Neurotransmitter namens Glutamat enthalten, wobei der Glutamatgehalt hoch genug ist, um die umliegenden Neuronen direkt zu aktivieren. Um dies nachzuweisen, haben die Forscher zunächst Taenia solium-Larven homogenisiert und ihre Ausscheidungsprodukte gesammelt. Anschließend haben sie Neuronen diesen Produkten ausgesetzt und gezeigt, dass die Neuronen durch diese Exposition zum Feuern von Aktionspotenzialen aktiviert wurden. Wurden aber die Glutamatrezeptoren in den Neuronen vor der Exposition mit den Larvenprodukten blockiert, so fand keine Aktivierung der Neuronen statt.
Die Forscher untersuchten dann mit Hilfe von fluoreszierenden Kalzium-Indikatoren (Calcium-Imaging), wie sich die lokale Aktivierung von Neuronen durch das Glutamat aus den Larven auf lokale Gehirnschaltkreise auswirkt. Der Anstieg des intrazellulären Kalziums steht stellvertretend für die neuronale Aktivität, so dass sich mit Hilfe der Kalzium-Bildgebung die neuronale Aktivität in den Gehirnschaltkreisen sichtbar machen lässt. Diese bildgebenden Experimente bestätigten, dass das von den Larven ausgeschiedene Glutamat eine lokale neuronale Aktivierung auslöste, die zu einer anschließenden Aktivierung synaptisch verbundener Neuronen in entfernteren Schaltstellen führte. Die Forscher haben auch andere, von den Larven abgesonderte Produkte untersucht, die möglicherweise das Feuern von Neuronen beeinflussen könnten (wie Substanz P, Acetylcholin und Kalium), aber keines dieser Produkte hatte die gleiche weitreichende Wirkung wie Glutamat. Die Ergebnisse deuten also darauf hin, dass Glutamat aus den Larven eine potenzielle Rolle bei der Entstehung und/oder Ausbreitung von Krampfanfällen spielt.
Als Nächstes zeigten de Lange et al., dass die Larvenprodukte ähnlich erregende Wirkungen in-vitro auf Gehirngewebe von Tiermodellen und auf reseziertes menschliches Gehirngewebe haben. Sie wiesen auch nach, dass Glutamat umgebendes Hirngewebe erregen kann - in anderen Studien war dies als Treiber für spätere Epilepsie nachgewiesen worden. Darüber hinaus war von anderen Teams bereits gezeigt worden, dass von Hirntumoren freigesetztes Glutamat epileptische Anfälle auslösen kann; Forscher suchen auch nach Möglichkeiten, die Glutamatfreisetzung gezielt zu steuern, um solche Anfälle zu verhindern.
Ähnliche Strategien könnten bei der Bekämpfung der Neurozystizerkose von Vorteil sein, um Anfälle zu reduzieren oder möglicherweise sogar die Entwicklung einer chronischen Epilepsie zu verhindern. Dies ist von entscheidender Bedeutung, da Millionen von Neurozystizerkose-Patienten unter Krampfanfällen und Epilepsie leiden. Allerdings ist die neue Studie noch kein strikter Beweis, dass Larvenprodukte Anfälle oder Epilepsie verursachen, da sie auf der Ebene einzelner Zellen und kleiner Schaltkreise mit Mini-Mengen (Pikoliter) von Larvenprodukten durchgeführt wurde: bisher wissen wir nur, wie Neurozystizerkose zu neuronaler Übererregbarkeit führt. Abbildung 3. fasst die Ergebnisse der Studie [1] zusammen.
| Abbildung 3. . Neurozystizerkose und Epilepsie. Taenia Solium ist ein Parasit, der das Gehirn infizieren und eine Neurozystizerkose verursachen kann, die eine häufige, aber schlecht verstandene Ursache für erworbene Epilepsie ist. de Lange et al. [1] haben gezeigt, dass Taenia Solium erhebliche Mengen eines Neurotransmitters namens Glutamat enthält, der Neuronen und Schaltkreise von Neuronen aktivieren kann. |
Künftige Studien werden erforderlich sein, um die Auswirkungen der Larvenprodukte und der Larven selbst auf größere Schaltkreise in vivo zu verstehen und um zu untersuchen, ob/wie diese pathologische Erregung zu langfristigen Veränderungen im Gehirn führt, die spontane, wiederkehrende Anfälle auslösen. Auch nicht-glutamaterge Mechanismen, einschließlich neuroimmunologischer Prozesse, könnten für diese Veränderungen von Bedeutung sein.
Mit den aufregenden experimentellen Hinweisen und darüber hinaus mit naturnahen krankheitsrelevanten Modellen, welche die Untersuchung neuer Behandlungsansätze ermöglichen werden, bringt uns die neue Studie einem Verstehen von Epilepsie als Folge menschlicher Neurozystizerkose näher. Die Studie ist auch deshalb wichtig, weil der Neurozystizerkose - einer Krankheit, von der unverhältnismäßig viele Menschen in einkommensschwachen und unterversorgten Ländern betroffen sind - in der Vergangenheit nicht genügend Aufmerksamkeit geschenkt wurde. Die aktuellen Ergebnisse aus Kapstadt könnten die dringend benötigte Forschung auf dem Gebiet der Neurozystizerkose in Gang gebracht haben, da sie zeigen, was Taenia solium-Larven mit den sie umgebenden neuronalen Netzwerken anstellen. Es ist Aufregend!
[1] Anja de Lange, Hayley Tomes et al., 2023 Cestode larvae excite host neuronal circuits via glutamatergic signaling. eLife12:RP88174 https://doi.org/10.7554/eLife.88174.1
* Eine Zusammenfassung des Artikels von Anja de Lange, Hayley Tomes, et al., 2023, [1] verfasst von Zin-Juan Klaft und Chris Dulla ist am 23.08.2023 unter dem Titel "Epilepsy: How parasitic larvae affect the brain " im eLife-Magazin erschienen: https://doi.org/10.7554/eLife.91149. Der Text wurde von der Redaktion ins Deutsche übersetzt, mit einigen Textstellen und Abbildungen 1 und 2 ergänzt und ohne die im Originaltext zitierten Literatustellen wiedergegeben. eLife ist ein open access Journal, alle Inhalte stehen unter einer cc-by Lizenz.
Weiterführende Links
WHO: Taeniasis/cysticercosis (2022): https://www.who.int/news-room/fact-sheets/detail/taeniasis-cysticercosis
JJ Medicine: Pork Tapeworm (Taeniasis) | How It Infects, Symptoms & Cysticercosis, Diagnosis, Treatment (2022). Video 16:44 min. https://www.youtube.com/watch?v=z01Wu0x0us4
Parasitäre Erkrankungen im ScienceBlog:
- Gottfried Schatz, 26.07.2012: Unheimliche Gäste — Können Parasiten unsere Persönlichkeit verändern?
- Redaktion; 21.08.2015: Paul Ehrlich – Vater der Chemotherapie
- Inge Schuster, 26.06.2022: Getrübtes Badevergnügen - Zerkarien-Dermatitis
- Bill and Melinda Gates Foundation, 02.05.2014: Der Kampf gegen Malaria
- Elena Levashina, 16.05.2019: Zum Einsatz genetisch veränderter Moskitos gegen Malaria
- Redaktion, 09.10.2015: Naturstoffe, die unsere Welt verändert haben – Nobelpreis 2015 für Medizin
Live-Videos aus dem Körper mit Echtzeit-MRT
Live-Videos aus dem Körper mit Echtzeit-MRTDo, 24.08.2023 — Andreas Merian
Die Magnetresonanztomographie, kurz MRT, gehört längst zum medizinischen Alltag: Nach Sportverletzungen oder Unfällen, auf der Suche nach Tumoren oder zur Untersuchung des Gehirns werden MRT- produzierte Aufnahmen genutzt. Bisher musste man sich dabei allerdings mit Standbildern zufriedengeben. Doch der Arbeitsgruppe von Jens Frahm am Max-Planck-Institut für Multidisziplinäre Naturwissenschaften (Göttingen) gelingt es, mit der MRT Videos aufzunehmen. Die Aufnahmen in Echtzeit ermöglichen der Medizin neue Einblicke, zum Beispiel in das schlagende Herz, in Gelenke in Bewegung oder in die komplexen Vorgänge beim Singen, Sprechen oder Schlucken. Der Spektroskopiker Dr. Andreas Merian gibt einen Überblick über diese Verfahren.*
Nach einem schweren Sportunfall landet man oftmals in der Röhre. Abbildung 1. Während man in der Enge liegt, kann man dem Magnetresonanztomographen bei der Arbeit zuhören: es brummt und klackt und summt.
|
Abbildung 1: Magnetresonanztomographie. © J. Frahm , Investigative Radiology, Vol. 54 , Nr. 12 , 2019; istockphoto.com |
Nach der Untersuchung sichtet ein Arzt oder eine Ärztin die hochaufgelösten Schwarzweißbilder, auf denen die unterschiedlichen Gewebe klar zu unterscheiden sind und ihre Struktur gut zu erkennen ist. So wird festgestellt, ob durch den Unfall Bänder oder Sehnen in Mitleidenschaft gezogen wurden. Neben der Orthopädie kommt das Bildgebungsverfahren auch in vielen anderen Bereichen der Medizin zum Einsatz, zum Beispiel auf der Suche nach Tumoren oder bei der Untersuchung des Gehirns.
Doch was passiert eigentlich, während man in der Röhre liegt? Wie entstehen die Bilder und wie unterscheidet sich das Verfahren von Röntgen und Ultraschall? Der Physiker Jens Frahm vom Max-Planck-Institut für Multidisziplinäre Naturwissenschaften (Göttingen) war bei der Entwicklung der Magnetresonanztomographie von Anfang an dabei und sorgte dafür, dass sie die nötige Geschwindigkeit für den klinischen Alltag erreichte.
Rotierende Kerne
|
Abbildung 2: Kernresonanz. Kernspin und damit kernmagnetischer Dipol (oben) der Wasserstoffatomkerne des Körpers sind ohne äußeres Magnetfeld ungeordnet (unten links). Wird ein starkes statisches Magnetfeld angelegt, so richten sich die kernmagnetischen Dipole entlang der Feldlinien aus (unten rechts).© HNBM, MPG/CC BY-NC-SA 4.0 |
Der grundlegende physikalische Effekt hinter der MRT ist die magnetische Kernresonanz. Über 60 Prozent der Atome im menschlichen Körper sind Wasserstoffatome. Und deren Kerne haben einen Eigendrehimpuls, auch Kernspin genannt. Den Kern mit Eigendrehimpuls kann man sich wie einen Ball vorstellen, der sich um sich selbst dreht. Da die Wasserstoffkerne Protonen und damit durch den Kernspin bewegte Ladungen sind, erzeugen sie ein Magnetfeld. Der entstehende kernmagnetische Dipol richtet sich in einem von außen angelegten statischen Magnetfeld entlang der Feldlinien aus (Abbildung 2).
In einem MRT-Gerät wird durch supraleitende Spulen ein üblicherweise 1 bis 3 Tesla starkes statisches Magnetfeld erzeugt, das die Wasserstoffkerne im Körper der untersuchten Person ausrichtet. Dadurch entsteht eine makroskopische Magnetisierung entlang der Längsachse der Röhre (Abbildung 3). Ein elektromagnetisches Wechselfeld kann die Magnetisierung aus dieser Richtung auslenken. Damit das Wechselfeld die Magnetisierung kippen kann, muss seine Frequenz der Resonanzfrequenz entsprechen. Diese ist vom Atomkern und der Stärke des angelegten statischen Magnetfelds abhängig. Für Protonen bei 1 Tesla beträgt sie 42,58 MHz und liegt im UKW-Radiowellenbereich. Nach der Kippung kehrt die Magnetisierung langsam wieder in die Ausgangslage zurück. Dabei erzeugt sie ein elektromagnetisches Wechselfeld, das eine Spannung in einer Messspule induziert und so aufgezeichnet wird. Die Stärke des Signals weist auf die Protonendichte im Gewebe hin, während die Zeit bis zum Abklingen des Signals charakteristisch für die chemische Bindung der Wasserstoffatome und deren molekulare Umgebung ist.
Vom Signal zum Bild
Im Magnetresonanztomographen regt man also die magnetische Kernresonanz der Wasserstoffatome im Gewebe an, um Signale und damit Informationen aus dem Körper zu erhalten. Doch wie kann das Signal einem exakten Ort zugeordnet werden und wie setzt sich aus einer Vielzahl derartiger Informationen ein Bild zusammen?
Hier kommt der zweite Teil des Namens der Bildgebungsmethode ins Spiel, die Tomographie, was Schnittbild oder Schichtaufnahme bedeutet. Bei der MRT wird somit eine ausgewählte Schicht des Körpers dargestellt. Die sogenannte Ortskodierung ermöglicht es, aus den Messdaten Abbilder des Untersuchungsobjekts zu berechnen. Dazu werden zusätzlich zum statischen magnetischen Feld weitere Magnetfelder angeschaltet, die sich in ihrer Stärke entlang einer Achse unterscheiden. Man spricht von Gradientenfeldern (Abbildung 3). In der MRT werden klassisch drei senkrechte Gradientenfelder verwendet, die es möglich machen, ein Signal genau seinem Ursprungsort zuzuordnen. Der erste Gradient wählt die Schicht aus, die abgebildet werden soll, und die beiden anderen Gradienten erzeugen ein Gitter, wodurch Signale Bildpunkten zugeordnet werden können (Abbildung 3). Dabei macht man sich zunutze, dass ein zusätzliches Magnetfeld die Resonanzfrequenz der Wasserstoffkerne ändert und man so eine Schicht zur Anregung auswählen bzw. anschließend nachvollziehen kann, von welchem Ort ein Signal stammt. Die exakte Schaltung von Gradienten- und Wechselfeldern hängt vom spezifischen Verfahren ab und ist hochkomplex. Doch allgemein gilt: Um ein vollständiges Schichtbild zu erhalten, müssen die Gradientenfelder so oft an und wieder ausgeschaltet werden, wie das Bild letztlich Zeilen bzw. Bildpunkte in einer Dimension haben soll. Meist werden in der MRT Bilder mit bis zu 512 x 512 Bildpunkten aufgenommen. Damit die Aufnahme möglichst schnell abläuft, werden die zur Ortskodierung eingesetzten Gradientenfelder sehr schnell geschaltet, wodurch die lauten Geräusche in der Röhre entstehen.
|
Abbildung 3: Schema des Magnetresonanztomographs. Die zu untersuchende Person wird in der Röhre positioniert. Die äußersten Spulen (rot) erzeugen das starke statische Magnetfeld, das die Kernspins der Wasserstoffkerne ausrichtet. Die Spulen für die Gradientenfelder (grün) schalten kurzzeitig weitere (statische) Magnetfelder für die Ortskodierung zu. Radiofrequenzspulen (orange) erzeugen das magnetische Wechselfeld zur Auslenkung der Magnetisierung und dienen als Empfängerspulen für die Signale. © HNBM, MPG / CC BY-NC-SA 4.0 |
Mittels komplexer mathematischer Verfahren lässt sich dann aus der großen Datenmenge der vielen Einzelmessungen ein zweidimensionales Schnittbild berechnen. Bis Mitte der 1980er Jahre dauerte die Aufnahme eines Schnittbildes ca. 5 Minuten, eine dreidimensionale Messung des Körpers mit beispielsweise 256 x 256 x 256 Bildpunkten sogar mehrere Stunden. Da sich während dieser Zeit der Patient auch nicht bewegen durfte, führte das dazu, dass die MRT im klinischen Alltag selten eingesetzt wurde.
Doch 1985 gelang Jens Frahm und seinem Team ein Durchbruch. „Durch FLASH eliminierten wir die Wartezeit zwischen den Einzelmessungen und beschleunigten die MRT so um einen Faktor größer 100. Plötzlich konnten einzelne Schichtbilder in Sekundenschnelle aufgenommen werden und dreidimensionale Aufnahmen dauerten nur noch wenige Minuten“, sagt Jens Frahm. Das patentierte FLASH-Verfahren wurde innerhalb eines halben Jahres von allen Herstellern von MRT-Geräten übernommen und kommt heute in allen kommerziellen Geräten zum Einsatz. Dank der Geschwindigkeit des neuen Verfahrens etablierte sich die MRT in der diagnostischen Bildgebung.
MRT vs. Röntgen und Ultraschall
Doch warum ist die MRT in der Medizin eigentlich so gefragt? Mit Röntgen, Computertomographie (CT) und Sonographie waren ja bereits verschiedene Bildgebungsverfahren etabliert. Welche Vorteile bietet die MRT gegenüber diesen Methoden?
Beim Röntgen nutzt man die kurzwellige elektromagnetische Röntgenstrahlung. Diese wird von der einen Seite auf die zu untersuchende Körperpartie gestrahlt und auf der anderen Seite detektiert. Je mehr Gewebe zwischen Strahlungsquelle und Detektor liegt und je dichter dieses Gewebe ist, desto mehr Röntgenstrahlung wird absorbiert oder gestreut. Dichte anatomische Strukturen wie Knochen heben sich somit hell gegen die dunkleren Weichteile wie Muskeln ab. Knochenbrüche können so zum Beispiel leicht diagnostiziert werden. Unterschiedliche weiche Gewebe lassen sich durchs Röntgen nur schwer unterscheiden, da hierzu der Kontrast nicht ausreichend ist. Die Computertomographie basiert auch auf Röntgenstrahlung, ermöglicht aber statt einer Durchleuchtung eine Schichtbildgebung und 3D-Aufnahmen. Da die kurzwellige Röntgenstrahlung ionisierend auf biologisches Gewebe wirkt, besteht durch die Strahlenbelastung ein erhöhtes Krebsrisiko. Deshalb wird bei einer Untersuchung nur die betreffende Stelle geröntgt und empfindliche Körperpartien werden durch eine Bleischürze geschützt. Die Strahlenbelastung verbietet es außerdem, Videos mit vielen Bildern pro Sekunde mit Röntgenapparat oder CT aufzunehmen.
Bei der Sonographie werden mechanische Ultraschallwellen in den Körper gesandt und deren Echo aufgezeichnet. Mit der Sonde, die den Ultraschall aussendet und detektiert, wird über die betreffenden Körperstellen gefahren. Aus der Laufzeit und der Amplitude des Echos berechnet ein Computer dann in Echtzeit Bilder. Von der Sonde werden die Ultraschallwellen fächerartig in den Körper ausgesandt. So entsteht typischerweise ein Schnittbild entlang der Ebene dieses Fächers. Moderne Geräte ermöglichen aber auch 3D-Bilder. Je stärker ein Gewebe den Schall zurückwirft, desto heller erscheint es auf den Bildern. Dadurch entsteht der Kontrast im Bild. Da die Eindringtiefe des Ultraschalls begrenzt ist, werden tief liegende oder verdeckte anatomische Strukturen kaum oder nicht aufgelöst. Dies schränkt die Nutzung der Sonographie ein. Je nach medizinischer Fragestellung können Ärztinnen und Ärzte einzelne Bilder aufnehmen oder in Echtzeit mit Videobildrate das Geschehen im Körper verfolgen. Die sonographische Untersuchung hängt in ihrer Qualität stark vom Untersuchenden ab und ist schlecht wiederholbar. Vorteil der Sonographie ist, dass sie nichtinvasiv und risikoarm ist, weshalb sie beispielsweise in der Schwangerschaftsvorsorge eingesetzt wird.
Stärken der MRT sind der hervorragende Weichteilkontrast und die hohe räumliche Auflösung. So entstehen scharfe Bilder des gesamten Körperinneren. Außerdem sind sowohl die Radiowellen als auch die statischen Magnetfelder gesundheitlich unbedenklich. Ganzkörperscans oder wiederholte Untersuchungen bedeuten also kein zusätzliches gesundheitliches Risiko. Seit der Einführung von FLASH hat sich die Geschwindigkeit der MRT lange Zeit nicht verändert. Das bedeutete, dass zwar MRT-Aufnahmen im medizinischen Alltag kein Problem, aber MRT-Videos bewegter Vorgänge aus dem Körperinneren ein Ding der Unmöglichkeit waren: An Videos mit Bildraten von 20 Bildern pro Sekunde oder mehr war nicht zu denken. „Nach der Entwicklung von FLASH sahen wir zunächst keine weitere Möglichkeit, die MRT zu beschleunigen“, sagt Jens Frahm. Doch die Idee einer noch schnelleren MRT spukte ihm weiter im Kopf herum.
Echtzeit durch Hochleistungsrechner
Bis es soweit war, dauerte es 25 Jahre. Aber seit dem Durchbruch, den Jens Frahm mit seinem Team 2010 feierte, kann er sagen: „Wir haben es geschafft, die MRT-Bildgebung weiter zu beschleunigen! So ist uns sozusagen der Schritt vom Foto zum Film gelungen. Und das eröffnet ganz neue diagnostische Möglichkeiten, zum Beispiel weil das schlagende Herz genau dargestellt werden kann.“ Im Vergleich zur MRT vor 1985 gelang es Jens Frahm und seinem Team, die Aufnahmegeschwindigkeit insgesamt um einen Faktor 10.000 zu steigern. Nun können die Forschenden Schichtbilder mit einer Bildrate von bis zu 100 Bildern pro Sekunde aufnehmen. Sie nennen ihre Technik Echtzeit-MRT. „Möglich gemacht haben diese Entwicklung zum einen Fortschritte in der numerischen Mathematik und zum anderen die Verfügbarkeit von leistungsstarken Grafikkartenrechnern“, erläutert Jens Frahm. Denn die Beschleunigung des Verfahrens beruht nicht wie bei FLASH darauf, dass die Einzelmessungen schneller werden. „Wir messen einfach weniger oft und nutzen dann ein neues mathematisches Verfahren, um aus den für eine klassische Berechnung ungenügenden Daten ein aussagekräftiges Bild zu erzeugen.“ Dazu wenden die Forschenden die radiale Ortskodierung an (Abbildung 4).
|
Abbildung 4: Echtzeit-MRT durch radiale Ortskodierung: Anstatt wie herkömmlich durch die Gradientenfelder ein Gitter zu erzeugen, laufen die verwendeten radialen Gradienten wie Speichen durch die Mitte eines Rades (links). Dabei werden für jedes Schichtbild aber weniger Einzelmessungen als nötig aufgenommen. Die fehlenden Informationen werden anschließend durch die Lösung eines komplexen mathematischen Problems ermittelt. So lassen sich z.B. Videos des Schluckvorgangs mit einer Zeitauflösung von nur 40 ms aufnehmen. Auf den Einzelbildern aus dem Video hebt sich die getrunkene Flüssigkeit weiß ab (rechts). © links: HNBM, MPG / CC BY-NC-SA 4.0; rechts © J. Frahm et al.: Real-Time Magnetic Resonance Imaging. Investigative Radiology, Vol. 54, Nr. 12, 2019 |
Entscheidend ist, dass für jedes Schichtbild je nach Anwendung um einen Faktor 10 bis 40 weniger Einzelmessungen durchgeführt werden als eigentlich nötig. Dadurch wird die Aufnahme genau um diesen Faktor schneller. Die fehlenden Informationen werden anschließend durch die Lösung des nichtlinearen inversen Problems rekonstruiert. Durch dieses mathematische Verfahren wird das Bild nicht direkt aus den Daten rekonstruiert, sondern ausgehend von einem Startbild – üblicherweise dem letzten aufgenommenen Bild – geschätzt. Aus dem geschätzten Bild lassen sich die Daten berechnen, die die Messspulen aufgenommen haben müssten, um dieses zu erzeugen. Und die kann man wiederum mit den tatsächlich aufgenommenen Daten abgleichen. In einem schrittweisen Prozess kann das geschätzte Bild so optimiert werden, dass es möglichst genau zu den Messwerten passt. Das klappt dank der Entwicklungen von Jens Frahms Team so gut, dass die Bildqualität ausreichend für die medizinische Diagnostik ist. Da dieser Ablauf die eigentliche Bildentstehung in der MRT auf den Kopf stellt, spricht man von einem inversen Problem. Die Lösung dieses Problems und damit die Echtzeit-MRT erfordert eine sehr große Rechenleistung. Was sie allerdings nicht benötigt, ist ein besonderes MRT-Gerät. So könnten alle bereits in Kliniken vorhandenen Geräte durch einen leistungsfähigen Grafikkartenrechner für die Datenverarbeitung erweitert werden.
Live-Videos aus dem Körper
Der Echtzeit-MRT eröffnen sich viele Anwendungsfelder: So kann ein Kardiologe direkt das schlagende Herz beobachten und beispielsweise Herzrhythmusstörungen genau analysieren. Auch das Schlucken und Schluckbeschwerden können durch die Echtzeit-MRT erstmals untersucht werden. Neben klassischen medizinischen Anwendungen ist Jens Frahm auch auf großes Interesse in der Musik und Phonetik gestoßen: Wie genau werden beim Sprechen Töne erzeugt? Wie beim Beatboxen? Und was unterscheidet einen herausragenden Hornisten von einem Anfänger oder Fortgeschrittenen? Durch die neuen Möglichkeiten der Echtzeit-MRT können beispielsweise die Bewegungen der Zunge im Mundraum genau untersucht und quantifiziert werden.
|
Abbildung 5: Messen in Millisekunden: Schichtbild aus dem Gehirn eines 4 Jahre alten Kindes. Links: Die herkömmliche MRT mit einer Messzeit von 38 s liefert ein verschwommenes Bild, da sich das Kind offensichtlich während der Messung bewegt hat. Rechts: Die Echtzeit-MRT mit einer Messzeit von 50 ms erzeugt ein scharfes Bild, das zur Diagnostik genutzt werden kann. © Verändert nach: D. Gräfe et al.: Outpacing movement — ultrafast volume coverage in neuropediatric magnetic resonance imaging. Pediatr Radiol 50, 2020. / CC BY 4.0 |
Die neue Methode beschleunigt auch die Untersuchung ganzer Körperteile mittels überlappender Schichtbilder, die nun in nur wenigen Sekunden gemessen werden (Abbildung 5). Dies ist gerade in der Kinderheilkunde ein großer Vorteil. Denn Säuglinge und Kleinkinder halten nicht lange genug still, um mit der konventionellen MRT beispielsweise eine vollständige Bildgebung des Schädels durchzuführen. Daher ist bisher oft eine risikobehaftete Narkose notwendig. „Unsere Kooperationspartner an der Universitätsklinik Leipzig haben in den letzten Jahren festgestellt, dass mit der Echtzeit-MRT in mindestens der Hälfte aller Fälle keine Narkose notwendig ist“, sagt Jens Frahm.
Der Forscher ist an seinem Ziel angekommen: Live-Videos aus dem Körper dank MRT. Und seit dem Durchbruch 2010 zeigte sich, dass die beschleunigte Methode zahlreiche neue Anwendungen ermöglicht. „Wir müssen aber erst lernen, die Echtzeit-MRT diagnostisch zu nutzen. Auch für das medizinische Personal ergeben sich neue Anforderungen und notwendige Erprobungsphasen. Die technischen Fortschritte müssen in belastbare Untersuchungsprotokolle ‚übersetzt’ werden, die die jeweiligen medizinischen Fragestellungen optimal beantworten.“
* Der Artikel von Andreas Merian ist unter dem Titel: "Liveschaltung in den Körper - Neue Einblicke mit der Echtzeit-MRT" https://www.max-wissen.de/max-hefte/techmax-33-echtzeit-mrt/ im Techmax33-Heft der Max-Planck-Gesellschaft im Frühjahr 2023 erschienen. Mit Ausnahme des Titels wurde der unter einer cc-by-nc-sa Lizenz stehende Artikel unverändert in den Blog übernommen.
Weiterführende Links
Echtzeit-MRT-Film: Sprechen Video 2:16 min (2018) https://www.youtube.com/watch?v=6dAEE7FYQfc Copyright: Jens Frahm / Max-Planck-Institut für biophysikalische Chemie
Jens Frahm - Schnellere MRT in Echtzeit: Video 6:05 min. (2018) https://www.youtube.com/watch?v=lYQXsMWfJT4
Roland Wengenmayr: Liveschaltung zum Krankheitsherd. (2017) https://www.mpg.de/11248354/W003_Material_Technik_054-061.pdf
Seetang - eine noch wenig genutzte Ressource mit hohem Verwertungspotential - soll in Europa verstärkt produziert werden
Seetang - eine noch wenig genutzte Ressource mit hohem Verwertungspotential - soll in Europa verstärkt produziert werdenDo. 17.08.2023 — Redaktion

![]() Als wertvolle und noch wenig genutzte Ressource aus den Ozeanen und Meeren bieten Algen ein weites Spektrum an gesundheitlichen und ökologischen Vorteilen. Auf Grund ihres hohen Verwertungspotenzials wachsen Nachfrage und Markt für Seetang, "Meersalat" und andere Algen. Um deren Produktion in Europa zu steigern, hat die Europäische Kommission 2022 einen Aktionsplan herausgegeben, in dem intensivierte Forschung zur Optimierung der Algenarten und Züchtungstechnologien eine zentrale Rolle spielen. Der vorliegende Artikel gibt einen Überblick über die Forschung, die von der EU finanziert wird.*
Als wertvolle und noch wenig genutzte Ressource aus den Ozeanen und Meeren bieten Algen ein weites Spektrum an gesundheitlichen und ökologischen Vorteilen. Auf Grund ihres hohen Verwertungspotenzials wachsen Nachfrage und Markt für Seetang, "Meersalat" und andere Algen. Um deren Produktion in Europa zu steigern, hat die Europäische Kommission 2022 einen Aktionsplan herausgegeben, in dem intensivierte Forschung zur Optimierung der Algenarten und Züchtungstechnologien eine zentrale Rolle spielen. Der vorliegende Artikel gibt einen Überblick über die Forschung, die von der EU finanziert wird.*
Wenn auch die Vorstellung noch nicht verlockend ist, dass man fermentierten Seetang den Mahlzeiten daheim zufügt, so wird sich das bald ändern, glaubt Ólavur Gregersen. Seit zehn Jahren experimentieren Gregersen und seine Familie auf den Färöer-Inseln mit allen Arten einer durch Algen inspirierten Küche. Die Ergebnisse reichen von Algenbutter bis zu seinem neuen Lieblingsgericht: Algenpesto.
Tägliche Dosis
"Wir achten wir darauf, dass wir täglich einen Esslöffel Algen zu einer unserer Mahlzeiten essen", so Gregersen. Ob zum Frühstück über das Joghurt gestreut, über den Salat zum Mittagessen oder über Fleisch und Fisch am Abend - er ist überzeugt, dass Algen nicht nur gut schmecken, sondern auch von großem Nutzen sowohl für die Gesundheit als auch die Umwelt sind.
Was die menschliche Gesundheit betrifft, so ist Seetang vollgepackt mit essenziellen Nährstoffen wie Eiweiß und Omega-3-Fettsäuren. Die Europäische Behörde für Lebensmittelsicherheit hat 2019 auf Seetang als ein "Superfood" hingewiesen und dessen hohen Nährwert angeführt.
| Abbildung 1. Wegen des Nutzens für Gesundheit und Umwelt will Europa die Produktion von Meeresalgen ausweiten. Bildnachweis: Bjoertvedt, CC BY-SA 4.0 |
Auch aus ökologischer Sicht bieten die Algen viele Vorteile: Sie "saugen" CO2 auf und benötigen weder Zusatzstoffe noch Dünger oder Frischwasser. Einem Bericht der Vereinten Nationen aus dem Jahr 2023 zufolge hat die Algenzucht das Potenzial, "viele" Vorteile für die Umwelt zu bieten, darunter auch für den Kampf gegen den Klimawandel. In Kenntnis des gesamten Potentials hat die Europäische Kommission 2022 einen Aktionsplan zur Steigerung der Produktion von Meeralgen in Europa herausgegeben [1]. Die Intensivierung der Forschung ist ein zentraler Bestandteil der Strategie, ebenso wie eine verbesserte Gesetzgebung, eine gezielte Unternehmensförderung und eine stärkere öffentliche Wahrnehmung. Abbildung 1.
Meeralgen können im offenen Meer oder in Tanks an Land gezüchtet werden. Die EU gehört derzeit zu den weltweit größten Importeuren von Meeresalgen, die aus China, Südkorea und Chile zur Nutzung als Lebensmittel und für industrielle Zwecke (beispielsweise Düngemittel) geliefert werden.
Im Jahr 2021 belief sich der Weltmarkt für Meeralgen auf fast 14 Milliarden Euro und er soll nach Angaben der niederländischen Regierung bis 2028 voraussichtlich auf mehr als 22 Milliarden Euro anwachsen.
Da die Nachfrage in Europa von 270 000 Tonnen im Jahr 2019 laut EU auf 8 Mio. Tonnen bis 2030 steigen dürfte, könnte der europäische Markt für Meeralgen im Jahr 2030 einen Wert von 9 Mrd. EUR erreichen. Durch die steigende Produktion in Europa könnten rund 85 000 Arbeitsplätze geschaffen werden.
Lebensmittel, Futtermittel und mehr
Gregersen ist Unternehmer und Mitbegründer des Unternehmens Ocean Rainforest, das seit 2010 auf den Färöer Inseln Pionierarbeit bei der Züchtung von Meeresalgen vor der Küste leistet. Er koordiniert auch ein europäisches, mit EU-Mitteln gefördertes Forschungsprojekt, um neue Produkte auf Algenbasis zu entwickeln. Die vierjährige Initiative mit dem Namen SeaMark läuft bis Juni 2026 [2].
Gregersen sieht in Europa ein großes ungenutztes Potenzial für Seetang, der in der asiatischen Küche jaseit Jahrhunderten ein Grundnahrungsmittel ist. Er möchte beweisen, dass in Europa die Züchtung von Meeralgen gewinnbringend sein kann und für diese vielfältige Verwendungsmöglichkeiten bestehen. Im Rahmen des Projekts werden 12 Produkte entwickelt, die von Lebensmittelzutaten und Futtermittelzusatzstoffen bis hin zu Nahrungsergänzungsmitteln und Verpackungsmaterial reichen [2]. Konkrete Beispiele sind Algenfasern, die auf Grund ihrer wasserbindenden und gelbildenden Eigenschaften in Lebensmitteln Anwendung finden, Beta-Glucane für die Hautpflege und Verbindungen für pelletierte Bio-Verpackungsmaterialien.
"Mit Seamark wollen wir zeigen, dass es möglich ist, den Prozess der Algenzüchtung effizienter zu gestalten und dass es in Europa einen großen Markt für diese Produkte gibt", so Gregersen.
Ein Teil des Projekts befasst sich mit der Weiterentwicklung der milchsauren Vergärung (Laktofermentation) von Meeresalgen, damit diese in Tierfutter verwendet werden können. Erste Ergebnisse an mit Algen gefütterten Schweinen haben gezeigt, dass diese infolge verbesserter Mikroflora im Darm mehr Milch produzierten und weniger Futter benötigten. Dies wird nun mit 500 Mutterschweinen in einem Betrieb in Dänemark untersucht.
Wachstumspotenzial
Was die Züchtung betrifft, so ist die für SeaMark besonders interessante Algenart eine Braunalge, die als Zuckertang bekannt ist, wie Lasagne-Nudeln aussieht und einen süßen Geschmack hat. Sie ist auch unter dem Namen Saccharina latissima bekannt und wächst in der Natur an den Küsten Nordeuropas und im Süden bis nach Galicien in Spanien. Zuckertang lässt sich schnell züchten und ist daher ideal für die Produktion, solange die Kosten niedrig gehalten werden können. Abbildung 2.
| Abbildung 2. Der Forscher Mike Murphy hält eine Saccharina latissima (Zuckertang) hoch. Bild: Wikipedia, gemeinfrei |
Eine eng verwandte Art ist in der japanischen Küche bekannt und wird in Japan als "Kombu" geerntet und verkauft.
Auf den Färöer Inseln, wo Temperatur, Nährstoffe und Licht optimal für das Wachstum von Algen sind, hat das SeaMark-Team neue Geräte getestet, die im Meer die Anzucht der Algen beschleunigen - das Äquivalent zur Aussaat von Samenreihen auf einem Acker. Unabhängig davon werden im Rahmen des Projekts Maschinen getestet, die die Ernte beschleunigen, sobald die Biomasse bereit steht. Die Geräte können laut Gregersen zeitaufwändige Handarbeit ersetzen, die Produktionskosten senken und den Weg für eine höhere Produktion ebnen.
Ocean Rainforest weitet seine Aktivitäten mit Versuchen zum Offshore-Anbau in Norwegen und zum Anbau von Meeresalgen an Land in Island aus.
Seetang-Stämme
An der Westküste Irlands stützt sich SeaMark auf das Fachwissen des Algenspezialisten Dr. Ronan Sulpice von der Universität von Galway. Sulpice hilft bei der Identifizierung der besten Algenstämme für die Züchtung von Zuckertang und der als Meersalat bekannten Ulva, die neben ihrem Nährwert auch Stoffe enthält, die zur Herstellung von Arzneimitteln extrahiert werden können. Ziel ist es, hohe Erträge und optimale Qualität - insbesondere hinsichtlich des Proteingehalts - zu gewährleisten.
Sulpice leitet auch ein separates, von der EU finanziertes Forschungsprojekt, um Algenstämmen zu identifizieren, die sich ideal für die Aquakultur eignen. Das Projekt mit der Bezeichnung ASPIRE dauert bei einer Laufzeit von zwei Jahren bis September 2024 [3].
Um Saatgut für die Zucht zu finden, fahren Algenzüchter in der Regel ins Meer hinaus oder die Küste entlang, um Wildstämme zu sammeln. Diese Praxis bedeutet jedoch, dass die gezüchteten Algen von Jahr zu Jahr stark variieren können. Angesichts des zunehmenden Interesses an diesen Pflanzen möchte Sulpice den Farmern helfen, die Auswahl der Sorten zu verbessern und die Produktion zu steigern.
"Die Art und Weise, wie wir derzeit in Europa Saatgut für Meeresalgen züchten, ähnelt der Art und Weise, wie wir vor 10 000 Jahren Landpflanzen angebaut haben - dies ist noch sehr urtümlich, aber die Entwicklung schreitet fort", sagte er. ASPIRE bietet die Möglichkeit, Algensorten zu züchten, die nach den Wünschen der Erzeuger und Verbraucher ausgewählt wurden.
Portugal, Irland untersuchen
Sulpice hat bereits portugiesischen Züchtern, die Ulva anbauen, geholfen, indem er verschiedene Stämme, die an sein Labor in Irland geschickt wurden, untersucht hat. Die Ergebnisse haben erhebliche Unterschiede in den Wachstumsraten zwischen den besten und den schlechtesten Stämmen gezeigt; einige lieferten fünfmal mehr Biomasse. Dank dieser Erkenntnisse konnte ein Betrieb in Portugal seine Produktivität verdoppeln, so Sulpice.
Das Hauptaugenmerk von ASPIRE liegt jedoch auf einer rötlich-braunen Meeresalge namens Palmaria palmata. Sie hat einen hohen Proteingehalt, wächst im Atlantik von Schweden bis Portugal und schmeckt gebraten wie Speck. Die Nachfrage nach Palmaria palmata, auch bekannt als Lappentang, Dulse oder Dillisk, ist groß und die Ernte kann bis zu 250 € pro Kilogramm einbringen. Im Vergleich dazu kostete importierter Seetang in Teilen Europas etwa 6 € pro Kilogramm im Jahr 2020.
Sulpice ist überzeugt, dass ASPIRE den Algenunternehmen in Europa, einschließlich der irischen Unternehmen, helfen kann, dieses Potenzial zu nutzen. Ein solches Unternehmen ist Mungo Murphy's Seaweed, eine Meeresalgenfarm im Bezirk Connemara im Westen Irlands. Seit 2014 züchtet Mungo Murphy's Algen wie Ulva und Palmaria palmata zusammen mit Abalonen (Meeresschnecken) und Seegurken in Becken an Land an. Laut Cindy O'Brien, die das Unternehmen gegründet und die Farm aufgebaut hat, ist dies eine Form von Aquakultur, die in Irland zunehmend an Bedeutung gewinnt. Sie betreibt diese jetzt zusammen mit ihrer Tochter Sinead. Abbildung 3.
| Abbildung 3. Sinead O'Brien mit einer Handvoll Seetang. © Mungo Murphy’s Seaweed Co |
Attraktivität für den Verbraucher
Mungo Murphy's hofft, dass die Zusammenarbeit mit Sulpice die Möglichkeit bietet, die angebauten Produkte weiter zu entwickeln. "Diese Zusammenarbeit wird es uns ermöglichen, mehr Rohmaterial durch Aquakultur zu produzieren und nicht von wild geerntetem Seetang abhängig zu sein", sagt Cindy O'Brien.
Die auf der Farm gezüchteten Algen werden zu einer Reihe von Verbraucherprodukten verarbeitet. Dazu gehören Kosmetika wie Algen-gefüllte Badesäckchen und Gesichtsmasken und Lebensmittel wie Algengewürze für Suppen, Brote, Salate und sogar Popcorn. O'Brien sagt, dass die Menschen oft von der Vielseitigkeit der Algen überrascht sind. Mungo Murphy empfiehlt Köchen Algen in ihren Gerichten zu verwenden und hebt ihren Einsatz in Konsumgütern hervor.
ASPIRE ist nun in der Hälfte seiner Laufzeit; O'Brien rechnet damit, dass das Projekt dazu beitragen wird, die allgemeine Attraktivität von Algen zu erhöhen."Das wird uns helfen, mehr gesunde Produkte für den menschlichen Verzehr herzustellen", sagte sie.
[1] United Nations Environment Programme (2023). Seaweed Farming: Assessment on the Potential of Sustainable Upscaling for Climate, Communities and the Planet. Nairobi.https://wedocs.unep.org/bitstream/handle/20.500.11822/42642/seaweed_farming_climate.pdf?sequence=3&isAllowed=y
[2] SeaMark - Seaweed based market applications. https://cordis.europa.eu/project/id/101060379
[3] ASPIRE - Accelerated Seaweed Production for Innovative and Robust seaweed aquaculture in Europe.. https://cordis.europa.eu/project/id/101066815/de
*Dieser Artikel wurde ursprünglich am 9. August 2023 von Andrew Dunne in Horizon, the EU Research and Innovation Magazine unter dem Titel "From butter to baths, seaweed’s potential is being tapped in Europe" https://ec.europa.eu/research-and-innovation/en/horizon-magazine/butter-baths-seaweeds-potential-being-tapped-europe publiziert. Der unter einer cc-by-Lizenz stehende Artikel wurde von der Redaktion möglichst wortgetreu aus dem Englischen übersetzt. Abbildung 2 und Beschriftung wurden von der Redaktion eingefügt.
Über Braunalgen im ScienceBlog:
Susanne Coelho, 02.09.2021: Das Privatleben der Braunalgen: Ursprünge und Evolution einer vielzelligen, sexuellen Entwicklung
Und man erforscht nur die im Lichte, an denen im Dunkel forscht man nicht - Die Unknome Datenbank will auf unbekannte menschliche Gene aufmerksam machen
Und man erforscht nur die im Lichte, an denen im Dunkel forscht man nicht - Die Unknome Datenbank will auf unbekannte menschliche Gene aufmerksam machenFr, 11.08.2023 — Redaktion

![]() Das menschliche Genom kodiert für etwa 20 000 Protein. Viele dieser Proteine sind noch nicht charakterisiert und ihre Funktion ist unbekannt. Der Schwerpunkt der Forschung liegt auf bereits gut untersuchten Proteinen; dies gibt zur Besorgnis Anlass, dass in den vernachlässigten Teilen des Genoms/Proteoms wichtige biologische Prozesse kodiert sind. Um dem entgegenzuwirken, hat ein britisches Forscherteam eine "Unknome-Datenbank" entwickelt, in der die Proteine danach geordnet sind, wie wenig über sie bekannt ist. Die Datenbank soll die Auswahl bislang schlecht oder gar nicht charakterisierter Proteine von Menschen oder Modellorganismen erleichtern, damit sie gezielt untersucht werden können.
Das menschliche Genom kodiert für etwa 20 000 Protein. Viele dieser Proteine sind noch nicht charakterisiert und ihre Funktion ist unbekannt. Der Schwerpunkt der Forschung liegt auf bereits gut untersuchten Proteinen; dies gibt zur Besorgnis Anlass, dass in den vernachlässigten Teilen des Genoms/Proteoms wichtige biologische Prozesse kodiert sind. Um dem entgegenzuwirken, hat ein britisches Forscherteam eine "Unknome-Datenbank" entwickelt, in der die Proteine danach geordnet sind, wie wenig über sie bekannt ist. Die Datenbank soll die Auswahl bislang schlecht oder gar nicht charakterisierter Proteine von Menschen oder Modellorganismen erleichtern, damit sie gezielt untersucht werden können.
Das Human Genome Project
Mit der (nahezu) vollständigen Sequenzierung des menschlichen Genoms vor 20 Jahren ist zweifellos eine neue Ära der Biowissenschaften angebrochen. Tausende Wissenschafter, die rund um den Globus am Human Genome Project beteiligt waren, konnten einen lange angezweifelten Erfolg von "Big Science" feiern und eröffneten ein Eldorado an genetischen Informationen, das für jeden Interessierten frei zugänglich ist. Forscher wie Geldgeber (aus staatlichen Organisationen und Industrie) gingen davon aus, dass man nun schnell zu einem neuen Verständnis der Biologie des Menschen und seiner Krankheiten gelangen würde. Man erhoffte so in wenigen Jahren viele neue Zielstrukturen (Targets), die zumeist Proteine sind, für die Entwicklung erfolgversprechender Medikamente zu entdecken. Um den damaligen US-Präsidenten Bill Clinton anlässlich der Präsentation des ersten drafts des Human Genome Projects im Juni 2000 zu zitieren "werde das Projekt die Diagnose, Prävention und Therapie der meisten, wenn nicht aller Erkrankungen des Menschen revolutionieren “ und als eine wesentliche Aufgabe sah Clinton: "wir müssen die Fülle von Genomdaten durchforsten, um jedes menschliche Gen zu identifizieren. Wir müssen die Funktion dieser Gene und ihrer Proteinprodukte herausfinden, und dann müssen wir dieses Wissen schnell in Behandlungen umsetzen, die das Leben verlängern und bereichern können" [1].
20 Jahre später sind auch die letzten Lücken im menschlichen Genom geschlossen. Mit neuen Technologien, die einen Preissturz der Sequenzierungen herbeiführten und sie beinahe schon für jeden erschwinglich machten, wurden bislang die Genome von mehr als einer Million Menschen sequenziert. Abbildung 1 zeigt ein vereinfachtes Schema der Genomsequenzierung.
| Abbildung 1 Schema der Gensequenzierung. Francis S. Collins, berühmter Pionier der Genforschung und damals Direktor des National Human Genome Research Institute (NHGRI) der NIH war de facto Leiter des aus Tausenden Forschern bestehenden International Human Genome Sequencing Consortium im Humangenomprojekt. Das NHGRI spielte darin eine wichtige Rolle Viele der neuen Technologien wurden im Rahmen und mit Unterstützung des Genomtechnologie-Programms des NHGRI entwickelt. (Bild: https://www.genome.gov/about-genomics/fact-sheets/DNA-Sequencing-Fact-Sheet; cc 0) |
Es wurden bislang zahlreiche Gene identifiziert , die mit Krankheiten assoziiert sind; dies kann frühe Diagnosen ermöglichen, bevor noch klinische Symptome auftreten. Zu den wichtigsten Erkenntnissen der Genomanalyse zählen zweifellos die Variationen in unseren Genen, die jeden von uns zu einem unterschiedlichen Individuum machen und sich auch auf unseren gesundheitlichen Status und auf das Krankheitsrisiko auswirken. In Folge ist eine Personalisierte Medizin entstanden, die diese Informationen in gezielte individuelle Behandlungen umsetzen möchte. (Basierend auf dem Genom eines Patienten können beispielsweise die für ihn geeignetsten - d.i. wirksamsten, nebenwirkungsärmsten - Medikamente ermittelt werden.)
Viele Gene und Proteine liegen noch im Dunkeln
Die Kenntnis aller Gensequenzen hat allerdings nicht zu einem wesentlichen Anstieg der Entdeckungsrate neuer Genfunktionen geführt, diese ist seit 2000 sogar noch zurück gegangen [2]. Es gibt einen hohen Anteil an Genen, deren Funktion unzureichend oder noch gar nicht charakterisiert ist. Dies gilt auch für die Charakterisierung der Proteine. Insgesamt enthält das Humangenom den Bauplan (d.h. es kodiert) für etwa 20.000 Proteine, Tausende davon waren zuvor in biochemischen und/oder genetischen Studien noch nicht identifiziert worden. Für viele dieser neuen Proteine ist auch 20 Jahre nach ihrer Entdeckung die Funktion noch unbekannt.
Ein rezenter Überblick über die biomedizinische Literatur bis 2017 zeigt, dass von rund 2000 Proteinen praktisch noch keine Informationen vorliegen und von rund 4600 Proteinen nur spärliche (d.i. 1 bis 10) Veröffentlichungen, dass also 37 % unserer Proteine nur unzureichend beschrieben sind [2]. Abbildung 2. Dagegen handelt es sich bei den am häufigsten - d.i. in mehr als 500 Veröffentlichungen - publizierten Genen und auch Proteinen um Biomoleküle, die ohnehin bereits gut charakterisiert sind.
Wie die Autoren des Übersichtartikels schlussfolgern "gibt es keine offensichtlichen wissenschaftlichen oder finanziellen Gründe für den Rückgang bei der Entdeckung biomolekularer Mechanismen; wahrscheinlich regen die derzeitigen Instrumente der Wissenschaftsförderung Forscher nicht dazu an oder entmutigen sie sogar sich mit den schwierigen Probleme der Entdeckung von Genfunktionen abzugeben."[2].
Natürlich ist auch eine gewisse Bequemlichkeit nicht von der Hand zu weisen. Der Ausflug ins Neuland erfordert ja proteinspezifische Reagenzien - Antikörper, Inhibitoren - und die Suche nach in vitro Systemen, die das neue Protein in ausreichendem Maße exprimieren.
| Abbildung 2. Status der bis 2017 vorhandenen Literatur zu Proteinen und für Proteine codierende Gene im humanen Genom. A) Anteil der Proteine, die in <1 bis >500 Veröffentlichungen beschrieben wurden. B) 94 % der gesamten Literatur über Protein-kodierende Gene bezieht sich auf solche, über die in mehr als 500, bzw. 100 bis 500 Arbeiten berichtet wurde. (Bild aus [2]: Sinha et al., Proteomics 18, 2018. https://doi.org/10.1002/pmic.201800093 PMID: 30265449. Lizenz: cc-by-nc.) |
Die Unknome Datenbank ...........
Die wissenschaftliche Forschung hat sich also bis jetzt auf gut untersuchte Proteine konzentriert und schlecht erforschte Gene mit möglicherweise wichtigen physiologischen Funktionen vernachlässigt. Dies hat zu der Besorgnis geführt, dass wichtige Grundlagen oder klinische Erkenntnisse sowie das Potenzial für therapeutische Interventionen unerkannt bleiben; mehrere Initiativen wurden daher gestartet, um das Problem anzugehen.
Eine dieser Initiativen ist die von Wissenschaftern in Großbritannien eingerichtete frei zugängliche, benutzergseteuerte Datenbank "Unknome" (zusammengesetzt aus "Unknown" und "Genome"), die anderen Wissenschaftern einen Anreiz geben soll , Licht in das Dunkel der unerforschten Gene/Genprodukte zu bringen (homepage: https://unknome.mrc-lmb.cam.ac.uk/about/) [3].
In der "Unknome"-Datenbank wird jedes Protein anhand eines "Bekanntheitsgrades" (Score) danach eingestuft, wie viel oder wie wenig die Wissenschaftler darüber - u.a. über Funktion, artenübergreifende Konservierung in Spezies, Lokalisierung in Zellen, etc. - wissen.
..............und ihre Brauchbarkeit zur Identifizierung von Funktionen bislang unbekannter Gene
Um die Brauchbarkeit von Unknome als Grundlage für experimentelle Arbeiten zu bewerten, haben die Forscher eine Reihe von 260 menschlichen Proteinen ausgewählt, die in orthologer Form (d.i. in hoch konservierter Basenabfolge) auch in dem Modellorganismus der Fliege Drosophila vorliegen, und deren Funktion in beiden Spezies noch unbekannt war (Score < 1) [3]. Um den Beitrag dieser orthologen Gene zu einem breiten Spektrum biologischer Prozesse zu testen, haben sie diese Gene nacheinander (mit Hilfe von RNA-Interferenz) (partiell) ausgeschaltet. Für 62 dieser Gene war ein kompletter Knockout mit dem Überleben der Fliege nicht vereinbar. Unter den restlichen nicht-essentiellen Genen wurden 59 entdeckt, die zu wichtigen biologischen Funktionen beitragen, darunter zu Fertilität, Entwicklung, Gewebewachstum, Qualitätskontrolle von Proteinen (Entfernung schadhafter Proteine), Widerstandsfähigkeit gegen Stress, Fortbewegung, Signalübertragung über den Notch-Signalweg [3].
Ob und welche Effekte die orthologen Gene beim Menschen haben, ist noch nicht untersucht. Jedenfalls kann aber gefolgert werden, dass in den bislang vernachlässigten Teilen des Genoms/Proteoms wichtige biologische Prozesse kodiert sind.
Fazit
Die Unknome-Datenbank ist eine Ressource für Forscher, welche die Chancen unerforschter Bereiche der Biologie nutzen wollen. In ihrer Studie zeigen die britischen Forscher auf, dass trotz jahrzehntelanger umfangreicher genetischer Untersuchungen es offensichtlich viele Fliegengene gibt, deren essentielle Funktionen noch unbekannt sind; dasselbe gilt auch für die orthologen Gene des Menschen. Die Forscher hoffen, dass sich diese Datenbank mit zunehmender Nutzung in den kommenden Jahren verkleinern und neue biologische und therapeutische Erkenntnisse liefern wird. Dabei ist nicht auszuschließen, dass man auf völlig neue Bereiche biologischer Funktionen stößt.
[1] June 2000 White House Event: https://www.genome.gov/10001356/june-2000-white-house-event
[2] S. Sinha et al., Darkness in the Human Gene and Protein Function Space: Widely Modest or Absent Illumination by the Life Science Literature and the Trend for Fewer Protein Function Discoveries Since 2000. Proteomics 2018, 18, 1800093. DOI: 10.1002/pmic.201800093
[3] Rocha JJ, et al. (2023) Functional unknomics: Systematic screening of conserved genes of unknown function. PLoS Biol 21(8): e3002222. https://doi.org/10.1371/journal.pbio.3002222
Luftverschmutzung in Europa: Ammoniak sollte vordringlich reduziert werden
Luftverschmutzung in Europa: Ammoniak sollte vordringlich reduziert werdenFr, 04.08.2023— IIASA

![]() Bei der Verringerung der Luftverschmutzung steht Europa noch vor zahlreichen Herausforderungen. Ein Zuviel an reaktivem Stickstoff (Nr) - dieser schließt Stickoxide (NOx) und Ammoniak (NH3) mit ei n - trägt in Europa stark zur Luftverschmutzung durch Feinstaub (PM2.5) bei und stellt eine Bedrohung für die öffentliche Gesundheit dar. In der Entwicklung kosteneffizienter Fahrpläne zur Reduzierung der PM2,5-Belastung müssen sowohl die Effizienz der Reduzierung als auch die Implementierungskosten berücksichtigt werden. Ein internationales Team aus Forschern des IIAS A (International Institute for Applied Systems Analysis (IIASA, Laxenburg bei Wien), der Universität für Bodenkultur (Wien), der Universität von Zielona Gora (Polen) und der Peking University haben in einer neuen Studie die Reduzierung von Ammoniakemissionen als kosteneffiziente Maßnahme zur Verringerung der Feinstaubkonzentration in der Atmosphäre ermittelt.*
Bei der Verringerung der Luftverschmutzung steht Europa noch vor zahlreichen Herausforderungen. Ein Zuviel an reaktivem Stickstoff (Nr) - dieser schließt Stickoxide (NOx) und Ammoniak (NH3) mit ei n - trägt in Europa stark zur Luftverschmutzung durch Feinstaub (PM2.5) bei und stellt eine Bedrohung für die öffentliche Gesundheit dar. In der Entwicklung kosteneffizienter Fahrpläne zur Reduzierung der PM2,5-Belastung müssen sowohl die Effizienz der Reduzierung als auch die Implementierungskosten berücksichtigt werden. Ein internationales Team aus Forschern des IIAS A (International Institute for Applied Systems Analysis (IIASA, Laxenburg bei Wien), der Universität für Bodenkultur (Wien), der Universität von Zielona Gora (Polen) und der Peking University haben in einer neuen Studie die Reduzierung von Ammoniakemissionen als kosteneffiziente Maßnahme zur Verringerung der Feinstaubkonzentration in der Atmosphäre ermittelt.*
Zu den weltweit, auch in Europa führenden Umweltrisikofaktoren für vorzeitige Todesfälle zählen Verschmutzungspartikel mit einem Durchmesser von weniger als 2,5 Mikrometer in der uns umgebenden Luft; diese werden auch als Feinstaub oder PM2,5 (PM: Particulate Matter)bezeichnet. Zwar konnten bereits die Emissionen von Luftschadstoffen wie Schwefeldioxid, Feinstaub und Stickoxiden erfolgreich reduziert werden konnten, es werden aber die neuen von der Weltgesundheitsorganisation (WHO) festgelegten Richtlinien für Luftqualität (PM2,5-Jahresmittel 5 μg/m3) in vielen Teilen des Kontinents immer noch überschritten: im Jahr 2019 war dies für 97 % der europäischen Messstationen der Fall.
Ein Zuviel an reaktivem Stickstoff (Nr), inklusive Stickoxiden (NOx = NO + NO2), Ammoniak (NH3), Nitrat (NO3-) und Ammonium (NH4+), ist eine anerkannte Umweltbedrohung für Ökosysteme und verschlechtert die Qualität von Luft, Boden und Wasser. Die anthropogenen Stickstoffquellen haben seit 1960 dramatisch zugenommen, wodurch sich der globale Stickstoffkreislauf und die daraus resultierenden schädlichen Auswirkungen auf die menschliche Gesundheit und die Ökosysteme verschärft haben. Die Begrenzung der anthropogenen Stickstoffemissionen (hauptsächlich NOx und NH3) hat für den Umweltschutz hohe Priorität.
Wie die Autoren der neuen Studie, die soeben im Fachjournal Nature Communications veröffentlicht wurde [1] berichten, könnte das Aufhören der Ammoniak- und Stickoxidemissionen die PM2,5-Konzentration in Europa im Jahresdurchschnitt um 2,3 µg/m3 senken und jährlich etwa 100 000 vorzeitige Todesfälle verhindern. Abbildung 1. Für diese Aussagen beziehen die Autoren Schätzungen der Emissionen mit ein, Modellierungen von Luftqualität und der Relation Exponierung-Mortalität, Experimente zur Kontrolle von Nr. und Daten der Implementierungskosten.
Wenn es darum geht die Umweltverschmutzung zu verringern, hat die Senkung der Stickoxide eine permanente Wirkung. Die Reduzierung von Ammoniak ist sofort nicht gleich wirksam, wird aber entscheidend, wenn man eine erhebliche Verringerung der Verschmutzung anstrebt. Zudem ist bei gleich großer Verringerung der PM2,5-Belastung die Reduzierung der Ammoniakemissionen fünf- bis zehnmal so kosteneffizient; dies macht deutlich, dass ein Fokus auf die Verringerung der Ammoniakemissionen von entscheidender Bedeutung ist, um eine weitgehende Senkung der Luftverschmutzung in ganz Europa zu erreichen.
|
Abbildung 1. Der Beitrag der Emissionen von reaktivem Stickstoff (Nr) zur PM2,5-Luftverschmutzung über Europa für das Jahr 2015. Veränderungen der PM2,5-Konzentrationen (Delta PM2,5) bei Aufhören der a) anthropogenen Nr (NOx+NH3)-Emissionen, b) der NOx-Emissionen und c)der NH3-Emissionen (c). N-Anteile an der PM2,5-Luftverschmutzung durch Emissionen von d) Nr, e) NOx (e), und f) NH3. (Abbildung und Legende (von Redn. übersetzt und eingefügt aus Liu et al., 2023, Lizenz: cc-b [1].) |
" In unserem Modellierungsansatz konnten wir unterschiedliche chemische Regulatorsysteme feststellen. Die Auswirkungen sind in den verschiedenen Teilen Europas unterschiedlich, da die anfängliche chemische Zusammensetzung der Atmosphäre variiert", erklärt Zehui Liu, Hauptautor der Studie und Forscher am Laboratory for Climate and Ocean-Atmosphere Studies (Universität Peking, China).
"Wir sehen den gleichen Effekt, wenn die Emissionsminderung erfolgreich anläuft - sobald die Stickoxide reduziert sind, wird es immer wichtiger, Ammoniak zu entfernen".
Die Autoren sind der Überzeugung, dass ihre Ergebnisse einen Beitrag zur Festlegung politischer Prioritäten leisten werden. "Die meisten Stickoxidemissionen stammen von Fabriken und Fahrzeugen, und wir haben bereits Schritte unternommen, um diese zu kontrollieren. In der Landwirtschaft, die die Hauptquelle für Ammoniakemissionen ist, haben wir jedoch kaum Fortschritte gemacht. Das bedeutet eine Chance durch die Umsetzung wirksamer Maßnahmen im Agrarsektor positive Ergebnisse zu erzielen", bemerkt Wilfried Winiwarter, Mitautor der Studie und leitender Wissenschaftler in der IIASA-Forschungsgruppe für Umweltverschmutzung.
"Wir haben festgestellt, dass die Verbesserungen der Luftqualität von Region zu Region unterschiedlich sind. Eine weitere Verbesserung der Luftqualität bei Feinstaub würde darüber hinaus auch strengere Kontrollmaßnahmen für andere Schadstoffe als Stickoxide und Ammoniak erfordern", ergänzt Harald Rieder, ein weiterer Mitautor der Studie und Professor an der Universität für Bodenkultur (BOKU) Wien.
"Wir können auch für andere Teile der Welt, wie China und Indien Erfahrungen sammeln. Nachdem wir in China erfolgreich mit der Verringerung der Luftverschmutzung begonnen haben, müssen wir nun die nächsten Schritte zur Emissionsreduzierung festlegen", schließt Lin Zhang, Mitautor der Studie und Professor an der Universität Peking.
[1]Liu, Z., Rieder, H.E., Schmidt, C., Mayer, M., Guo, Y., Winiwarter, W., and Zhang, L. (2023). Optimal reactive nitrogen control pathways identified for cost-effective PM2.5 mitigation in Europe. Nature Communications 14, 4246. DOI: 10.1038/s41467-023-39900-9; open access.
*Der Blogartikel basiert auf der IIASA-Presseaussendung “ Ammonia reduction should be prioritized in Europe’s fight against air pollution
https://iiasa.ac.at/news/jul-2023/ammonia-reduction-should-be-prioritized-in-europes-fight-against-air-pollution vom 26. Juli 2023. Diese wurde von der Redaktion aus dem Englischen übersetzt, geringfügig für den Blog adaptiert und mit Texten und mit Abbildung 1 aus der zugrundeliegenden Veröffentlichung [1] ergänzt. IIASA ist freundlicherweise mit Übersetzung und Veröffentlichung seiner Nachrichten in unserem Blog einverstanden.
Luftverschmutzung im ScienceBlog:
- Susanne Donner 13.07.2023: Schadstoffe - Pathogene Effekte auf die grauen Zellen
- IIASA, 20.12.2020: COVID-19, Luftverschmutzung und künftige Energiepfade
- Inge Schuster, 16.11.2017: Einstellung der EU-Bürger zur Umwelt (Teil 1) – Ergebnisse der ›Special Eurobarometer 468‹ Umfrage
- IIASA, 18.05.2017: Überschreitungen von Diesel-Emissionen — Auswirkungen auf die globale Gesundheit und Umwelt
- IIASA, 25.09.2015: Verringerung kurzlebiger Schadstoffe – davon profitieren Luftqualität und Klima
- Johannes Kaiser & Angelika Heil, 31.07.2015: Feuer und Rauch: mit Satellitenaugen beobachtet
- Inge Schuster, 12.12.2014: Was macht HCB so gefährlich?
Welche Bedeutung messen EU-Bürger dem digitalen Wandel in ihrem täglichen Leben bei? (Special Eurobarometer 532)
Welche Bedeutung messen EU-Bürger dem digitalen Wandel in ihrem täglichen Leben bei? (Special Eurobarometer 532)Sa, 29.07.2023 — Redaktion 
![]()
Internet und digitale Werkzeuge sind nicht länger ein Spielzeug für anfängliche Anwender, sondern für Bürger, Unternehmen, Organisationen und Regierungen zu einem wesentlichen Bestandteil der heutigen Gesellschaft geworden. Im "Politikprogramm 2030 für die digitale Dekade" sind EU-Mitgliedstaaten und EU-Kommission gemeinsame Verpflichtungen eingegangen, die mit konkreten und überprüfbaren Zielvorgaben und Zielpfaden den digitalen Wandel vorantreiben sollen. Vier Ziele stehen dabei im Fokus: die Erhöhung der digitalen Kompetenzen, die sichere und nachhaltige digitale Infrastruktur, der digitale Wandel in Unternehmen und die Digitalisierung öffentlicher Dienste. Wie die EU-Bürger den digitalen Wandel und dessen Auswirkungen auf ihr persönliches Leben und die Gesellschaft betrachten, hat kürzlich eine europaweite Umfrage erhoben (Special Eurobarometer 532: The Digital Decade [1]).
Wie bereits erstmals im Jahr 2021 [2] wurde auch in der aktuellen Umfrage ermittelt, wie EU-Bürger die Zukunft von digitalen Werkzeugen und Internet und sehen und welche Auswirkungen ihrer Meinung nach Internet, digitale Produkte, Dienstleistungen und Werkzeuge bis 2030 auf ihr persönliches Leben haben werden.
Im Auftrag der EU-Kommission ließ Kantar Public (Kantar Belgium) die Umfrage in den 27 Mitgliedstaaten vom 2. bis 26. März 2023 durchführen; insgesamt 26 376 Personen ab 15 Jahren und aus unterschiedlichen sozialen und demographischen Gruppen - rund 1000 Personen je Mitgliedsland - nahmen teil. Diese wurden persönlich (face to face) in ihrem Heim oder in Video-Ferninteraktion in ihrer Muttersprache interviewt.
Der neue Bericht: Die digitale Dekade [1]
knüpft an die Ergebnisse der früheren Umfrage [2] an und behandelt das Thema in drei großen Fragenkomplexen.
Der erste Teil untersucht, wie wichtig digitale Werkzeuge und Internet nach Meinung der EU-Bürger in ihrem Leben bis 2030 sein werden und welche Auswirkungen aus deren Anwendung resultieren werden. Teilnehmer wurden gefragt, ob sie erwarteten, dass digitale Werkzeuge und Internet ihr tägliches Leben erleichtern werden, beispielsweise beim Zugang zu allgemeiner und berufsbildender Bildung, zu Gesundheitsdiensten oder zu Kontakten mit anderen Menschen.
Der zweite Teil des Berichts ermittelt, wie die EU-Bürger die nationalen Prioritäten im Bereich der digitalen Technologien einschätzen und welche Bedeutung sie der Zusammenarbeit der EU- Mitgliedstaaten in den digitalen Technologien beimessen (beispielsweise in Hinblick auf erhöhte Investitionen in innovativere und sicherere digitale Technologien oder, um sicherzustellen, dass die gleichen digitalen Technologien und Dienste für alle und überall in der EU zugänglich sind).
Der dritte Teil des Berichts befasst sich damit, wie EU-Bürger Anwendung und Schutz der Grundrechte im Onlinebereich wahrnehmen. Konkret wurden sie gefragt, ob ihnen bewusst ist, dass Rechte wie das Recht auf freie Meinungsäußerung Meinungsfreiheit, Privatsphäre oder Nichtdiskriminierung sowohl online als auch offline gelten sollten. Sie sollten auch angeben, wie gut ihrer Meinung nach die EU ihre Rechte im Onlinebereich schützt und wie gut die digitalen Rechte und Grundsätze in ihrem Land angewandt werden, z. B. in Bezug auf die Wahrung der Privatsphäre im Internet, d.i. das Respektieren der Vertraulichkeit von Kommunikation und Informationen auf Geräten.
Da die Darstellung der wesentlichen Umfrage-Ergebnisse die übliche Länge der ScienceBlog-Artikel weit übersteigt, wird über das für Europas Zukunft so wichtige Thema in zwei Teilen berichtet. Der gegenwärtige Artikel zeigt nur die Meinungen zum digitalen Wandel und dessen Auswirkungen auf zehn wesentliche Lebensbereiche auf (Teil 1 der Umfrage). Es werden dazu die Antworten der Bürger im EU27-Durchschnitt den Antworten unserer Landsleute in Österreich und unseren Nachbarn in Deutschland gegenüber gestellt.
Die Bedeutung der Digitalisierung im Alltag.................
Der (weitaus) überwiegende Anteil der Bevölkerung in allen Mitgliedstaaten ist der Ansicht, dass digitale Werkzeuge und Internet in ihrem Leben bis 2030 eine sehr wichtige oder ziemlich wichtige Rolle spielen werden. Insgesamt gesehen ist dies die Meinung von rund vier Fünftel der Bürger (79 %) im EU27-Schnitt. Die Länderanalyse zeigt allerdings große Unterschiede - während über 90 % der Bevölkerung in Holland, Schweden und Dänemark den digitalen Wandel für ihr persönliches Leben als wichtig betrachten, messen die Länder in Richtung Osten/Südosten dem offensichtlich weniger Bedeutung bei; Österreich (71 %) und Rumänien (59 %) sind die Schlusslichter. Abbildung 1.
| Abbildung 1: Abbildung 1. Länderanalyse der Antworten auf Frage 1: Wie wichtig werden Ihrer Meinung nach digitale Technologien in ihrem Leben bis 2023 sein? Die Antworten "sehr wichtig" und "ziemlich wichtig" sind zusammengenommen in % der Antworten je Land angegeben. (Bild leicht modifiziert aus [1]) |
Im Vergleich zu 2021 ist in 22 Mitgliedstaaten der Anteil der Befragten, die angeben, dass digitale Werkzeuge und das Internet bis 2030 in ihrem Leben wichtig sein werden, aber zurückgegangen, vor allem in Belgien und Tschechien um 10%, in Finnland um 9 %, in Deutschland um 7 %; der EU-Schnitt lag 2021 bei 81 %, in Österreich bei 72 %, in Rumänien bei 61 % [2].
.......in wesentlichen Lebensbereichen
Insgesamt wurden die Teilnehmer zur Bedeutung der Digitalisierung bis 2030 für sie persönlich in zehn wesentlichen Lebensbereichen befragt. Die Antworten, gelistet nach fallender Wichtigkeit im EU27-Schnitt, sind in Abbildung 2 aufgezeigt und die jeweiligen Antworten aus Österreich und Deutschland gegenüber gestellt. Offensichtlich haben Teilnehmer aus Österreich und Deutschland die Rolle der Digitalisierung in allen diesen Bereichen für weniger wichtig gehalten, als dies im EU27-Schnitt der Fall war.
| Abbildung 2: Antworten auf Fragen zur persönlichen Wichtigkeit des digitalen Wandels in bestimmten Lebensbereichen; im EU27-Schnitt, sowie in Österreich und Deutschland. Angaben in % der Antworten; Je EU27 und Land bedeuten dunklere Blautöne: sehr wichtig, hellere Blautöne: ziemlich wichtig, hellere Rosetöne: nicht sehr wichtig, dunkle Rosetöne: völlig unwichtig und Grautöne: keine Aussage. (Bilder von Redn. erstellt/modifiziert aus Abbildung QB2 und Daten von [1]). |
Listet man die Wichtigkeit, die nach Meinung der Europäer digitale Technologien in ihrem persönlichen Leben bis 2030 spielen werden, so liegt an erster Stelle deren zentrale Rolle bei Kontakten mit anderen Menschen, Freunden und Familie: dies erwartet die weitaus überwiegende Mehrheit der EU27 (82 %); in Österreich sind dies 76 %, in Deutschland 74 %. Fast ebenso viele Europäer (81 %) sehen auch den digitalen Zugang zu öffentlichen Dienstleistungen als wichtig an (in Österreich 73 %, in Deutschland 77 %).
An dritter Stelle der Wichtigkeit scheint der digitale Wandel im Gesundheitswesen auf: 76 % im EU27- Schnitt (67 % in Österreich, 71 % in Deutschland) erwarten eine Verbesserung im Zugang zu Gesundheitsdienstleistungen oder in deren Inanspruchnahme. Ähnlich viele Europäer (EU27: 74 %, jeweils 70 % in Österreich und Deutschland) haben ähnliche Erwartungen in Bezug auf Online-Handel und Dienstleistungen mit anderen EU-Ländern, sowie für den Zugang zu und die Nutzung von Verkehrsdienstleistungen (EU27: 73 %, Österreich 62 %, Deutschland: 66 %).
In weiterer Folge geht es um die Rolle, die digitale Technologien für den Zugang zu Bildungs- und Aus- und Weiterbildungsmöglichkeiten spielen werden - etwa sieben von zehn Europäern (71 %) sehen diese als wichtig an; in Österreich sind dies nur 63 %, in Deutschland 68 %. Den Zugang zu, die Interaktion mit und/oder die Erstellung von Online-Materialien/Inhalten halten ebenso 71 % der EU27 für wichtig; in Österreich sind dies 62 %, in Deutschland 63 %.
Etwas mehr als zwei Drittel der EU-Bürger (68 %) erwarten, dass sich der digitale Wandel positiv auf die Art und Weise auswirkt, wie sich die Menschen am demokratischen Leben beteiligen; in Österreich sind dies 63 %, in Deutschland 65%.
Rund zwei Drittel (66 %) der Europäer meinen, dass digitale Technologien, wie beispielsweise zur Überwachung der persönlichen Emissionen, des Energieverbrauchs oder der Teilnahme an online-Meetings, einen wichtigen Beitrag im Kampf gegen den Klimawandel leisten können; 59 % der Österreicher und Deutschen teilen diese Meinung.
Eine Mehrheit (63 %) der Europäer erwartet, dass digitale Technologien einen großen Einfluss auf die Fernarbeit (Homeoffice) haben werden; in Österreich sind dies 57 % in Deutschland 56 %.
Eine demographische Analyse
der Antworten zeigt eine klare Abhängigkeit von Alter und Ausbildung der Befragten. Die Ansicht, dass digitale Werkzeuge und das Internet in Leben eine wichtige Rolle spielen werden, nimmt von der Altersgruppe 15 bis 24 Jahre bis zur Altersgruppe 40 - 54 Jahre kontinuierlich ab. Ab 55 Jahren erscheinen digitale Technologien dann im wesentlichen für den Kontakt mit anderen Menschen (71 %), den Zugang zu öffentlichen Diensten (68 %) und zu Gesundheitsdiensten (65 %) wichtig, in den meisten anderen Lebensbereichen ist die Wichtigkeit auf um die 50 % und darunter gesunken.
Insgesamt sehen Befragte mit Hochschulbildung digitale Technologien mit größerer Wahrscheinlichkeit als für ihr Leben wichtig an (90 %), als Absolventen mit einem Sekundarschulabschluss (16 bis 19 Jahre) (75 %) und Personen, die nur bis zum Alter von 15 Jahren zur Schule gingen (49 %).
Fazit
Wenn im Durchschnitt 79 % der EU-Bürger die Ansicht äußern, dass digitale Technologien für ihr Leben bis 2030 wichtig sein werden, so weist dies auf den ersten Blick auf eine sehr hohe Zustimmung zu einem schnellen Fortschreiten der europäischen Digitalisierung hin. Allerdings ist seit der ersten Umfrage 2021 der Anteil positiven Antworten in 22 der 27 EU-Mitgliedsländern gesunken, und es wird in dieser Umfrage auch nicht erhoben, ob und welche erforderlichen Kompetenzen die Befragten bereits besitzen. Verstörend sind die Antworten der Teilnehmer aus den "reichen" Ländern Österreich und Deutschland, welche die Rolle der Digitalisierung in allen Lebensbereichen für weniger wichtig halten, als dies im EU27-Schnitt der Fall ist. Insbesondere ist die Nutzung der digitalen Technologien für allgemeine und berufliche Bildung hervorzuheben, die 35 % der Österreicher und 28 % der Deutschen für unwichtig halten, und die Nutzung von Online-Materialien/Inhalten - diese halten 33 % der Österreicher und 32 % der Deutschen für unwichtig.
Im Übrigen weist die angegebene Wichtigkeit digitaler Technologien das gleiche Gefälle in Richtung Osten/Südosten auf, wie das Interesse der EU-Bürger an Wissenschaft und Technologie und ihre diesbezügliche Informiertheit [3].
[1] Special Eurobarometer 532: The Digital Decade (June, 2023). doi: 10.2759/14051
[2] Special-Eurobarometer 518: Digital rights and principles (December 2021). doi: 10.2759/30275
[3] I. Schuster, 3.10.2021: Special Eurobarometer 516: Interesse der europäischen Bürger an Wissenschaft & Technologie und ihre Informiertheit
Wenn das Wasser die Menschen verdrängt - Megastädte an Küsten
Wenn das Wasser die Menschen verdrängt - Megastädte an KüstenDo, 20.07.2023 - Duška Roth

![]() Seit 2007 leben mehr Menschen in Städten als auf dem Land. Nach Schätzungen der Vereinten Nationen werden 2030 fünf von achteinhalb Milliarden Menschen in Städten wohnen. Schon heute gibt es 34 Städte beziehungsweise Metropolregionen mit mehr als 10 Millionen Einwohnern. Sie werden Megastädte oder Megacitys genannt und sind geprägt von Gegensätzen zwischen größter Armut und höchstem Luxus. Viele von ihnen liegen nah an der Küste. Damit sind sie durch den steigenden Meeresspiegel unmittelbar bedroht.*
Seit 2007 leben mehr Menschen in Städten als auf dem Land. Nach Schätzungen der Vereinten Nationen werden 2030 fünf von achteinhalb Milliarden Menschen in Städten wohnen. Schon heute gibt es 34 Städte beziehungsweise Metropolregionen mit mehr als 10 Millionen Einwohnern. Sie werden Megastädte oder Megacitys genannt und sind geprägt von Gegensätzen zwischen größter Armut und höchstem Luxus. Viele von ihnen liegen nah an der Küste. Damit sind sie durch den steigenden Meeresspiegel unmittelbar bedroht.*
Urbane Räume sind Anziehungspunkte für Menschen auf der Suche nach einem besseren Leben. Der stetige Zuwachs an Menschen stellt gerade die Metropolen des globalen Südens vor gewaltige strukturelle Probleme: Dort schreitet der Anstieg der Bevölkerung so rasant voran, dass Arbeitsplätze fehlen, die Behörden überfordert sind und der Ausbau von Infrastrukturen und Wohnraum nicht Schritt halten kann. Die Folgen sind sozioökonomische und räumliche Fragmentierungen der Stadtgesellschaft. Es entstehen informelle Stadtviertel, die je nach Region als Slums oder Favelas bezeichnet werden und von schlechter Baustruktur und prekären Lebensverhältnissen geprägt sind. Menschen, die dort wohnen, sind auf mehreren Ebenen marginalisiert: Sie sind sowohl räumlich als auch wirtschaftlich, politisch und sozial ausgegrenzt. So haben sie etwa einen schlechten oder gar keinen Zugang zu sauberem Wasser oder medizinischer Versorgung und können nur eingeschränkt auf Extremereignisse wie Erdbeben oder Hochwasser reagieren. Megastädte in Küstennähe sind daher vom Anstieg des Meeresspiegels infolge des Klimawandels besonders bedroht (Abbildung 1). Die meisten Megastädte und die größten Metropolregionen der Welt befinden sich in Asien – einer Region, die bereits heute von klimabedingten Extremereignissen stark betroffen ist und laut Experten auch in der Zukunft noch häufiger und stärker betroffen sein wird. Dort treffen Naturgefahren auf gesellschaftlich vermittelte Vulnerabilität.
| Abbildung 1. Städte weltweit. Die Karte zeigt den prozentualen Anteil von Städten mit 500.000 oder mehr Einwohnern (Daten aus dem Jahr 2018). Vierzehn Länder oder Gebiete besitzen einen niedrigen Urbanisierungsgrad, d. h. weniger als 20 % ihrer Bevölkerung leben in städtischen Gebieten. Im Gegensatz dazu liegt der Anteil der städtischen Bevölkerung in 65 Ländern bereits bei über 80 Prozent. Viele Megastädte (rote Kreise) befinden sich an Küsten oder in Küstennähe. © United Nations, DESA, Population Division (2018) / CC BY 3.0 IGO |
Land unter in Asien
Der globale Meeresspiegel ist im 20. Jahrhundert zunehmend angestiegen (Abbildung 2). Aufgrund der Trägheit des Klimasystems wird er – selbst bei einem sofortigen Stopp aller Treibhausgas-Emissionen – für mehrere Jahrhunderte weiter steigen. Hunderte Millionen Menschen, die weltweit in Küstennähe leben, sind durch diesen Anstieg bedroht. In Asien könnten Metropolen wie Shanghai, Hanoi, Dhaka, Mumbai, Jakarta oder Kolkata überflutet werden. Besonders gefährdet sind Delta-Gebiete – flache Regionen, die durch den Zusammenschluss von Flussarmen und deren Sedimentablagerungen entstehen. Solche Landstriche sind klassische Siedlungsgebiete für Menschen. Ein Grund sind die Schwemmböden, die mit nährstoffreichen Sedimenten aus dem Fluss versorgt werden. Diese fruchtbaren Böden bringen hohe Erträge in der Landwirtschaft, ihre Nutzung führt aber gleichzeitig zu Bodenabsenkungen: Als Folge der Trockenlegung im Zuge des Ackerbaus ziehen sich tiefer liegende Erdschichten zusammen. Dadurch entstehen Absenkungen, die sich über große Gebiete verteilen. In Ballungszentren wie Dhaka und Kolkata kommen Bodenversiegelung und die exzessive Entnahme von Grundwasser hinzu. So entstehen regionale Absenkungen von mehreren Zentimetern pro Jahr, die den lokalen Meeresspiegelanstieg verstärken. Häufiger auftretende Extremwetterlagen verursachen Sturmfluten, die im Zusammenspiel mit einem erhöhten Meeresspiegel katastrophale Folgen haben. Wohngebiete werden überflutet, das anbrandende Meerwasser trägt die Küste ab und vernichtet Lebensraum und Anbauflächen. Dies betrifft vor allem dicht besiedelte, informelle Siedlungen, die häufig in Küstennähe errichtet werden.
| Abbildung 2. Anstieg des Meeresspiegels. Das Diagramm zeigt die Veränderung des globalen Meeresspiegels seit 1993, wie sie von Satelliten beobachtet wird. Infolge der globalen Erwärmung steigt der Meeresspiegel in einem Ausmaß an, wie es in den letzten 2.500 Jahren noch nie vorgekommen ist. © NASA‘s Goddard Space Flight Center |
Als Ethnograf in der Megastadt
Die Metropolregion Kolkata befindet sich im bengalischen Delta am Fluss Hugli, einem Mündungsarm im westlichen Gangesdelta. Die Gegend liegt durchschnittlich nur sechs Meter über dem Meeresspiegel. Kolkata ist die Hauptstadt des Bundesstaates Westbengalen und mit ihren 4,5 Millionen Einwohnern (Zahlen vom letzten verfügbaren Zensus im Jahr 2011) die siebtgrößte Stadt Indiens. In der Metropolregion leben demnach 14,1 Millionen Einwohner. Andere, inoffizielle Schätzungen gehen von bis zu 30 Millionen aus. Damit bildet diese Region auf einer Fläche von 187,33 Quadratkilometern den drittgrößten Ballungsraum des Landes. Die Bevölkerungsdichte entspricht derjenigen, als würden alle Einwohner von Sachsen, Sachsen-Anhalt, Thüringen, Berlin, Mecklenburg-Vorpommern und Brandenburg auf einem Gebiet so groß wie Potsdam leben. Obwohl der Wohnraum immer knapper wird, ziehen mehr und mehr Menschen nach Kolkata. Was macht die Megastadt so attraktiv? Arne Harms, Wissenschaftler am Max-Planck-Institut für ethnologische Forschung in Halle, nennt zwei Faktoren: „Kolkata ist ein Sehnsuchtsziel für Migranten aus Indien, die in die Region ziehen, um ihr Überleben zu sichern. Jobs als Tagelöhner findet man dort relativ leicht. Aber Kolkata ist auch eine Stadt des kulturellen Erbes und für die Bengalen sehr bedeutsam.“ Hinzu kommt, dass viele Menschen aus den Küstenregionen in die Städte ziehen, weil sie wegen eines drohenden oder akuten Hochwassers ihre Wohnungen verlassen müssen. Um zu verstehen, wie ökologische und ökonomische Krisen oder die Globalisierung das Leben an der Küste verändern, verknüpft Harms Ansätze aus der politischen Ökologie und der Infrastrukturforschung.
Die politische Ökologie betrachtet Umweltprobleme in ihrem historischen, politischen und ökonomischen Kontext. Welche Rolle spielen wirtschaftliche Verhältnisse und Machtdynamiken für den Zugang zu Trinkwasser oder den Schutz vor Fluten? Wie beeinflusst der steigende Meeresspiegel politische und wirtschaftliche Entscheidungen und Abläufe? Fragen wie diese ermöglichen es, komplexe Zusammenhänge aufzudecken. Die Infrastrukturforschung beschäftigt sich zum Beispiel mit der Frage, wie Dämme, Straßen und Brücken die Entscheidungen und das Handeln der Menschen beeinflussen. Bei der Datenanalyse untersuchen Forschende die Interessen und Machtverhältnisse der beteiligten Akteure und deren konkrete Handlungsmuster. Harms Forschung dreht sich um das zentrale Thema, wie sich das Leben in Küstenregionen durch den steigenden Meeresspiegel und die Urbanisierung verändert. Um das herauszufinden, sammelt er bereits seit 2009 Daten – in Interviews und Gruppendiskussionen, durch teilnehmende Beobachtung und Experteninterviews sowie die Auswertung von Dokumenten und Archivmaterial.
Hochwasser als Teil des Lebens
Die rasant wachsenden Slums in Kolkata sind besonders anfällig für extreme Naturereignisse. Aus Mangel an Alternativen entstehen sie oft an gefährdeten Standorten und sind daher ausgesprochen vulnerabel. Arne Harms befasst sich vor allem mit der sozialen Vulnerabilität der Bewohner: Inwiefern sind gesellschaftliche, soziale, ökonomische und naturräumliche Kategorien dafür verantwortlich, dass sich Menschen in vulnerablen Situationen befinden? Gleichzeitig untersucht er auch die Resilienz der Menschen – die Kapazitäten, aus denen sie schöpfen, um ihr Überleben zu sichern. „An der bengalischen Küste sind Überflutungen ein rhythmisch wiederkehrendes Phänomen“, sagt der Wissenschaftler. Diese Rhythmik betrifft zum einen die Jahreszeiten: In der Regenzeit sind die Wasserstände höher und die Deiche einem höheren Druck ausgesetzt. Dadurch erodiert das Land stärker. Die Rhythmik folgt aber auch den Mondphasen, denn bei Vollmond und Neumond sind die Gezeitenfluten höher als an anderen Tagen. Weil beides verzahnt ist, gelten die Vollmonde der Regenzeiten als die gefahrvollste Zeit: Deiche brechen und Menschen verlieren ihre Häuser. Schließlich ist auch das Leben mit Landverlust rhythmisch, denn mit den wiederkehrenden Fluten fallen immer wieder Teile der Küste der Erosion zum Opfer.
Die Menschen in der Region wissen um die Gefahr wiederkehrender Hochwasser und reagieren innerhalb ihrer Möglichkeiten. Sie ziehen weiter ins Landesinnere, wenn sie bei Bekannten oder Verwandten unterkommen oder es sich leisten können, ein Stück Land zu kaufen. Viele ziehen in notdürftig errichtete Hütten am Straßenrand. Dort werden sie häufig wieder von Flut und Erosion eingeholt, und der Prozess der Vertreibung durch das herannahende Wasser beginnt von neuem. In unmittelbaren Küstengebieten am Rand der Metropolregion bietet sich ein ähnliches Bild: „Die meisten Menschen, mit denen ich gesprochen habe, suchten in einiger Entfernung von der Küste Sicherheit, nur um vom herannahenden Meer nach ein paar Monaten oder Jahren wieder eingeholt zu werden“, sagt Arne Harms. „Sie erleben also einen Wechsel von sicheren, trockenen Perioden und Zeiten, in denen sie unmittelbar vom Meer bedroht sind. Etliche haben bereits fünf- oder sechsmal ihre Bleibe verloren.“ Doch trotz der immer wiederkehrenden Gefahr durch das Wasser ziehen nur wenige weiter weg: „Die gewohnte Umgebung, die lokalen Netzwerke und gemeinsame Geschichten bieten den Menschen Sicherheit“, sagt der Wissenschaftler. Hinzu kommt, dass die Migration in weit entfernte Städte im Landesinneren viele Gefahren birgt: „Die meisten, die auf diversen Migrationspfaden unterwegs sind, kommen mit ziemlich üblen Geschichten nach Hause. Viele werden von Mittelsmännern um ihren Lohn gebracht. Sexuelle Übergriffe sind an der Tagesordnung, besonders auf Frauen, die ihr Geld als Hausangestellte verdienen. In der Ferne erleben die Menschen Entrechtung, Willkür, und Ausbeutung. Deshalb bleiben sie lieber dort, wo sie sind, und verharren in ihrer vulnerablen und marginalisierten Lage.“
Hinzu kommt, dass die Behörden von Kolkata mit dem rasanten Bevölkerungswachstum längst überfordert sind. Oft werden die marginalisierten Teile der Bevölkerung bei der Stadtplanung benachteiligt. Stattdessen setzen sich immer mehr die Ideale der Mittelklasse durch. Informelle Siedlungen finden darin keinen Platz, denn sie gelten als Orte von Krankheit, Armut und Schmutz. Harms schildert, dass die Menschen in den Slums faktisch entrechtet werden. Sie sehen sich vom Bauboom überrollt, durch Gentrifizierung verdrängt: „Neue Skyscraper-Bauprojekte sind nur für die Mittelklasse vorgesehen, welche die Arbeitskraft der marginalisierten Bevölkerung nutzt, etwa als Bauarbeiter oder Hausangestellte. Rehabilitationsprojekte für informelle Siedlungen werden am Stadtrand geplant, weit entfernt von guten Arbeitsplätzen und Einkommensmöglichkeiten. Sie sind daher oft von vornherein zum Scheitern verurteilt.“ In Folge siedeln sich die Menschen wieder auf Freiflächen an, die näher am Zentrum liegen. So schaffen sie neue informelle Strukturen und Siedlungen. Es entsteht ein Kreislauf, „ein Ziehen und Zerren um Land und Ressourcen“, so Harms. Der Wissenschaftler hat in Kolkata mit Herrn Das gesprochen. Dieser ist im Alter von 37 Jahren in die Stadt gezogen. Wegen der Fluten und der Küstenerosion hatte er zuvor fünfmal seine Bleibe verloren. Im Dorf an der Küste sah er daher für sich und seine Familie keine Zukunft. Doch wie er sagt, werde auch das Leben in dem Viertel, in dem er jetzt wohnt, immer schwieriger – vor allem wegen wachsender Probleme, an Trinkwasser zu kommen. Zudem steht er kurz davor, sein Haus erneut zu verlieren: Die informelle Siedlung, in der er mit seiner Familie wohnt, muss weiteren Hochhäusern weichen. So haben Flut und Erosion einerseits sowie Gentrifizierung andererseits für die Menschen im bengalischen Delta letztlich die gleichen Auswirkungen: Sie wissen nicht, wo sie bleiben sollen (Abbildung 3).
| Abbildung 3. Vulnerable Bevölkerung. Menschen in küstennahen Metropolregionen müssen aufgrund von Hochwasser immer wieder umziehen. Die farbigen Pfeile zeigen Kreisläufe. Rot: Menschen verlieren ihren Wohnraum und siedeln sich in einiger Entfernung von der Küste wieder an. Dort werden sie nach kurzer Zeit von der nächsten Überschwemmung eingeholt. Blau: Menschen ziehen an den Stadtrand. Es bilden sich informelle Siedlungen, die überschwemmt werden, die Menschen müssen neue Wohnplätze suchen. Grün: In den Slums werden Menschen durch Gentrifizierung verdrängt und siedeln sich wieder auf Freiflächen an, die näher am Zentrum liegen. So entstehen neue informelle Siedlungen. © MPG / CC BY-NC-SA 4.0 |
Wenn das Wasser steigt
Eine Möglichkeit, den Überflutungen durch den steigenden Meeresspiegel entgegenzuwirken, ist der Bau von Deichen. „Diese bieten aber nur eine trügerische Sicherheit“, sagt Arne Harms. „Durch das Absinken des Bodens bildet sich hinter dem Deich eine Art Tasse, die bei einem Dammbruch vollläuft und aus der das Wasser nicht mehr ablaufen kann. Deiche bieten daher keinen ausreichenden Schutz. Sie vermitteln zunächst Sicherheit, doch wenn sie brechen, sind die Folgen umso gravierender.“ Eine andere Lösung besteht darin, das Wasser abzupumpen. In Kolkata haben Stadtplaner und Investoren einen großen innerstädtischen Bereich, den Salt Lake District, weitgehend überschwemmungssicher gemacht. Sie haben das Land höher gelegt, Pumpen installiert, Schleusen und Dammsysteme gebaut – ein gigantischer Aufwand zum Hochwasserschutz. Heute wohnt in dem Gebiet eine wohlhabende Bevölkerung. In seinem Umkreis wachsen dagegen die informellen Siedlungen derjenigen, die Dienstleistungen für die Reichen erledigen. „Diese Menschen leben mit einem nochmals höheren Hochwasserrisiko, weil dort zusätzlich das Wasser aufläuft, das aus den geschützten Bereichen abgepumpt wird“, sagt Arne Harms. „So gehen derlei Prestigeprojekte an einer Lösung vorbei, weil sie die Frage der sozialen Verwundbarkeit ausklammern.“ Bisherige Hochwasserschutzprogramme in der Region konzentrieren sich vor allem auf Schutzräume für den Ernstfall, sogenannte Cyclone Shelter. Während diese bei Sturmfluten Menschenleben retten, können sie dem Problem versinkender Landschaften nichts entgegensetzen. So müssen die Menschen, die in informellen Siedlungen an den gefährdeten Stadträndern leben, in ihrem Alltag mit der ständig drohenden Katastrophe zurechtkommen. Um ihren Lebensraum zu erhalten, reparieren sie teils in Eigenregie und damit illegal die Deiche. So schützen sie – weitgehend unbemerkt – auch die Menschen im Inland.
Verteilte Katastrophe
Auf der Grundlage seiner Forschung im Raum Kolkata entwickelte Arne Harms die These der „Verteilten Katastrophe“. Sie schlägt nicht als ein weithin sichtbares Schadensereignis zu, sondern ist verteilt in Raum und Zeit. In den Küstenregionen tritt sie in breiter Front auf, geprägt durch Jahreszeiten und Gezeiten. Die Menschen, die mit dieser konstanten Bedrohungslage leben, sehen die „echte Katastrophe“ in der Küstenerosion. Sie raubt ihnen das Land und damit die wichtigste Grundlage von Ökonomie, Zugehörigkeit und Geschichte. „Die Sorgen jener Menschen im bengalischen Delta, die mit bröckelnden Deichen, wiederkehrenden Überschwemmungen und dem Verschwinden von Land zu kämpfen haben, bleiben von staatlichen Institutionen und den mit humanitärer Hilfe beauftragten NGOs bisher weitgehend unberücksichtigt“, sagt der Wissenschaftler. „Trotz ihrer gravierenden Auswirkungen bleibt die Küstenerosion unter dem Radar des Nothilfeapparats. Daher ist nur wenig über das Leid der Menschen bekannt, die am Rand der erodierenden Küstenlinie Indiens ausharren.“ Um soziale Gerechtigkeit zu fördern, fordert der Max-Planck-Forscher deshalb eine neue Definition der Katastrophe: „Der Begriff „Katastrophe“ bezeichnet meist großflächige Schadensereignisse, die massenhaft Tod und Zerstörung bringen. Eine neue Definition müsste auch solche Dynamiken beinhalten, die räumlich und zeitlich entgrenzt und nur selten tödlich sind, die aber eine existenzielle Bedrohung darstellen und daher von den Menschen als Katastrophe erlebt werden.“ Was fehlt, sind nach Ansicht des Wissenschaftlers vor allem Ansätze einer gerechten Umsiedlung sowie ein ausgeklügelter Küstenschutz, der auch verarmte Küstengebiete und deren Bewohner berücksichtigt: „Nur so lässt sich die Bedrohung durch den weiter steigenden Meeresspiegel in den dicht besiedelten Küstenregionen Asiens abmildern.“
* Der Artikel von Duška Roth ist unter dem Titel: " Megastädte an Küsten- Wenn das Wasser die Menschen verdrängt " im Geomax 28-Heft der Max-Planck-Gesellschaft im Sommer 2023 erschienen https://www.max-wissen.de/max-hefte/geomax-28-megastaedte/. Mit Ausnahme des Titels wurde der unter einer cc-by-nc-sa Lizenz stehende Artikel unverändert in den Blog übernommen.
Schadstoffe - Pathogene Effekte auf die grauen Zellen
Schadstoffe - Pathogene Effekte auf die grauen ZellenDo. 13.07.2023— Susanne Donner
Viele tausend Schadstoffe befinden sich in Luft, Wasser, Boden und Nahrung und Milliarden Menschen sind diesen ausgesetzt. Schadstoffe können dem Gehirn zusetzen, Stress, Entzündung und Zelltod auslösen. Am Lebensanfang, wenn das Gehirn sich entwickelt und am Lebensende, wenn natürliche Abbauvorgänge einsetzen, ist es besonders empfindlich für Umwelteinflüsse. Luftverschmutzung, besonders die Belastung mit Feinstaub, trägt zu kognitiven Defiziten am Lebensanfang und zu Demenzen am Lebensende bei. Die Chemikerin und Wissenschaftsjournalistin Susanne Donner berichtet über die ernste Bedrohung der globalen Gesundheit, die mit der zunehmenden Menge an Schadstoffen einhergeht.*
Auch an sonnigen Tagen ist Mexiko-Stadt in einen weiß-bräunlichen Dunst gehüllt, den die 21 Millionen Bewohner nur sehen können, wenn sie sich aus dem Ballungsraum entfernen und auf ihre Stadt hinunterschauen. Wer sich dagegen in der Innenstadt aufhält, bemerkt nur eines: Haut und Kleidung sind nach einem Tagesausflug vom Zócalo bis zur barocken Kathedrale mit einem bräunlichen Schleier überzogen.
Die Kinderärztin und Neurowissenschaftlerin Lilian Calderón-Garcidueñas kennt das zur Genüge, da sie sowohl in Mexiko-Stadt als auch in den USA arbeitet. Schon lange hegte sie den Verdacht, dass die feinstaubschwangere Luft Kindern schadet. Mit einer Untersuchung wollte sie endlich für Klarheit sorgen: Sie verglich die geistigen Fähigkeiten von 55 Kindern aus der Hauptstadt mit denen aus einer Kleinstadt des Landes. Und tatsächlich schnitten die Großstadtsprösslinge in den Tests deutlich schlechter ab. In Magnetresonanztomografie-Aufnahmen des Gehirns entdeckte Calderón-Garcidueñas bei 56 Prozent der Kinder Entzündungsherde im präfrontalen Cortex. Hier, im Frontallappen, werden Sinneswahrnehmungen zusammengeführt. Er spielt deshalb eine Schlüsselrolle beim Planen von Handlungen, beim Entscheiden, und auch beim Lernen.
Verursacht die schmutzige Luft die Entzündungen im Gehirn und die kognitiven Defizite? Auf der Suche nach einer Antwort untersuchte Calderón-Garcidueñas das Gehirn von sieben jungen Hunden aus Mexiko-Stadt. Und tatsächlich fand sie auch hier Entzündungsherde bei vier Tieren. Mehr noch: Sie konnte Ablagerungen von Feinstaub nachweisen und eine abnorme Aktivierung von Mikroglia, den Immunzellen des Gehirns. „Die Luftverschmutzung beeinträchtigt das reifende Gehirn, sie erklärt kognitive Defizite in gesunden Kindern“, warnte die Kinderärztin 2008 in einer viel beachteten Veröffentlichung im Journal Brain and Cognition [1].
Seither hat Mexico-Stadt Forschende wegen der brisanten Frage angezogen, ob schlechte Luft das Gehirn zerstört und wenn ja, auf welche Weise und wie sehr. Am Lebensanfang und am ende könnte das Organ besonders verletzlich reagieren, weil es erst noch reift und später auf natürliche Weise abbaut.
Luftverschmutzung und Demenzen treten zusammen auf
Obwohl es oft heißt, neurodegenerative Erkrankungen, zuvorderst die Demenzen, seien eine Unausweichlichkeit der enormen Lebenserwartung in modernen Zivilisationen, liefern mehr und mehr epidemiologische Studien ein brisantes Gegenargument: Je höher die Feinstaubbelastung der Luft, desto mehr häufen sich Demenzen. Daten, die diesen Zusammenhang unterfüttern, kommen aus London, aus Quebec, aus Nordschweden aus Stockholm: Dort hatten Forschende des Karolinska-Instituts im Jahr 2020 etwa 3.000 Erwachsene mit einem Durchschnittsalter von 74 Jahren begleitet. 364 entwickelten innerhalb von 11 Jahren eine Demenz. Und obwohl die Luft der schwedischen Hauptstadt als sauber gilt, fand die Neurobiologin des Instituts Giulia Grande, dass die Gehalte an Feinstaub in der Luft in Wohnortnähe das Risiko einer Demenz beeinflussten [2]. Ungefähr 800.000 Fälle an Demenzen in Europa könnten jedes Jahr auf die Luftverschmutzung zurückgehen, rechneten andere Forscher 2021 hoch.
„Diese Befunde sind sehr unpopulär“, bedauert Bruce Lanphear, Gesundheitswissenschaftler an der kanadischen Simon Fraser University. „Wir sollen lieber an einer Therapie gegen Alzheimer forschen, weil man damit Geld verdienen kann, als den Ursachen auf den Grund zu gehen. Denn, wenn die Luftverschmutzung schuld ist, würde das ein Absenken der Grenzwerte erfordern. Das wollen weder Politiker noch Industrie gern hören.“ Abbildung 1.
| Abbildung 1. Luftverschmutzung - ein globales Problem |
Blei raubt die Intelligenz
Lanphear widmet sich seit vielen Jahren einem der potentesten Schadstoffe für das Gehirn: dem Blei. Das Schwermetall ist auch im Feinstaub vertreten, weil es etwa im Flugbenzin enthalten ist und damit aus den Triebwerken gewirbelt wird. Vor allem aber ist es eine Substanz, von der man gesichert weiß, wie gefährlich sie für den Menschen ist, weil sie seit vielen Jahrhunderten eingesetzt wird. Und noch heute enthalten bestimmte Gläser und Kunststoffe Blei, auch Knöpfe an Bekleidung, Farben und Stifte, teils sogar Modeschmuck. In verschiedenen Pkw-Kraftstoffen ist Blei zwar EU-weit seit der Jahrtausendwende verboten. Genauer gesagt heißt das aber, dass die erlaubten Mengen lediglich gering sind.
Überall auf der Welt empören sich Toxikologen und Toxikologinnen, die an Blei forschen, wie Lanphear oder die international renommierte Kinderärztin und Umweltmedizinerin Ruth Etzel aus den USA, dass Blei bis heute in vielen Anwendungen nicht verboten ist. Die Belastungen von Kindern und Kleinkindern auch in Deutschland sind nach wie vor zu hoch, warnt das staatliche Bundesinstitut für Risikobewertung.
Nachgewiesen ist nämlich, dass das Schwermetall toxisch auf Nervenzellen wirkt. Es schädigt insbesondere die Entwicklung des reifenden Gehirns. Wenn die Menge an Blei von weniger als einem Mikrogramm je Deziliter Blut auf zehn Mikrogramm je Deziliter steigt, sinkt der IQ der Kinder um 6,9 Punkte, ermittelte Lanphear anhand der Daten an 1.300 Kindern. Andere Studien bestätigten, dass Blei die Intelligenz schmälert. Kleinkinder zwischen null und drei Jahren verleiben sich aber hierzulande 1,1 bis 3,3 Mikrogramm Blei täglich über Essen und Trinken ein – die Atemluft ist da noch nicht einmal berücksichtigt. „Es ist eine Tragödie. Wir wissen bei Blei so gut wie bei kaum einer anderen Chemikalie, wie schädlich es ist. Aber die Regierungen haben die Belastungen nicht auf ein akzeptables Maß gesenkt“, urteilt Etzel.
„Das Problem ist“, sagt Lanphear, „fünf IQ-Punkte weniger mögen auf der individuellen Ebene keine große Rolle spielen. Aber auf der Bevölkerungsebene bedeutet das riesige Effekte, wenn ganze Populationen durchschnittlich etwa fünf IQ-Punkte verlieren.“ Der Anteil der Personen mit kognitiven Beeinträchtigungen, die Hilfe oder besondere Förderung brauchen, steigt gewaltig und der Anteil der Hochbegabten schwindet messbar.
Schadstoffbedingte Demenz?
"Das kognitive Defizit am Lebensanfang wird zudem nie wieder aufgeholt“, argumentiert Lanphear in einer Linie mit seinen Forschungskollegen. Viele vermuten, dass die Last an Blei im Körper auch eine spätere Demenz begünstigt. Diese könnte umso früher einsetzen, je mehr des Schwermetalls gespeichert ist. Und weil sich die Indizien in dieser Richtung verdichten, ergründet hierzulande neuerdings auch das Umweltbundesamt, wie Schadstoffe neurodegenerative Krankheiten befeuern.
„Seit etwa dreißig Jahren sinkt die Inzidenz an Demenzen in den USA – auch wenn die Gesamtzahl aufgrund der Überalterung weiter stark steigt. Im selben Zeitraum gingen die Bleibelastungen zurück, weil wir als erstes Land weltweit bleihaltiges Benzin verboten haben. Ich sehe da einen direkten Zusammenhang: sinkende, gleichwohl immer noch zu hohe Bleibelastungen auf der einen Seite und zurückgehende Inzidenzen bei Demenzen auf der anderen Seite“, sagt Lanphear.
Im Gehirn unterbindet Blei die Erregungsleitung zwischen den Nervenzellen. Es erschwert, dass sich Synapsen ausbilden und verstärken. Insgesamt stört das Schwermetall somit das Oberstübchen fundamental beim Denken, Fühlen und Handeln. „Blei ist aber nicht der einzige Schadstoff, der dem Gehirn schadet“, sagt der Neurowissenschaftler Stephen Bondy von der University of California in Irvine. Polychlorierte Biphenyle, die schon in Fahrradlenkern und Sportplätzen gefunden wurden, Aluminium und Kupfer sind nur drei weitere Verdächtige. Bondy selbst gab einem Mäusestamm aluminiumhaltiges Wasser zu trinken, wie es in Kanada typischerweise aus dem Wasserhahn kommt. Die Tiere entwickelten daraufhin typische Alzheimer-Veränderungen im Gehirn, berichtet er. Mit Kupfer beschäftigt sich eine weltumspannende Forschungsszene, die zeigen konnte, dass das Metall die Fehlfaltung von Proteinen anstachelt. Unter dem Einfluss von Kupfer entstünden aus normalen Proteinen „Ringe des Bösen“, schilderten jüngst Forscher der schweizerischen Eidgenössischen Materialprüfungs- und Forschungsanstalt.
Zu viel Verschmutzung, um zu erforschen, was sie macht
Viele tausend Schadstoffe zirkulieren in Luft, Boden und Wasser. „Wenn ein Stoff irgendwo verboten wird, kommt oft ein neuer auf den Markt, von dem wir erst einmal nichts wissen“, klagt Bondy. Jede Forschungsgruppe muss vor der Vielfalt kapitulieren und richtet notgedrungen ihr Augenmerk auf ausgewählte Stoffe. Unter den Pestiziden, Weichmachern und Flammschutzmitteln sind viele Kandidaten mit kryptischen Namen, die Nervenzellen in Kultur oder auf Ebene der Genexpression beeinträchtigen. Sie könnten an den Geistesgaben zehren und Autismus, Alzheimer und ADHS den Weg bereiten.
Beispielsweise entdeckte das Team um Barbara Maher von der Universität Lancaster im Hirnstamm von 186 jungen Bewohnern in Mexiko-Stadt bestimmte fehlgefaltete Proteine. Sie deutete diese als frühe Vorboten häufiger Altersleiden, etwa einer Demenz oder der Parkinson’schen Erkrankung. In den geschädigten Hirnstämmen konnte Maher unter den Feinstaubablagerungen vornehmlich Manganpartikel nachweisen, vermutlich aus dem Straßenverkehr, und nanoskalige Titanpartikel. Letztere sind Bestandteil des umstrittenen Lebensmittelzusatzstoffes E 171. Sie könnten aus Süßigkeiten oder Zahncreme stammen und vom Darm ins Gehirn vordringen, vermutet Maher.
Schleichwege ins Gehirn
Das führt zur grundlegenden Frage, wie Schadstoffe überhaupt in das gut geschützte Gehirn vordringen können. Die Blut-Hirnschranke bildet schließlich eine Barriere zwischen dem Blut und der Hirnsubstanz. Die feinen Blutgefäße sind im Nervengewebe dicht mit Endothelzellen ausgekleidet, die eng miteinander über sogenannte „tight junctions“ verknüpft sind. Sie verhindern, dass Substanzen leichterdings ins Nervengewebe übertreten können. Die Gefäße sind zudem von Astrozyten umgeben, die ebenfalls die Passage von gefährlichen Stoffen kontrollieren.
Und doch können Medikamente, Substanzen aus Zigaretten und eben auch verschiedene Schadstoffe an dieser Einlasskontrolle vorbei aus dem Blut ins Gehirn gelangen. Besonders fettliebende kleine Moleküle haben es leicht. Und feste Partikel wie Blei können eine Abkürzung nehmen. Über die Nase steigen sie in den Riechkolben auf und werden von dort, wenn sie nur klein genug sind, direkt ins Gehirn verfrachtet. Eingeatmete Nanopartikel lassen sich so in Tieren und mit optischen Spezialverfahren neuerdings auch im Gehirn des Menschen nachweisen. Barbara Mahers Team machte zum ersten Mal die Mangan- und Titanteilchen im Hirnstamm der 186 Probanden sichtbar.
Abbildung 2 (von Redn. eingefügt) fasst zusammen, wie Schadstoffe ins Hirn gelangen und welche Auswirkungen sie dort zeigen.
| Abbildung 2. Schematische Zusammenfassung: Feinstaub kann in das Gehirn eindringen und dort neurotoxische Wirkungen hervorrufen, was mit einem erhöhten Risiko der Neurodegeneration verbunden ist. Feinstaubpartikel können über das Riechsystem oder den Trigeminusnerv ins Gehirn gelangen, oder sie passieren die Blut-Hirnschranke (BBB), nachdem sie in den Blutkreislauf gelangt sind. Die abgelagerten Feinstaubpartikel können Neurotoxizität, oxidativen Stress, Neuroinflammation sowie Schäden an der BBB und dem neurovaskulären System verursachen. Diese Auswirkungen im Gehirn können zu neurodegenerativen Erkrankungen führen. Epidemiologische Studien haben die Belastung durch Feinstaub mit kognitiven Beeinträchtigungen und einem erhöhten Risiko für die Entwicklung neurodegenerativer Erkrankungen wie Alzheimer (AD) in Verbindung gebracht. (Abbildung und Legende von Redn. in modifizierter (übersetzter) Form eingefügt aus You, R. et al (2022) [3] Lizenz: cc-by.) |
Zahlreiche Schadstoffe, die mit der Nahrung aufgenommen werden, treffen im Darm auf eine Gemeinschaft Hunderter verschiedener Arten von Mikroorganismen. Wenn dieses „Mikrobiom“ gestört wird, kann dies die „Entsorgung“ beeinträchtigen und den Weitertransport der Schadstoffe ins Blut und dann ins Gehirn verstärken. Und ausgerechnet die westliche Ernährungsweise derangiert die Flora der 38 Billionen Winzlinge im Darm empfindlich. Morgens Croissant und Weißbrot mit Schinken, abends Pizza und Chips lässt die Vielfalt im Darm schrumpfen und macht ihn schlimmstenfalls durchlässig. Vom „leaky gut“, dem durchlässigen Darm, sprechen Forschende.
Schadstoffe im Gehirn sorgen für Entzündungen und Stress
Feste Partikel wie Blei erinnern das Immunsystem des Gehirns, die Mikroglia, an eingedrungene Bakterien. t„Sie werden aktiviert, können den Partikel aber weder „töten“ noch abtransportieren. Sie wissen nicht, was sie tun sollen und kämpfen weiter und weiter. Es kommt zur chronischen Entzündung, die sehr schädlich ist“, veranschaulicht der Neurowissenschaftler Stephen Bondy von der University of California in Irvine. Noch dazu können gerade Metalle wie Aluminium und Kupfer die Fehlfaltung von Proteinen katalysieren. An ihrer Oberfläche entstehen auch reaktive Sauerstoffspezies, die umliegende Zellen attackieren.
Viele Fachleute vermuten auch, dass Schadstoffe dem Gehirn indirekt schaden, indem sie Herz und Kreislauf schwächen. Bekanntlich zieht die Feinstaubbelastung der Luft Schlaganfälle, Herzinfarkte und Gefäßschädigungen nach sich. Und wenn in der Folge Organe schlechter mit Blut und Nährstoffen versorgt werden, leiden auch diese. Guilia Grande, die Neurobiologin am Karolinska Institut, glaubt die Herz-Kreislaufgesundheit sei der entscheidende Mittler, weshalb schlechtere Luft mehr Demenzen nach sich zieht.
Die Effekte von Schadstoffen auf das Gehirn merkt man erst einmal nicht, gibt Bondy zu bedenken. „Wir müssen nicht husten und nach Luft schnappen. Aber sie sind irreversibel und heimtückisch.“ Vor diesem Hintergrund kann er nur raten, viel hinaus an die frische Luft zu gehen. Das mag erstaunen, fahren doch genau dort die Autos, die Feinstaub produzieren. Aber drinnen ist die Luft noch einmal um Größenordnungen schlechter, weil aus zahllosen Konsumgütern Schadstoffe ausdünsten.
[1]. Calderón-Garcidueñas, L. et al.: Air pollution, cognitive deficits and brain abnormalities: a pilot study with children and dogs. Brain and Cognition, 2008, 68. https://pubmed.ncbi.nlm.nih.gov/18550243/
[2]. Wu, J. et al., Air pollution as a risk factor for Cognitive Impairment no Dementia (CIND) and its progression to dementia: A longitudinal study. Environment International 160 (2022) 107067. https://doi.org/10.1016/j.envint.2021.107067
[3]. You, R. et al.: The pathogenic effects of particulate matter on neurodegeneration: a review. Journal of Biomedical Science, 2022, 29 (25),https://pubmed.ncbi.nlm.nih.gov/35189880/
----------------------------------------------------
Zum Weiterlesen
Oudin, A. et al.: Association between air pollution from residential wood burning and dementia incidence in a longitudinal study in Northern Sweden. PLoS ONE, 2018, 13(6) https://pubmed.ncbi.nlm.nih.gov/29897947/
Smargiassi, A. et al.: Exposure to ambient air pollutants and the onset of dementia in Quebec Canada. Environmental Research, 2020, 190 (109870) ( zum Abstract: https://pubmed.ncbi.nlm.nih.gov/32739624/ )
Lanphear, B.: The Impact of Toxins on the Developing Brain. Annual Reviews of Public Health 2015, 36 (211–30) ( zum Abstract: https://pubmed.ncbi.nlm.nih.gov/25581143/ )
*Der Artikel ist erstmals am 2.Mai 2023 unter dem Titel "Dauerstress für die Grauen Zellen" auf der Website https://www.dasgehirn.info/ erschienen https://www.dasgehirn.info/krankheiten/eindringlinge/dauerstress-fuer-die-grauen-zellen . Der unter einer CC-BY-NC-SA Lizenz stehende Text wurde unverändert in den Blog gestellt, eine Abbildung aus der im Text zitierten Arbeit von You et al., (2022) [3] wurde von der Redaktion in modifizierter Form eingefügt.
Die Webseite https://www.dasgehirn.info/ ist eine exzellente Plattform mit dem Ziel "das Gehirn, seine Funktionen und seine Bedeutung für unser Fühlen, Denken und Handeln darzustellen – umfassend, verständlich, attraktiv und anschaulich in Wort, Bild und Ton." (Es ist ein Projekt der Gemeinnützigen Hertie-Stiftung und der Neurowissenschaftlichen Gesellschaft e.V. in Zusammenarbeit mit dem Zentrum für Kunst und Medientechnologie Karlsruhe).
Artikel im ScienceBlog:
Inge Schuster, 25.09.2019: Pflanzen entfernen Luftschadstoffe in Innenräumen
IIASA, 18.05.2017: Überschreitungen von Diesel-Emissionen — Auswirkungen auf die globale Gesundheit und Umwelt
IIASA, 25.09.2015: Verringerung kurzlebiger Schadstoffe – davon profitieren Luftqualität und Klima
Johannes Kaiser & Angelika Heil, 31.07.2015: Feuer und Rauch: mit Satellitenaugen beobachtet
Inge Schuster, 12.12.2014: Was macht HCB so gefährlich?
Der Global Migration Data Explorer - ein neues Instrument zur Visualisierung globaler Migrationsströme
Der Global Migration Data Explorer - ein neues Instrument zur Visualisierung globaler MigrationsströmeDo, 06.07.2023— IIASA

![]() Wie viele Menschen sind in den letzten 30 Jahren in ein Land eingewandert, von wo sind sie gekommen und wie viele haben das Land verlassen und wohin sind sie gezogen? Mit dem Global Migration Data Explorer haben IASA-Forscher ein neues Instrument entwickelt, das den Mangel an Daten über globale Migrationsströme beheben und eine visuelle Methode zur Erforschung der weltweiten Migrationsmuster bereitstellen soll.*
Wie viele Menschen sind in den letzten 30 Jahren in ein Land eingewandert, von wo sind sie gekommen und wie viele haben das Land verlassen und wohin sind sie gezogen? Mit dem Global Migration Data Explorer haben IASA-Forscher ein neues Instrument entwickelt, das den Mangel an Daten über globale Migrationsströme beheben und eine visuelle Methode zur Erforschung der weltweiten Migrationsmuster bereitstellen soll.*
Der Global Migration Data Explorer [1] baut auf dem Erfolg seines Vorgängers [2] auf. Er inkludiert nun Schätzungen aus Zeiträumen bis 2020, die auf weiterentwickelten Schätzmethoden beruhen, und erweitert den Anwendungsbereich, um verschiedene Messungen von Migration und Aufschlüsselungen der Migrationsmuster nach Geschlecht einzubeziehen.
Die Website wurde von Guy Abel, einem Wissenschafter in der Forschungsgruppe Migration and Sustainable Development des IIASA- Population and Just Societies Program und der Universität Shanghai, und Xavier Bolló, einem Spezialisten für Datenvisualisierung, entwickelt. Es bietet den Nutzern eine einzigartige Möglichkeit, sich mit der komplexen Dynamik der globalen Migration zu beschäftigen. Die Webseite stellt sechs verschiedene Schätzmethoden vor, die Forscher nutzen können, um Einblicke in die Migrationsströme zu gewinnen. Solche Methoden der Abschätzung sind unentbehrlich, da es kaum verlässliche Daten über internationale Migrationsströme gibt, und dies die Messung von Mustern und Trends in den globalen Migrationsströmen erschwert.
"Die internationale Migration wird zu einer immer wichtigeren Komponente des Bevölkerungswachstums und zu einer Triebkraft des sozioökonomischen Wandels", erklärt Abel. "Gute Daten über die internationale Migration sind entscheidend, um migrationsbezogene Komponenten der internationalen Entwicklungsagenda und Vereinbarungen wie die Ziele für nachhaltige Entwicklung und den Globalen Pakt für sichere, geordnete und reguläre Migration zu überwachen . Diese Website füllt eine entscheidende Lücke, indem sie Schätzungen der Migrationsströme bereitstellt, welche für Forscher, die sich mit Migrationssystemen, Demografie, Klimawandel und Epidemiologie beschäftigen, von unschätzbarem Wert sind."
Die frei zugängliche Website [2] bietet eine intuitive und interaktive Plattform, die es jedermann möglich macht globale Migrationsmuster zu untersuchen. Der von Bolló entwickelte Code für die Darstellung der Daten ist auf GitHub verfügbar, was Transparenz und weiterführende Zusammenarbeit fördert. Die Nutzer können auf die Daten, die den Visualisierungen zugrunde liegen, zugreifen, und diese sind über die Website frei zugänglich. Ein Beispiel (von der Redaktion eingefügt) zeigt die globalen Migrationsströme der letzten 30 Jahre. Abbildung 1. Mit einem Klick auf eine bestimmte Erdregion - beispielsweise Europa - wird das dortige Ausmaß der Migration - woher kommen die Migranten und wohin gehen sie - in Zeiträumen von 5 Jahren /oder insgesamt in den letzten 30 Jahren angezeigt.
| Abbildung 1. Der Global Migration Data Explorer. Links: Migrationsströme im Zeitraum 1990 bis 2020; nach Europa sind rund 68,9 Millionen Menschen eingewandert und rund 31,1 Millionen abgewandert. Rechts: im Zeitraum 2015 bis 2020 sind rund 15 Millionen Migranten nach Europa gekommen und 8,6 Millionen sind abgewandert. Ein Klick auf Europa (oder andere Gegenden) zeigt die einzelnen Staaten und deren Migrationsmuster in den verschiedenen Zeiträumen (nicht dargestellt). https://global-migration.iiasa.ac.at/index.html (Bild von Redn. eingefügt.). |
In das Tool fließen die UN-Schätzungen der im Ausland geborenen Bevölkerung ein - diese gelten als zuverlässigste Datenquelle für die globale Migration. Während die UN-Daten eine Momentaufnahme der Migrantenbevölkerung zu einem bestimmten Zeitpunkt darstellen, bieten die Schätzungen der Migrationsströme eine zeitliche Messung der Migration; dies macht sie für Forscher, die die Ursachen und Folgen der Migration untersuchen, wichtiger. (Beispiel: Migration von und nach Europa von Redn. eingefügt: Abbildung 2.)
| Abbildung 2. Migration von und nach Europa im Zeitraum von 1990 bis 2020. Diagramm mit den Daten aus dem Global Migration Data Explorer (https://global-migration.iiasa.ac.at/ von Redn. erstellt. |
Das Team plant das Visualisierungstool zu erweitern, um die Binnenmigration in verschiedenen Ländern, wie z. B. China, zu erfassen. Darüber hinaus will man zusätzliche Visualisierungen einführen, einschließlich einer kartenbasierten Methode, um die Benutzererfahrung zu verbessern und ein tieferes Verständnis der Migrationsmuster zu ermöglichen.
"Wir hoffen, dass dieses Visualisierungstool von der Öffentlichkeit genutzt wird, um zu einem besseren Verständnis von Migrationsmustern und relativen Ausmaßen der Migration in und aus einem bestimmten Land zu gelangen", sagt Anne Goujon, IIASA Population and Just Societies Program Director. "Besonders erfreulich ist es, wenn man hört, dass Lehrer ihr Interesse bekundet haben, die Website für den Unterricht ihrer Schüler zu nutzen; dies unterstreicht den Bedarf an aktuellen Daten und die Anwendbarkeit des Tools über den akademischen Bereich hinaus".
Zwei wichtige Veröffentlichungen unterstützen die Visualisierungs- und Schätzungsmethoden der Website. Ein 2019 im Fachjournal Nature veröffentlichter Artikel vergleicht die sechs wichtigsten Schätzmethoden, die für die Schätzung der globalen Migration vorgeschlagen wurden [3]. Außerdem wird eine Reihe von Validierungstests vorgestellt, mit denen die Genauigkeit dieser Schätzungen durch den Vergleich mit gemeldeten Migrationsdaten (dies vor allem aus wohlhabenden westlichen Ländern) bewertet wird. Darüber hinaus konzentriert sich eine neuere Veröffentlichung auf die Erstellung von Schätzungen der globalen Migrationsströme nach Geschlecht und weitet die Validierungsübungen auf geschlechtsspezifische Daten aus [4].
[1] Guy J. Abel & Xavier Bolló: Global Migration Data Explorer. Explore changes in global migrant flow patterns over the past 30 years. . https://global-migration.iiasa.ac.at/stocks.html
[2] The global flow of people. Explore new estimates of migration flows between and within regions for five-year periods, 1990 to 2010. Click on a region to discover flows country-by-country. http://download.gsb.bund.de/BIB/global_flow/
[3] Abel, G.J., Cohen, J.E. Bilateral international migration flow estimates for 200 countries. Sci Data 6, 82 (2019). https://doi.org/10.1038/s41597-019-0089-3
[4] Abel, G.J., Cohen, J.E. Bilateral international migration flow estimates updated and refined by sex. Sci Data 9, 173 (2022). https://doi.org/10.1038/s41597-022-01271-z
*Der Blogartikel basiert auf der IIASA-Presseaussendung “ New IIASA online tool to visualize global migration patterns“ vom 5. Juli 2023. Diese wurde von der Redaktion aus dem Englischen übersetzt, geringfügig für den Blog adaptiert und mit Texten und Abbildungen aus der Webseite https://global-migration.iiasa.ac.at/ (oder aus deren Daten zusammengestellt) versehen. IIASA ist freundlicherweise mit Übersetzung und Veröffentlichung seiner Nachrichten in unserem Blog einverstanden.
Artikel im ScienceBlog
- IIASA, 06.06.2019: Ist Migration eine demographische Notwendigkeit für Europa?
- Inge Schuster, 10.08.2017:Migration und naturwissenschaftliche Bildung
- IIASA, 17.5.2018: Die Bevölkerungsentwicklung im 21. Jahrhundert - Szenarien, die Migration, Fertilität, Sterblichkeit, Bildung und Erwerbsbeteiligung berücksichtigen
- IIASA. 9.9.2016: Wie sich Europas Bevölkerung ändert - das "Europäische Demographische Datenblatt 2016"
Fundamentales Kohlenstoffmolekül vom JW-Weltraumteleskop im Orion-Nebel entdeckt
Fundamentales Kohlenstoffmolekül vom JW-Weltraumteleskop im Orion-Nebel entdecktDo, 29.06.2023 — Redaktion
Die Kohlenstoffchemie ist für die Astronomen von zentralem Interesse, da alles bekannte Leben auf Kohlenstoff basiert. Ein internationales Forscherteam hat nun mit Hilfe des James Webb Weltraumteleskops zum ersten Mal ein als Methylkation (CH3+) bekanntes Molekül in der protoplanetaren Scheibe um einen jungen Stern nachgewiesen. Dieses einfache Molekül hat eine einzigartige Eigenschaft: Es reagiert relativ ineffizient mit Wasserstoff, dem am häufigsten vorkommenden Element in unserem Universum, aber leicht mit anderen Molekülen und initiiert so das Wachstum komplexerer Moleküle auf Kohlenstoffbasis. Die entscheidende Rolle von CH3+ in der interstellaren Kohlenstoffchemie wurde bereits in den 1970er Jahren vorhergesagt, aber die einzigartigen Fähigkeiten von Webb haben es endlich möglich gemacht, es zu beobachten - in einer Region des Weltraums, in der sich möglicherweise Planeten bilden könnten, die Leben beherbergen.*
Das Methylkation
Kohlenstoffverbindungen bilden die Grundlage allen bekannten Lebens und sind daher von besonderem Interesse für Wissenschaftler, die verstehen wollen, wie sich das Leben auf der Erde entwickelt hat und wie es sich möglicherweise anderswo in unserem Universum entwickeln könnte. Die interstellare organische (d.i. Kohlenstoff-basierte) Chemie ist daher ein Gebiet, das für Astronomen, die Orte untersuchen, an denen sich neue Sterne und Planeten bilden, besonders faszinierend ist. Kohlenstoffhaltige Molekülionen (d.i. Moleküle mit positiver oder negativer elektrischer Ladung) sind von besonderer Bedeutung, da sie mit anderen kleinen Molekülen reagieren und selbst bei niedrigen interstellaren Temperaturen komplexere organische Verbindungen bilden. Ein solches kohlenstoffhaltiges Ion ist das Methylkation (CH3+). Abbildung 1.
| Abbildung 1. Das Methylkation. Das positiv geladene Ion reagiert kaum mit Wasserstoff, dem am häufigsten vorkommenden Molekül im Universum, dagegen effizient mit einer Vielzahl anderer Moleküle. CH3+ wird als seit langem als wichtiger Baustein der interstellaren organischen Chemie betrachtet. (Bild von Redn. eingefügt.). |
Bereits seit den 1970er Jahren postulieren Wissenschaftler die außerordentliche Wichtigkeit von CH3+, da dieses Ion mit einer Vielzahl anderer Moleküle reagiert. Dieses kleine Kation ist so bedeutsam, dass es als Eckpfeiler der interstellaren organischen Chemie gilt; bislang konnte es allerdings noch nie nachgewiesen werden.
Das James-Webb-Weltraumteleskop mit seinen einzigartigen Eigenschaften bot sich als ideales Instrument an, um nach diesem wesentlichen Kation zu suchen - und tatsächlich konnte es eine Gruppe internationaler Wissenschaftler mit Webb zum ersten Mal beobachten. Marie-Aline Martin von der Universität Paris-Saclay, Frankreich, eine Spektroskopikerin und Mitglied des Wissenschaftsteams, erklärt: "Dieser Nachweis von CH3+ bestätigt nicht nur die unglaubliche Empfindlichkeit von James Webb, sondern auch die postulierte zentrale Bedeutung von CH3+ in der interstellaren Chemie."
CH3+-Signal in protoplanetarer Scheibe entdeckt
Das CH3+-Signal wurde in dem als d203-506 bekannten System aus Stern und protoplanetarer Scheibe (d.i. eine rotierende Scheibe aus Gas und Staub, die sich um junge Sterne bildet und aus der sich schließlich Planeten bilden können) entdeckt, das sich in etwa 1350 Lichtjahren Entfernung im Orionnebel befindet. Abbildung 2. Der Stern in d203-506 ist zwar ein kleiner roter Zwerg mit einer Masse von nur etwa einem Zehntel der Sonnenmasse, doch das System wird von starker ultravioletter Strahlung benachbarter heißer, junger und massereicher Sterne bombardiert. Wissenschaftler gehen davon aus, dass die meisten protoplanetaren Scheiben, aus denen sich Planeten bilden, eine Periode solch intensiver ultravioletter Strahlung durchlaufen, da Sterne dazu neigen, sich in Gruppen zu bilden, die oft massereiche, ultraviolett-produzierende Sterne umfassen. Faszinierenderweise deuten Hinweise aus Meteoriten darauf hin, dass die protoplanetare Scheibe, aus der sich unser Sonnensystem bildete, ebenfalls einer enormen Menge an ultravioletter Strahlung ausgesetzt war - ausgesandt von einem stellaren Begleiter unserer Sonne, der schon lange gestorben ist (massereiche Sterne brennen hell und sterben viel schneller als weniger massereiche Sterne). Der verwirrende Faktor bei all dem ist, dass ultraviolette Strahlung lange Zeit als rein zerstörerisch für die Bildung komplexer organischer Moleküle angesehen wurde - und doch gibt es eindeutige Beweise dafür, dass der einzige uns bekannte lebensfreundliche Planet aus einer Scheibe entstanden ist, die dieser Strahlung stark ausgesetzt war.
| Abbildung 2 Die Webb-Bilder zeigen einen Teil des Orionnebels, der als Orion-Balken bekannt ist. Links: Aufnahme mit dem Webb-NIRCam-Instrument (Nahinfrarotkamera). Rechts oben: Teleskop mit dem Webb-MIRI (Mid-Infrared Instrument) ist auf den angezeigten kleineren Bereich fokussiert. Im Zentrum des MIRI-Bereichs befindet sich ein junges Sternsystem mit einer protoplanetaren Scheibe namens d203-506. Rechts unten: Der Ausschnitt zeigt ein kombiniertes NIRCam- und MIRI-Bild dieses jungen Systems. (Credits: ESA/Webb, NASA, CSA, M. Zamani (ESA/Webb), und das PDRs4All ERS-Team.) |
Das Team, das diese Forschung durchgeführt hat, könnte die Lösung für dieses Rätsel gefunden haben. Ihre Arbeit sagt voraus, dass das Vorhandensein von CH3+ tatsächlich mit ultravioletter Strahlung zusammenhängt, welche die notwendige Energiequelle für die Bildung von CH3+ darstellt. Außerdem scheint die Dauer der ultravioletten Strahlung, der bestimmte Scheiben ausgesetzt sind, einen tiefgreifenden Einfluss auf deren Chemie zu haben. So zeigen Webb-Beobachtungen von protoplanetaren Scheiben, die keiner intensiven ultravioletten Strahlung aus einer nahe gelegenen Quelle ausgesetzt sind, eine große Menge an Wasser - im Gegensatz zu d203-506, wo das Team überhaupt kein Wasser nachweisen konnte. Der Hauptautor Olivier Berné von der Universität Toulouse, Frankreich, erklärt: "Dies zeigt deutlich, dass ultraviolette Strahlung die Chemie einer proto-planetaren Scheibe völlig verändern kann. Sie könnte tatsächlich eine entscheidende Rolle in den frühen chemischen Stadien der Entstehung von Leben spielen, indem sie dazu beiträgt, CH3+ zu produzieren - etwas, das bisher vielleicht unterschätzt wurde."
Obwohl bereits in den 1970er Jahren veröffentlichte Forschungsergebnisse die Bedeutung von CH3+ vorhersagten, war es bisher praktisch unmöglich, das Molekül nachzuweisen. Viele Moleküle in protoplanetaren Scheiben werden mit Radioteleskopen beobachtet. Damit dies möglich ist, müssen die betreffenden Moleküle jedoch ein so genanntes "permanentes Dipolmoment" besitzen, d. h. die Geometrie des Moleküls muss so beschaffen sein, dass seine elektrische Ladung nicht ausgeglichen ist, das Molekül also ein positives und ein negatives "Ende" hat. Da CH3+ symmetrisch ist, ist seine Ladung ausgeglichen und hat somit kein permanentes Dipolmoment, das für Beobachtungen mit Radioteleskopen erforderlich ist. Theoretisch ist es möglich, die von CH3+ emittierten spektroskopischen Linien im Infrarotbereichzu beobachten, aber die Erdatmosphäre macht es praktisch unmöglich, diese von der Erde aus zu beobachten. Daher musste ein ausreichend empfindliches Weltraumteleskop eingesetzt werden, das Signale im Infraroten beobachten kann. Die Instrumente MIRI und NIRSpec von Webb waren für diese Aufgabe wie geschaffen. Tatsächlich war der Nachweis von CH3+ zuvor sehr schwierig und als das Team das Signal zum ersten Mal in seinen Daten entdeckte, war es nicht sicher, wie es zu identifizieren sei. Gestützt auf die Hilfe eines internationalen Teams mit einem breiten Spektrum an Fachwissen waren die Forscher aber in der Lage ihre Ergebnisse innerhalb von vier kurzen Wochen zu interpretieren.
Die Entdeckung von CH3+ war nur durch die Zusammenarbeit von beobachtenden Astronomen, astrochemischen Modellierern, Theoretikern und experimentellen Spektroskopikern möglich, welche die einzigartigen Möglichkeiten von JWST im Weltraum mit denen von Labors auf der Erde kombinierten, um die Zusammensetzung und Evolution unseres lokalen Universums erfolgreich zu untersuchen und zu interpretieren. Marie-Aline Martin fügt hinzu: "Unsere Entdeckung war nur möglich, weil sich Astronomen, Modellierer und Laborspektroskopiker zusammengetan haben, um die von James Webb beobachteten einzigartigen Merkmale zu verstehen."
*Der vorliegende Artikel ist unter dem Titel " Webb makes first detection of crucial carbon molecule in a planet-forming disc" am 26.Juni 2023 auf der Webseite der ESA erschienen:https://esawebb.org/news/weic2315/. Der Artikel wurde von der Redaktion möglichst wortgetreu übersetzt und durch die Strukturformel des Methylkations ergänzt.
ESA Webb Bilder, Texte und Videos stehen unter einer cc-by 4.0 Lizenz.
Webb ist eine internationale Partnerschaft zwischen der NASA, der ESA und der kanadischen Weltraumbehörde (CSA).
Webb ist das größte und leistungsstärkste Teleskop, das jemals ins All gebracht wurde. Im Rahmen eines internationalen Kooperationsabkommens stellte die ESA den Startdienst für das Teleskop mit der Trägerrakete Ariane 5 bereit. In Zusammenarbeit mit ihren Partnern war die ESA für die Entwicklung und Qualifizierung der Ariane-5-Anpassungen für die Webb-Mission sowie für die Beschaffung des Startservices durch Arianespace verantwortlich. Die ESA stellte auch den Arbeitsspektrographen NIRSpec und 50 % des Instruments für das mittlere Infrarot (MIRI) zur Verfügung, das von einem Konsortium aus national finanzierten europäischen Instituten (dem MIRI European Consortium) in Zusammenarbeit mit dem JPL und der Universität von Arizona entwickelt und gebaut wurde.
Weiterführende Links
ESA: Pan of the Orion Bar region. Video 0:30 min. https://esawebb.org/videos/weic2315a/
NASA: Webb Makes First Detection of Crucial Carbon Molecule. https://www.nasa.gov/feature/goddard/2023/webb-makes-first-detection-of-crucial-carbon-molecule
ESA: BR-348/DE: Webb – Weiter sehen https://esamultimedia.esa.int/multimedia/publications/BR-348/BR-348_DE.pdf ESA: Webb - Seeing farther.https://www.esa.int/Science_Exploration/Space_Science/Webb
ScienceBlog: Redaktion, 14.07.2022: James-Webb-Teleskop: erste atemberaubende Bilder in die Tiefe des Weltraums
Mikroorganismen spielen eine entscheidende Rolle bei der Kohlenstoffspeicherung in Böden
Mikroorganismen spielen eine entscheidende Rolle bei der Kohlenstoffspeicherung in BödenSa, 17.06.2023— Redaktion
![]() Böden können mehr organischen Kohlenstoff - d.i. Kohlenstoff in Form organischer Verbindungen - speichern als andere terrestrische Ökosysteme. Sie gelten daher als entscheidende Kohlenstoffsenken im Kampf gegen den Klimawandel. Eine bahnbrechende Studie zeigt nun, dass Mikroorganismen die entscheidende Rolle bei der Kohlenstoffspeicherung in Böden spielen. Die Ergebnisse der Studie haben Auswirkungen auf die Verbesserung der Bodengesundheit und die Eindämmung des Klimawandel.
Böden können mehr organischen Kohlenstoff - d.i. Kohlenstoff in Form organischer Verbindungen - speichern als andere terrestrische Ökosysteme. Sie gelten daher als entscheidende Kohlenstoffsenken im Kampf gegen den Klimawandel. Eine bahnbrechende Studie zeigt nun, dass Mikroorganismen die entscheidende Rolle bei der Kohlenstoffspeicherung in Böden spielen. Die Ergebnisse der Studie haben Auswirkungen auf die Verbesserung der Bodengesundheit und die Eindämmung des Klimawandel.
Schon seit mehr als hundert Jahren haben sich Studien mit dem Kohlenstoff-Kreislauf im Boden befasst, wobei es hauptsächlich darum ging zu quantifizieren, wie viel organischer Kohlenstoff aus Laubstreu und den Pflanzenwurzeln in den Boden eingebracht und wie viel davon zersetzt und in Form von CO2 aus dem Boden an die Luft abgegeben wird. Wie viel an organischem Kohlenstoff dem Boden zugeführt wird, ist durch die pflanzliche Primärproduktion bestimmt, wie viel an organischem Material im Boden zersetzt wird - hauptsächlich durch Mikroorganismen - ist für die Geschwindigkeit der Freisetzung von CO2 und damit für die Treibhausgas-Konzentration ausschlaggebend.
Mikroorganismen und Kohlenstoffspeicherung
Dass Mikroorganismen bei der Bildung, Erhaltung und Zersetzung von organischem Kohlenstoff im Boden (soil organic carbon - SOC) eine sehr wichtige Rolle spielen, ist seit langem bekannt, und es wurden bereits enorme Anstrengungen unternommen, um Menge, Abbaubarkeit und Abbaugeschwindigkeit externer Kohlenstoffquellen in Böden zu verfolgen. Diese Untersuchungen haben bislang aber nicht zu einer hinreichend verbesserten Quantifizierbarkeit der Speicherung von SOC geführt. Wesentliche Fragen - welche Faktoren das Gleichgewicht zwischen Kohlenstoffspeicherung im Boden und Zersetzung von SOC mit schlussendlicher Abgabe von CO2 an die Luft beeinflussen, und wie mit dem Klimawandel zusammenhängende Faktoren sich auf die Mikroorganismen und ihre Funktion im Boden auswirken - blieben noch unbeantwortet. Die Rolle der Mikroorganismen wird bis jetzt mehr oder weniger in qualitativer Weise betrachtet, wie dies etwa in einem Übersichtsartikel im Jahr 2020 [1] veranschaulicht wird. Abbildung 1.
| Abbildung 1. . Mikroorganismen im Kohlenstoffzyklus des Bodens. CO2 aus der Atmosphäre wird von Pflanzen (oder autotrophen Mikroorganismen) durch Photosynthese eingebaut und gelangt 1) in Form von einfachen organischen Verbindungen über Wurzelausscheidungen und in Form komplexer organischer Verbindungen über Blatt- und Wurzelstreu in den Boden. 2) Die organischen Verbindungen werden in der "Fabrik" der Mikroorganismen für Wachstum und Stoffwechsel genutzt und entweder 3) als CO2 in die Atmosphäre abgeatmet oder 4) in Form von teilweise zersetztem Material/Metaboliten und mikrobieller Nekromasse gespeichert. (Bild aus Dan Naylor et al., (2020) [1]; von Redn. mit deutscher Beschriftung versehen. Lizenz: cc-by.) |
Entscheidender Faktor bei der Kohlenstoffspeicherung
Ein aus mehr als 30 Forschern bestehendes internationales Team hat kürzlich im Fachjournal Nature eine bahnbrechende Studie veröffentlicht: Mittels eines neuartigen Ansatzes wurde erstmals versucht die Vorgänge zu quantifizieren, welche die Dynamik des organischen Kohlenstoffs - Speicherung und Zersetzung - im Boden bestimmen. Wie sich herausstellte, ist die Effizienz der mikrobiellen Kohlenstoffnutzung (carbon use efficiency - CUE) der ausschlaggebende Faktor für die Relation zwischen Speicherung und Zersetzung. CUE ist dabei eine Messgröße, die anzeigt wie viel Biomasse aus dem aufgenommenen Substrat produziert wird:
CUE = produzierte Biomasse/aufgenommenes Substrat
CUE gibt an, wie viel aus den aufgenommenen organischen Verbindungen für das mikrobielle Wachstum und damit für den in Zellen und in abgestorbener Biomasse (Nekromasse) gespeicherten organischen Kohlenstoff verwendet wird und wie viel im Stoffwechsel der Zellen abgebaut wird und schlussendlich als CO2 in die Atmosphäre entweicht. Ein hoher CUE-Wert kann dabei für verstärktes mikrobielles Wachstum stehen, das zur Akkumulation von organischem Kohlenstoff im Boden führt, aber auch einen Anstieg in der Produktion von Enzymen bedeuten, die organisches Material zersetzen und damit einen Verlust von SOC mit sich bringen. Abbildung 2.
| Abbildung 2. Wie wirkt sich eine hohe mikrobielle Effizienz der Kohlenstoffnutzung (CUE) auf die Speicherung von organischem Kohlenstoff im Boden (SOC) aus? Alternative a) Wachstum begünstigt durch erhöhte mikrobielle Biomasse, Nekromasse und Nebenprodukte die Speicherung von SOC. Alternative b) Erhöhte mikrobielle Biomasse kann über anschließende Enzymproduktion den Abbau von SOC fördern. (Bild aus Tao F. et al. (2023) [2], Lizenz: cc-by). ) |
Um herauszufinden, ob sich nun die Kohlenstoffnutzungseffizienz positiv oder negativ auf die Speicherung von SOC auswirkt, haben die Forscher die Relation zwischen CUE und SOC in umfassender Weise untersucht und auch welchen Einfluss die Wechselwirkungen mit Klima, Vegetation und Boden-Eigenschaften (edaphische Faktoren) darauf haben. Dazu haben sie eine Kombination aus Datensätzen auf globaler Ebene (Metaanalysen von global verteilten vertikalen Kohlenstoff-Bodenprofilen aus 173 Ländern) herangezogen, ein spezifisches mikrobielles Modell für Kohlenstoffprozesse im Boden entwickelt, das mit 57,267 Bodenkohlenstoffdaten kombiniert wurde und Deep Learning und Meta-Analysen eingesetzt.
Die Ergebnisse der Untersuchungen zeigen global eine positive Korrelation zwischen der mikrobiellen Kohlenstoffnutzungseffizienz und der Speicherung von organischem Kohlenstoff im Boden (Abbildung 3). Mehr organischer Kohlenstoff wird also für das mikrobielle Wachstum verwendet als für seinen Verlust in Zersetzungsprozessen, für die Frage wie viel organischer Kohlenstoff im Boden gespeichert wird, ist also das Wachstum der Mikroorganismen ausschlaggebender, als deren Stoffwechsel.
| Abbildung 3. Eine Erhöhung der mikrobiellen Effizienz der Kohlenstoffnutzung (CUE) ist mit einer erhöhten Speicherung von organischem Kohlenstoff im Boden (SOC) verbunden. a) Die aus der Metaanalyse von 132 Messungen resultierende CUE-SOC Relation. b) Ergebnisse aus der Assimilierung aller verfügbarer Kohlenstoff-Bodenprofile (n = 57,267) an das Mikroorganismen-Modell. Beispiel für ein Bodentiefe von 0 - 30 cm. Niedrigere mittlere Jahrestemperaturen (MAT) sind mit höheren Speicherungen verbunden. (Bild: Ausschnitt aus Tao F. et al. (2023) [2], Lizenz: cc-by) |
Wie die Studie zeigt, sind die mikrobiellen Prozesse entscheidend für die Speicherung von SOC. Wesentlich erscheint auch, dass die Kohlenstoffnutzungseffizienz in höheren Breitegraden (niedrigeren mittleren Jahrestemperaturen) höher ist, als in den wärmeren Zonen niedrigerer Breitegrade. Ein deep-learning Ansatz hat schließlich globale Muster von organischem Kohlenstoff im Boden in Abhängigkeit von sieben wesentlichen Prozessen im Kohlenstoff-Zyklus erzeugt: Daraus geht hervor, dass der Einfluss der mikrobiellen Kohlenstoffnutzungseffizienz auf die globale Speicherung und Verteilung von Kohlenstoff im Boden mindestens viermal stärker als der Einfluss anderer biologischer Faktoren oder Umweltbedingungen ist.
Schlussfolgerungen
Die neuen Erkenntnisse stehen am Anfang weiterer Studien zu einem besseren Verständnis der mikrobiellen Prozesse, die der Effizienz der Kohlenstoffnutzung zugrunde liegen. Untersuchungen mit unterschiedlichen Mikrobiomen, Substraten und landwirtschaftlichen Bewirtschaftungsmethoden können in einer verbesserten Kohlenstoffspeicherung und Bodengesundheit resultieren und bei der Vorhersage von SOC-Rückkopplungen als Reaktion auf den Klimawandel helfen.
[1] Dan Naylor et al.: Soil Microbiomes Under Climate Change and Implications for Carbon Cycling. Annu. Rev. Environ. Resour. 2020. 45:29–59. doi:10.1146/annurev-environ-012320-08272
[2] Tao, F., Huang, Y., Hungate, B.A. et al. Microbial carbon use efficiency promotes global soil carbon storage. Nature (2023). https://doi.org/10.1038/s41586-023-06042-3
Artikel zu verwandten Themen im ScienceBlog
- Gerd Gleixner, 05.08.2021:Erdoberfläche - die bedrohte Haut auf der wir leben
- Redaktion, 07.11.2019: Permafrost - Moorgebiete: den Boden verlieren in einer wärmer werdenden Welt
- IIASA, 15.08.2019: Wieviel CO₂ können tropische Regenwälder aufnehmen?
- Knut Ehlers, 01.04.2016: Der Boden - ein unsichtbares Ökosystem
- Rattan Lal, 27.11.2015: Boden – Der große Kohlenstoffspeicher
- Rattan Lal, 11.12.2015: Der Boden – die Lösung globaler Probleme liegt unter unseren Füßen
- Redaktion, 26.06.2015: Die Erde ist ein großes chemisches Laboratorium – wie Gustav Tschermak vor 150 Jahren den Kohlenstoffkreislauf beschrieb
- Hans-Rudolf Bork, 14.11.2014: Die Böden der Erde: Diversität und Wandel seit dem Neolithikum
Erste topische Gentherapie zur Behandlung der Schmetterlingskrankheit (dystrophe Epidermolysis bullosa) wurde in den USA zugelassen
Erste topische Gentherapie zur Behandlung der Schmetterlingskrankheit (dystrophe Epidermolysis bullosa) wurde in den USA zugelassenDo, 08.06.2023 — Ricki Lewis
Epidermolysis bullosa (EB) - volkstümlich als Schmetterlingskrankheit bezeichnet - wird durch Mutationen in Strukturproteinen der Haut ausgelöst und ist durch eine extrem verletzliche Haut charakterisiert. Vor 6 Jahren hat Eva Maria Murauer (EB-Haus, Salzburg) im ScienceBlog über eine Fallstudie berichtet, in der eine große, chronische Wunde am Bein mittels Gentherapie normalisiert werden konnte [1]. Vor wenigen Wochen hat nun die FDA mit Vyjuvek eine erste Gentherapie zur Behandlung der schweren dystrophischen Epidermolysis bullosa (DEB) in den USA zugelassen. Diese Gentherapie hat lange auf sich warten lassen, aber sie unterscheidet sich von einer Handvoll anderer zugelassener Gentherapien: Sie ist nicht nach einmaliger Anwendung abgetan. Die Genetikerin Ricki Lewis berichtet darüber.*
Mein nun schon zehn Jahre altes Buch "The Forever Fix: Gene Therapy and the Boy who Saved It" (Gentherapie und der Junge, der sie rettete) hat die Geschichten von Kindern erzählt, die einmalig anwendbare funktionsfähige Genkopien erhalten hatten, um ihre Mutationen zu kompensieren. Die ersten Gentherapien haben Menschen mit einer vererbten Form der Netzhautblindheit zum Sehen und Kindern mit schweren Immundefekten zum Überleben verholfen. Heute sprechen mehrere durch Einzelgen-Mutationen verursachte Blut-, Gehirn-, Muskel- und Stoffwechselkrankheiten auf einmalige Infusionen einer Gentherapie an.
Die Biologie, die hinter einer Einzelgen-Erkrankung steht, gibt Aufschluss darüber, wie eine bestimmte Gentherapie zielgerichtet, angewandt und die Wirkung aufrechterhalten werden kann. Im Vergleich zu "Slash-and-Burn"-Technologien wie Standard-Chemotherapie und Bestrahlung, die nicht nur die Zielzellen betreffen, ist eine Gentherapie sowohl rational als auch maßgeschneidert.
Bei der Netzhautblindheit werden die Gene in die nährende Zellschicht unter den Stäbchen und Zapfen injiziert, welche die elektrischen, ein visuelles Bild vermittelnde Signale an das Gehirn weiterleiten. Da sich die Zellen dieser Schicht normalerweise nicht teilen, bleiben die verabreichten Gene an Ort und Stelle, unterhalten die Stäbchen und Zapfen und bewahren das Sehvermögen, das andernfalls verloren gehen würde.
Einige andere Gentherapien verfolgen einen umgekehrten Ansatz; sie werden in Stammzellen eingebracht, sodass sie sich bei der Zellteilung ausbreiten können.
Für die Haut stellt die Gentherapie eine ganz andere Herausforderung dar, weil die Zellen in der Regel so häufig ersetzt werden - es liegt in der Natur des Organs, sich permanent zu erneuern. Die Anwendung einer Gentherapie ist also das Gegenteil von einmal und fertig; sie muss viel häufiger erfolgen.
Topische und redosierbare Gentherapie
Am 19. Mai hat die FDA Vyjuvek, eine Gentherapie für DEB, zugelassen. In der Ankündigung des Erzeugers Krystal Biotech wird sie als "die erste redosierbare Gentherapie" bezeichnet.
Die Ursache für DEB ist eine Mutation im Kollagen-Gen COL7A1, das eine der beiden Hauptkomponenten des Bindegewebes darstellt. In gesunder Haut fügen sich Kollagenproteine zu Fibrillen zusammen, die die Epidermis mit der darunter liegenden Dermis verbinden, ähnlich wie Mozzarella-Käse die Schichten einer Lasagne verankert. Ohne COL7A1-Kollagen lösen sich die Hautschichten ab, und es entstehen schmerzhafte und beeinträchtigende Blasen und Wunden. Täglich.
Betroffene Kinder mit einer rezessiven Form - RDEB - werden manchmal als "Schmetterlingskinder" bezeichnet, weil ihre Haut so zart ist, wie die zerbrechlichen Flügel eines Insekts. Doch dieses Bild hat keine Ähnlichkeit mit der Realität der Extremitäten, die durch ständige Blasenbildung, Schälen, Blutungen und Narbenbildung unbrauchbar werden. Abbildung 1.
| Abbildung 1. Ein Schmetterlingskind. (Bild: Yovanna.Gonzalez, commons.wikimedia.org, Lizenz: cc-by-sa) |
Bei der geringsten Berührung bilden sich sofort schmerzhafte Blasen, und die Haut schält sich ab. Das Entfernen von Verbänden ist eine Qual, die täglich und ein Leben lang durchgeführt werden muss. Die Krankheit kann zu Sehkraftverlust, Entstellung und anderen Komplikationen führen, von denen einige tödlich sind.
RDEB wird von zwei Trägereltern vererbt. Eine dominante Form - DDEB - ist weniger gravierend, verursacht Blasenbildung an Händen, Füßen, Knien und Ellenbogen.
DEB ist seit 2002 ein Kandidat für eine Gentherapie. In einem Bericht aus dem Jahr 2018 habe ich die Geschichte der Bemühungen nachgezeichnet [2].
Patienten als ihre eigenen Kontrollpersonen
Die eben zugelassene Gentherapie liefert funktionierende Kopien des COL7A1-Kollagengens; Vektoren sind Herpes-Simplex-Viren, die so optimiert sind, dass sie sich nur in den betroffenen Hautzellen vermehren. Ein Arzt trägt einmal pro Woche Tropfen des Vyjuvek-Gels auf die Wunden eines Patienten auf.
Es wurden zwei klinische Studien durchgeführt und die Ergebnisse im Dezember 2022 veröffentlicht. In einer Phase 3-Studie, die im New England Journal of Medicine veröffentlicht wurde, wurden 30 Personen im Alter von 1 bis 44 Jahren, die an RDEB leiden und eine an DDEB leidende Person untersucht [3]. Bei jedem Teilnehmer wurden zwei Wunden ähnlicher Größe ausgewählt, von denen eine wöchentlich mit der Gentherapie und die andere mit einem Placebo behandelt wurde. Nach 24 Wochen wurden die Wunden miteinander verglichen. Von den mit Vyjuvek behandelten Wunden hatten sich 65 % vollständig geschlossen, verglichen mit nur 26 % der mit Placebo behandelten Wunden. Vorhergehende Ergebnisse aus Phase 1/Phase 2-Studien sind in Nature Medicine veröffentlicht worden [4]. Abbildung 2.
| Abbildung 2. Die Behandlung mit Vyjuvek führt zur Heilung der Hautwunden. Links: Nach 3 Monaten hat sich der Großteil der Wunden vollständig geschlossen (repräsentative Fotos der Wunden w1 und w3 von Patient 10). Rechts: Histologische Analyse der Hautproben (von Patient 10) zeigt bereits nach 15 Tagen Behandlung die Expression von funktionellem Kollagen 7-Protein, welches Epidermis mit Dermis verknüpft (weiße Pfeile markieren die Grenze Epidermis/Dermis). Mit seiner Domäne NC1 (links, rot gefärbt) verankert Kollagen 7 die Fibrillen in der Basalmembran, mit der Domäne NC2 (rechts grün gefärbt) fördert es den Zusammenbau der Fibrillen. Blau gefärbt sind die Zellkerne. (Bild von Redaktion eingefügt; es sind Ausschnitte aus der oben zitierten Arbeit von Irina Gurevich et al., 2022 [4]. Lizenz cc-by). |
Studienleiter M. Peter Marinkovich, MD, Direktor der Blistering Disease Clinic am Stanford Health Care, bezeichnet DEB als eine verheerende Krankheit. "Bis jetzt hatten Ärzte und Krankenschwestern keine Möglichkeit, die Entstehung von Blasen und Wunden auf der Haut von dystrophischen EB-Patienten zu verhindern, und alles, was wir tun konnten, war, ihnen Verbände zu geben und hilflos zuzusehen, wie sich neue Blasen bildeten. Die topische Gentherapie von Vyjuvek ändert all dies. Sie heilt nicht nur die Wunden der Patienten, sondern verhindert auch eine erneute Blasenbildung, weil sie den zugrunde liegenden Hautdefekt der dystrophen EB korrigiert. Da sie sicher und einfach direkt auf die Wunden aufzutragen ist, erfordert sie nicht viel unterstützende Technologie oder spezielles Fachwissen; dies macht Vyjuvek auch für Patienten, die weit entfernt von spezialisierten Zentren leben, besonders zugänglich."
[1] Eva Maria Murauer, 02.03.2017: Gentherapie - Hoffnung bei Schmetterlingskrankheit. https://scienceblog.at/gentherapie-hoffnung-bei-schmetterlingskrankheit
[2] Ricki Lewis, 08.02.2018: Gene Therapy for the “Butterfly Children”https://dnascience.plos.org/2018/02/08/gene-therapy-for-the-butterfly-children/
[3] Shireen V. Guide et al., Trial of Beremagene Geperpavec (B-VEC) for Dystrophic Epidermolysis Bullosa. 15.12. 2022. N Engl J Med 2022; 387:2211-2219. DOI: 10.1056/NEJMoa2206663
[4] Irina Gurevich et al., In vivo topical gene therapy for recessive dystrophic epidermolysis bullosa: a phase 1 and 2 trial. Nat Med 28, 780–788 (2022).https://doi.org/10.1038/s41591-022-01737-y. open access.
*Der Artikel ist erstmals am 1. Juni 2023 in PLOS Blogs - DNA Science Blog unter dem Titel "Topical Gene Therapy FDA-Approved for Severe Skin Disease, Dystrophic Epidermolysis Bullosa" https://dnascience.plos.org/2023/06/01/topical-gene-therapy-fda-approved-for-severe-skin-disease-dystrophic-epidermolysis-bullosa/ erschienen und steht unter einer cc-by Lizenz . Die Autorin hat sich freundlicherweise mit der Übersetzung ihrer Artikel durch ScienceBlog.at einverstanden erklärt. Der leicht gekürzte Artikel folgt so genau wie möglich der englischen Fassung. Abbildung 2 wurde von der Redaktion aus der zugrundeliegenden klinischen Studie [4] eingefügt.
Anmerkung der Redaktion
DEB is eine sehr seltene Erkrankung, Schätzungen zur weltweiten Inzidenz gehen von etwa 100 000 Fällen aus (https://www.eb-researchnetwork.org/research/what-is-eb/).
Nach Mitteilung des Herstellers Krystal Biotech werden sich die Kosten von Vyjuvek auf 24 250 $/ Ampulle belaufen, die geschätzten jährlichen Behandlungskosten auf rund 631 000 $. Damit kommt Vyjuvek zwar in die Liste der weltweit 10 teuersten Medikamente, allerdings beläuft sich auch die bislang allein palliative Behandlung auf jährlich 200 000 bis 400 000 $ (https://ir.krystalbio.com/static-files/e6f347b3-0c14-43d9-b7d3-c9a274267294).
Prionen - wenn fehlgefaltete Proteine neurodegenerative Erkrankungen auslösen
Prionen - wenn fehlgefaltete Proteine neurodegenerative Erkrankungen auslösenDo, 01.06.2023— Michael Simm
Für eine Handvoll besonderer Proteine scheinen die Regeln der Biologie nicht zu gelten. Prionen sind infektiöse Proteine, deren Übertragung auch ohne Beteiligung von Erbsubstanz eine Reihe von Krankheiten auszulösen vermag. Dies geschieht nach Art einer Kettenreaktion, bei der die falsch gefalteten krankmachende Prionen ihr natürliches Gegenstück, das zelluläre Prionprotein, umfalten. So entstehen Aggregate und Nervenzellen sterben ab. Wie sie den Rinderwahn verursachen und auf Menschen überspringen konnten, ist eine gruselige Geschichte über die der deutsche Biologe und Wissenschaftsjournalist Michael Simm hier berichtet.*
Wäre Stanley Prusiner einige hundert Jahre früher geboren, hätte man ihn womöglich als Ketzer verbrannt. Oder zumindest aus der Gemeinschaft der Gläubigen ausgestoßen. Denn der Virologe an der Universität Kalifornien in San Francisco und Berkeley hatte an dem Glaubensbekenntnis seiner Zunft gerührt. Das „Zentrale Dogma der Molekularbiologie“ stammt aus dem Jahr 1958 und wurde von keinem geringeren als Francis Crick formuliert, einem Mitbegründer der modernen Biologie und eher unbescheidenen Genie. Dieses Dogma besagt, dass Informationen in biologischen Systemen stets von der Erbsubstanz DNA zum Botenstoff RNA fließen und schließlich in Proteine umgesetzt werden. Die Vorstellung, dass ein Protein genügen könnte, um eine Krankheit zu übertragen, war damit eigentlich unvereinbar. Doch genau dies hatte Prusiner behauptet. Oder zumindest hatte er diese Hypothese weiter verfolgt und ausgebaut. Denn genau genommen, kam die Idee vom infektiösen Protein bereits in den 1960er Jahren auf.
Im Zentrum seines Interesses standen zunächst Schafe, Studienobjekte, die an Scrapie (von engl. „kratzen“) verendet waren. Seit dem 18. Jahrhundert war diese tödliche Tierseuche mit ihren Gang- und Verhaltensstörungen in Europa bekannt, und auch das von Hohlräumen durchlöcherte Gehirn der Tiere war immer wieder beschrieben worden.
Mit Gewebeproben und Blut ließ sich das Leiden im Labor von kranken auf gesunde Schafe übertragen. Auch auf Ziegen, Nerze, Ratten und Mäuse. Ähnliches war US-Forschern bereits 1972 gelungen, als sie infektiöses Material aus dem Gehirn von Schafen, zunächst auf Mäuse und dann vom Mäusegehirn auf Javaneraffen (Macaca fascicularis) übertragen hatten – ein frühes Schlüsselexperiment, auch wenn die Autoren der Studie davon ausgingen, dass ein Virus hinter der Erkrankung steckte. Abbildung 1.
|
Abbildung 1. Vereinfachte Darstellung der Prion-Exposition von Schafen (rote Pfeile) und der Übertragung unter den Tieren (blaue Pfeile) . Bild von Redaktion modifiziert eingefügt (Quelle: Neil A. Mabbott: How do PrPSc Prions Spread between Host Species, and within Hosts? www.mdpi.com/journal/pathogens. Lizenz: cc-by) |
Dieser Verdacht bestätigte sich jedoch nicht. Der Erreger war ungewöhnlich widerstandsfähig, schlüpfte auch noch durch die engmaschigsten Filter und überdauerte verschiedenste Methoden zur Inaktivierung von Nukleinsäuren. Gab man hingegen eiweißspaltende Enzyme (Proteasen) hinzu, so verloren die Gewebeproben zumindest teilweise ihre Infektiosität. 1982 hatte Prusiner genug Beweise gesammelt. Er veröffentlichte seine Entdeckung in der Fachzeitschrift „Science“ unter der Überschrift: „Novel proteinaceous infectious particles cause scrapie“ – „Neue proteinartige infektiöse Partikel verursachen Scrapie“. Die mysteriösen Erreger nannte Prusiner „Prion“, abgeleitet vom englischen „proteinaceous infectious particles“ (proteinartige infektiöse Partikel).
Nobelpreis für einen Außenseiter
Etwa zehn weitere Jahre verbrachte der wissenschaftliche Außenseiter darauf, seine Prionenhypothese gegen die Zweifel und die Angriffe vieler forschender Kollegen zu verteidigen. Doch er sollte recht behalten und wurde – auch als Anerkennung für sein Durchhaltevermögen – 1997 mit dem Nobelpreis für Physiologie oder Medizin geehrt. Heute weiß man: Der Bauplan von Prion-Proteinen ist zwar genauso in einer Reihenfolge von DNA-Bausteinen festgeschrieben wie bei „normalen“ Proteinen. Der Weg von der DNA über die RNA zum Protein ist also durchaus, dem Dogma entsprechend, gegeben. Allerdings kann das im Genom kodierte Prionprotein (PrP) eine alternative Konformation, also eine alternative dreidimensionale Faltung, annehmen, mit weiteren Prionproteinen verklumpen und seine falsche Struktur dann wiederum normalen Prionproteinen aufzwingen. In seltenen Fällen und oftmals über lange Zeiträume hinweg kann dies zu einer über viele Jahre unbemerkten fatalen Kettenreaktion führen. Abbildung 2.
|
Abbildung 2. Der Übergang von der nativen Konformation des zellulären Prionproteins (PrPc) in eine pathologische Isoform, die als "Prionprotein-Scrapie" (PrPsc) bezeichnet wird, kann mehrere Prion-assoziierte neurodegenerative Erkrankungen auslösen. Bild von Redaktion in modifizierter Form eingefügt aus Yuhai Zhao et al., Molecules 2022, 27(16), 5123; https://doi.org/10.3390/molecules27165123; Lizenz cc-by) |
All dies wäre womöglich nur eine Randnotiz in der Veterinärmedizin geblieben, hätte man nicht eine ganze Reihe weiterer Krankheiten entdeckt, die Scrapie ähneln und auch Menschen betreffen können. Das Interesse des Humanmediziners Prusiner wurde nach eigenen Angaben geweckt, als ein Patient mit der seltenen Creutzfeldt-Jakob-Erkrankung (CJD) in seiner Obhut verstorben war. Typisch für diese bis heute unheilbare Krankheit sind Bewegungsstörungen, Gedächtnisverlust und Verwirrtheit, sowie ein schneller Verlauf, der in der Regel nach vier Monaten bis zwei Jahren tödlich endet.
Fasziniert war der Neurologe auch von Gerüchten über eine geheimnisvolle Krankheit namens Kuru, die unter einigen Urvölkern in der Ost-Hälfte Neuguineas (heute Papua-Neuguinea) kursierte, und dort bis etwa 1950 offenbar durch rituellen Kannibalismus verbreitet wurde. Die ersten Weißen, die 1933 ins Hochland vordrangen, trafen dort buchstäblich auf eine vergessene Welt. Die Menschen dort lebten noch in der Steinzeit, gehörten Dutzenden von Völkern an, die sich gegenseitig bekriegten und unterschiedliche Sprachen und Dialekte hatten.
Forscher unter Kannibalen
Dass Kannibalismus weit verbreitet war, bestätigen zahlreiche Berichte nicht nur von Missionaren, sondern auch von Anthropologen und der berühmten US-amerikanischen Ethnologin Margaret Mead . Nach einem Besuch des Ortes Timbunke am Fluss Sepik schrieb sie: „Der Fluss steht für Moskitos, Krokodile, Kannibalen und im Wasser treibende Leichen – und ich kann versichern, ich habe alles gesehen.“ Michael Rockefeller , Mitglied der bekannten US-Familie und Hobby-Ethnograf verschwand 1961 an der Südküste Neuguineas und wurde seinem Biographen zufolge von Angehörigen des Volkes des Asmat umgebracht.
Schließlich reiste auch Prusiner nach Neuguinea. Im Jahr 1982 traf er dort auf Carleton Gajdusek , einen Überflieger, der Physik, Chemie, Mathematik studiert, bei den Nobelpreisträgern Linus Pauling und Frank Burnet gelernt und schließlich in Harvard seinen Doktor der Medizin gemacht hatte. Gajdusek war dem Erreger von Kuru auf der Spur und hatte die Krankheit 1966 bereits auf Schimpansen übertragen. Zwei Jahre später gelang ihm das mit CJD, und 1972 mit Scrapie. Ebenso wie Prusiner hat auch Gajdusek den Medizin-Nobelpreis erhalten, allerdings bereits einige Jahre früher, im Jahr 1976. Die beiden Wissenschaftler haben sich laut Prusiners Erinnerungen nicht besonders gut vertragen. Sie waren wohl eher Konkurrenten, denn Kollegen, wie aus einem Rückblick hervorgeht , den Prusiner im Jahr 2008 veröffentlicht hat.
Kuru, eine Krankheit, die nach dem Ausbruch schnell zum Tod führte, hatte offenbar einiges mit CJD und Scrapie gemeinsam, wie Prusiner und andere Kollegen erkannten. Das Zittern und andere Bewegungsstörungen, der bei Kuru eine Demenz folgte, hatten beide Mediziner beobachtet. Für Gajdusek war die Krankheit jedoch der Parkinsonkrankheit ähnlich – für Prusiner nicht. Der wichtigste Streitpunkt aber war, dass Gadjusek nicht an Prusiners Prionen-Hypothese glaubte. Als Prusiner in einer Publikation über Kuru Gajdusek als Co-Autor nennen wollte, erlaubte Gadjusek dies erst, nachdem das Wörtchen „Prionen“ aus dem Artikel getilgt worden war.
Gajduseks machte, jenseits der Wissenschaft, in negativer Weise von sich reden: Von seinen Reisen in die Tropen hatte er – mit Einverständnis der Eltern – insgesamt 56 Kinder mitgebracht und bei sich zu Hause in einer Art Kommune erzogen. Als Vorwürfe von sexuellem Missbrauch laut wurden, bezeugte dies einer seiner „Schützlinge“, der mittlerweile studierte und im Alter von 14 Jahren mit Gadjusek zusammengelebt hatte. Der Forscher wurde daraufhin zu einem Jahr Gefängnis verurteilt, verlor seine Position als Leiter des Hirnlabors am Nationalen Schlaganfallinstitut (NINDS) und lebte nach Verbüßung seiner Strafe bis zum Tod in Paris, Amsterdam und Tromsö, was er nach eigenen Angaben bevorzugte, weil er dort in der ständigen Dunkelheit des Winters besser arbeiten konnte.
Heute werden Scrapie, Kuru und die übertragbare Variante der CJD (es gibt auch eine vererbte und eine spontan auftretende Form) aufgrund ihrer Pathologie als „Transmissible spongiforme Enzephalopathien“ (TSE) eingeordnet – zu Deutsch: „Übertragbare schwammartige Hirnleiden“. Auch der zusammenfassende Begriff „Prionenerkrankungen“ ist geläufig. Die wohl bekannteste aller TSE wurde erstmals 1984 in England nachgewiesen und als „Bovine spongiforme Enzephalopathie“ (BSE) benannt. Besser bekannt ist diese Tierkrankheit unter dem Namen Rinderwahn.
BSE entstammte einer gefährlichen Mixtur von unnatürlichen Praktiken der Fleischerzeugung sowie routinemäßigen Gesetzesverstößen und Vertuschungsversuchen, wie aus Zeugenbefragungen in einem offiziellen Untersuchungsausschuss hervorging. So hatte man Fleisch und Knochenmehl aus Schafskadavern quasi als „Kraftfutter“ an Rinder verfüttert. Bei der eigentlich vorgeschriebenen Sterilisation durch Hitze und Druck wurde geschlampt – oder gar zwecks Kostenoptimierung manipuliert, sodass Scrapie-Erreger aus infizierten Schafen überlebten, vom Magen der Rinder ins Nervensystem gelangten, sich an den neuen Wirt anpassten und die Rinder töteten. Fast 200.000 Tiere verendeten an BSE, und in der gesamten EU wurden fast vier Millionen Rinder gekeult.
Leichtsinn und Skrupellosigkeit am Rande der Apokalypse
Wenn Scrapie von Schafen auf Rinder übertragbar ist, besteht dann nicht auch die Gefahr einer Übertragung von BSE auf den Menschen? Die Befürchtung lag aus heutiger Sicht nahe. Dennoch gelangten verseuchte Hamburger, Würste und andere Fleischprodukte in die Mägen der Verbraucher in aller Welt. Gelatine aus Haut und Knochen wurde zu Pudding, Tortenbelag oder Gummibärchen verarbeitet. Und es dauerte 10 Jahre von den ersten Warnungen der Experten, bis die britische Regierung 1996 einräumte, dass da möglicherweise doch eine Verbindung bestehe zwischen dem Rinderwahn und einer neuen Variante der CJD, an der zu diesem Zeitpunkt bereits 10 Menschen gestorben waren. Nach aktuellem Stand sind es weltweit mehr als 200 Tote, davon die weitaus meisten in Großbritannien, knapp 30 in Frankreich, aber noch kein einziger Fall in Deutschland. Die Menschheit hat offenbar Glück gehabt. Wenn BSE ebenso leicht auf den Menschen übergesprungen wäre, wie viele Viren das tun, hätte es womöglich Millionen von Toten gegeben.
Viele Krankheiten – ein Mechanismus
Die durch Prionen von Rindern auf Menschen übertragene Form der CJD wird als vCJD (für „Variante“) gekennzeichnet. Und es gibt noch eine weitere Form, die iatrogene CJD (iCJD) mit einer eher kleinen Zahl von dokumentierten Fällen, bei denen die Krankheit durch medizinische Prozeduren wie die Verpflanzung von Hirnhäuten, Augenhornhaut oder der Gabe von Wachstumshormonpräparaten von Mensch zu Mensch übertragen wurde. Abbildung 3.
Alle bisher genannten Prionenkrankheiten verbreiten sich mittels Übertragung. Sie machen aber zusammen weniger als ein Prozent aller Fälle aus. Mit mehr als 85 Prozent ist die sporadische Creutzfeldt-Jakob-Krankheit unter den menschlichen Prionenerkrankungen am häufigsten, und für etwa zwei Todesfälle unter je 1 Millionen Einwohner pro Jahr verantwortlich. Der Rest – rund 15 Prozent – verteilt sich auf erbliche, also genetisch bedingte Prionenerkrankungen wie die familiäre Form der CJD, das Gerstmann-Sträussler-Scheinker (GSS)-Syndrom und die Fatale Familiäre Insomnie (FFI).
|
Abbildung 3. Modell der Proteinpolymerisierung für die Umwandlung von PrPC in PrPSc bei sporadischen, erblichen und erworbenen Prionenerkrankungen. Links im Bild: humanes Prion in Lösung (NMR-Struktur 1HJM, PDB DOI: https://doi.org/10.2210/pdb1HJM/pdbB). Bild in modifizierter Form von Redaktion eingefügt aus Hideyuki Hara & Suehiro Sahaguchi, Int. J. Mol. Sci. 2021, 22(22), 12439; https://doi.org/10.3390/ijms222212439; Lizenz cc-by) |
Bei den erblichen Prionen-Erkrankungen ist die Reihenfolge der Bausteine in dem natürlichen Gen PRNP auf Chromosom 20 verändert. Inzwischen kennt man mehr als 60 Varianten davon. Ein Vergleich der Genome von über 16.000 Patienten mit rund 600.000 nicht Betroffenen in drei Datenbanken ergab, dass nur wenige Varianten unweigerlich zum Ausbruch der Krankheit führen, viele Varianten dagegen auch bei gesunden Menschen auftraten. Bei den letzteren Fällen müssen offenbar weitere ungünstige Umstände hinzukommen, wie möglicherweise Umweltgifte oder Vorbelastungen durch andere Krankheiten.
Zentral für das Verständnis der Prionenkrankheiten sind die Prionproteine selbst. Die krankhafte, falsch gefaltete und verklumpte Form wird als PrPSc bezeichnet – wobei das „Sc“ auf die Schafskrankheit Scrapie verweist –, und die normale, „gesunde“ Form als zelluläres PrPC . Gelangen einige wenige PrPSc in eine Umgebung voller PrPC so werden auch die „gesunden“ Moleküle umgeformt und die Kettenreaktion nimmt ihren Lauf. Die Konzentration von PrPSC im Hirn und Rückenmark steigt rapide an, und führt schließlich zum Tod von Nervenzellen.
Eine verwirrende Vielfalt von Funktionen
Zwar gibt es Dutzende von Forschungsarbeiten zur Funktion des Proteinproteins . Diese sind jedoch oftmals widersprüchlich oder nicht ausreichend, um das Absterben der Nervenzellen zu begreifen – geschweige denn zu verhindern. PrPC zum Beispiel ist, laut einer aktuellen Übersichtsarbeit , bei der neuronalen Entwicklung beteiligt, bei der Zelladhäsion, der Neuroprotektion, der Regulation zirkadianer Rhythmen, der Aufrechterhaltung der Ionenhomöostase usw. Zwar kennt man inzwischen recht genau die Reaktionswege beim Umfaltungsprozess. Und auch die dreidimensionalen Strukturen der beteiligten Eiweißfragmente und Zwischenprodukte sind zunehmend bekannt. Nachgewiesen ist zudem, dass PrPSc bei über die Nahrung erfolgter Übertragung – etwa beim Verzehr von BSE-kontaminiertem Rindfleisch – offenbar vom Magen her in das Nervensystem gelangt und sich dort auf der Membran von Nervenzellen festsetzt. Wie genau es von diesem Punkt aus weitergeht, ist jedoch ziemlich unklar.
Wer nun denkt, dass Stanley Prusiner nach dem Nobelpreis und der Bestätigung seiner Prionenhypothese zufrieden die Hände in den Schoss legt, der irrt. Nach seiner Auffassung könnten Prionen, oder vielmehr ein prionenartiger Mechanismus von sich ausbreitender Eiweißfehlfaltung im Gehirn auch bei anderen neurodegenerativen Erkrankungen am Werk sein. Sowohl bei der Alzheimer-Demenz als auch der Parkinson-Krankheit und bei Chorea Huntington spielen jeweils unterschiedliche Proteine eine Rolle (Aß, Tau, α-Synuklein und Huntingtin), deren Verhalten doch sehr an das von Prionen erinnere. Die Parallelen werden von vielen Forschern durchaus anerkannt. Zwar gibt es keine harten Daten, dass auch nur eine einzige dieser Krankheiten im herkömmlichen Sinne ansteckend sein könnte, doch lauter Widerspruch gegen Stanley Prusiner ist seltener geworden. Im „Harrison“, dem wichtigsten Lehrbuch der Medizin, verantwortet Prusiner das Kapitel über Prionenkrankheiten – eine Art Ritterschlag, die bezeugt, dass dem ehemaligen Außenseiter längst wissenschaftliche Anerkennung zuteilwird.
*Der vorliegende Artikel ist auf der Webseite www.dasGehirn.info im Mai 2023 erschienen, in dessen Fokus "Eindringlinge - Attacke auf’s Gehirn!" stand:https://www.dasgehirn.info/krankheiten/eindringlinge/prionen-eiweisse-die-wahnsinnig-machen. Der Artikel steht unter einer cc-by-nc-nd Lizenz. Der Text wurde mit Ausnahme des Titels von der Redaktion unverändert verwendet; zur Visualisierung wurden 3 Abbildungen eingefügt.
dasGehirn ist eine exzellente deutsche Plattform mit dem Ziel "das Gehirn, seine Funktionen und seine Bedeutung für unser Fühlen, Denken und Handeln darzustellen – umfassend, verständlich, attraktiv und anschaulich in Wort, Bild und Ton." (Es ist ein Projekt der Gemeinnützigen Hertie-Stiftung und der Neurowissenschaftlichen Gesellschaft e.V. in Zusammenarbeit mit dem Zentrum für Kunst und Medientechnologie Karlsruhe).
Zum Weiterlesen
• Sigurdson CJ et al. Cellular and Molecular Mechanisms of Prion Disease. Annu Rev Pathol. 2019 Jan 24;14:497-516. doi: 10.1146/annurev-pathmechdis-012418-013109
• Prusiner SB, Miller BL. Prion Diseases. In: Jameson J, Fauci AS, Kasper DL, Hauser SL, Longo DL, Loscalzo J. eds. Harrison's Principles of Internal Medicine, 20e. McGraw Hill; 2018. (zum Buchkapitel)
----------------------------------------------------------------------
Catalyst University: Prions | Mechanism of Kuru & Relation to Creutzfeldt-Jakob Disease, Video 11:38 min. https://www.youtube.com/watch?v=Y1_V3oVbvxs
Homöopathie - Pseudomedizin im Vormarsch
Homöopathie - Pseudomedizin im VormarschFr 26.05.2023 — Inge Schuster
Vor über 200 Jahren ist die Homöopathie entstanden basierend auf Prinzipien, die sowohl von der wissenschaftlichen Seite als auch von einer Evidenz-basierten Medizin her in höchstem Maße unplausibel sind. Auch, wenn es bis jetzt keine zweifelsfreien Nachweise für Wirksamkeit und Sicherheit gibt, sind weltweit Hunderte Millionen Menschen von der Heilkraft homöopathischer Mittel überzeugt.
Vor den Werbespots, die zur Primetime über den Bildschirm flimmern, gibt es kaum ein Entrinnen. Neben den - milde ausgedrückt - grenzwertigen Spots einer Möbelkette gibt es vor allem auch solche, die im Zuseher die Hoffnung erwecken sollen, dass er bislang erfolglos behandelte Beschwerden effizient und ohne schädliche Nebenwirkungen loswerden kann. Die in den kurzen, 10 - 20 Sekunden dauernden Spots beworbenen Mittel verheißen rasche Besserung diverser Leiden, die von Schlafstörungen über Arthrosen, Nervenschmerzen, Schwindel-Attacken bis hin zu Erektionsstörungen reichen.
Für einen Großteil dieser allabendlich gegebenen Heilsversprechen zeichnet ein am Westrand von München situiertes Unternehmen - die PharmaSGP (https://pharmasgp.com/) - verantwortlich. Das Unternehmen hat 2012 sein erstes Produkt - Deseo gegen sexuelle Schwäche - eingeführt und dank des aggressiven Marketings seines stetig wachsenden Portfolios einen erstaunlichen Aufstieg erlebt; bei den Produkten handelt sich hauptsächlich um Homöopathika, rezeptfreie Mittel wie Rubaxx, Restaxil, Taumea und Neradin. Seit 2020 ist das Werk börsennotiert, der Umsatz von rund 63 Millionen Euro im Jahr 2019 wird sich heuer auf 91 bis 96 Millionen Euro steigern. PharmaSGP rühmt sich "Unsere Arzneimittel basieren auf natürlichen pharmazeutischen Wirkstoffen mit dokumentierter Wirksamkeit und haben nahezu keine bekannten Nebenwirkungen."
Homöopathische Mittel liegen auch anderswo im Trend. Trotz fundierter negativer Kritik an diesen Mitteln, die weder eine rationale wissenschaftliche Basis noch zweifelsfreie Nachweise für Wirksamkeit und Sicherheit vorweisen können, (s.u.) vertrauen weltweit immer mehr Menschen auf solche Alternativen. Globale Schätzungen gehen von mehr als 300 Millionen Menschen aus - in westlichen Ländern jeder Zehnte -, die bereits solche Mittel konsumieren und die Tendenz ist stark steigend. Laut einem aktuellen Report wird der globale Markt für Homöopathika mit 10,7 Milliarden US-Dollar im Jahr 2021 beziffert und bis 2031 ein sehr starkes Wachstum (jährliche Wachstumsrate 18,1 %) auf 32,4 Milliarden Dollar prognostiziert. (https://www.transparencymarketresearch.com/homeopathy-product-market.html). Das ist allerdings nur ein Bruchteil des globalen Pharmaka-Markts: dieser liegt 2022 bei rund 1,5 Billionen US-Dollar; Prognosen für 2026 gehen von einer Steigerung auf rund 2,24 Billionen Dollar (jährliche Wachstumsrate 7,7 %) aus (https://www.transparencymarketresearch.com/homeopathy-product-market.html).
Prinzipien der Homöopathie ........
Ende des 18. Jahrhunderts hat Samuel Hahnemann, ein deutscher Arzt, der sich auch mit Chemie und Pharmazie beschäftigte, "ein neues Princip zur Auffindung der Heilkräfte der Arzneysubstanzen" entwickelt. In einer Zeit, in der man Schwerkranke durch Verabreichung hochgiftiger Stoffe (beispielsweise Quecksilber zur Behandlung der "Franzosenkrankheit" Syphilis), Aderlässe und Durchfälle noch mehr erschöpfte, versprach Hahnemann sanfte Behandlungsmethoden ohne schwere Nebenwirkungen. Angeblich auf einem Selbstversuch zur Anwendung von Chinarinde bei Malaria basierend hat er das zentrale Prinzip, „Ähnliches mit Ähnlichem“ zu heilen - similia similibus curentur - formuliert: eine Substanz, die am Menschen ein bestimmtes Symptom verursacht, kann demnach zur Behandlung einer dasselbe Symptom hervorrufenden Krankheit angewandt werden. Aus der Beobachtung der Symptome, die ein potenziertes (s.u.) Homöopathikum am gesunden Menschen hervorruft, wird auf seine Indikation geschlossen - also beispielsweise Verdünnungen des hochgiftigen gefleckten Schierlings (Conium maculatum), der Sokrates tötete, zur Behandlung von allmählich auftretenden Lähmungen.
Das zweite Prinzip besagt, dass Substanzen durch einen Prozess der seriellen Verdünnung in ihrer Wirkung gesteigert, potenziert werden. Ausgang des Potenzierens ist dabei die sogenannte Urtinktur, die durch Lagerung von Heilpflanzen, tierischen oder mineralischen Komponenten in einer Wasser-Ethanolmischung herausgelöst wird. Diese Urtinktur wird dann im Verhältnis 1:10 (D) oder 1:100 (C) mit Ethanol oder Ethanol/Wasser weiter und weiterverdünnt und nach einem genau vorgegebenem Ritual "verschüttelt". Nach 4 seriellen D-Verdünnungen (D4) ist vom Stoffgemisch der Urtinktur nur mehr ein Zehntausendstel vorhanden, nach 4 C-Verdünnungen ein Hundertmillionstel. Hahnemann war davon überzeugt, dass homöopathische Mittel umso stärker wirken, desto mehr sie potenziert werden. Er konnte damals, in der "Frühzeit" der Chemie, aber noch nicht wissen, dass in den besonders hohen Potenzen (größer als D12, C6) die Verdünnung bereits so groß ist, dass keine Moleküle der Urtinktur mehr nachweisbar sind.
| Homöopathische Globuli und Dilutionen Bild: Wikipedia, gemeinfrei). |
............im Lichte der heutigen Wissenschaften
Über 200 Jahre nach ihrer Entstehung und im Lichte der modernen Wissenschaften erscheinen Prinzipien und Vorgangsweisen der Homöopathie in höchstem Maße unplausibel, sowohl von der wissenschaftlichen Seite als auch von einer Evidenz-basierten Medizin her.
Dafür, dass ähnliche Symptome hervorrufende Substanzen kausal in den Krankheitsprozess eingreifen können, fehlt jegliche wissenschaftliche Grundlage. Dass die Wirksamkeit durch Verdünnen potenziert wird, steht in krassem Widerspruch zu physikalisch-chemischen Gesetzmäßigkeiten und den Dosis Wirkungsbeziehungen der Medizin. Es müssen schließlich genügend Moleküle vorhanden sein, um Reaktionen auszulösen, an Rezeptoren zu binden und Signale an das System senden zu können. Die Annahme von einigen Verfechtern der Homöopathie, dass durch Ausgangsstoff und Verdünnungsvorgänge die Struktur des wässrigen Lösungsmittels verändert und so die Wirksamkeit potenziert wird, ist nicht haltbar: die Wechselwirkungen zwischen den Lösungsmittelmolekülen erfolgen in weniger als Picosekunden, so rasch, dass hier keine "Gedächtnisstruktur" aufgebaut werden kann.
Ein wesentliches Problem ist zudem die Qualität der homöopathischen Heilmittel. Es handelt sich ja nicht um Reinsubstanzen mit definierter Struktur und Stabilität. Ob die Urtinkturen nun aus Pflanzen (ganzen Pflanzen, Blüten, Samen, Blättern, Wurzeln) oder tierischen Stoffen (z.B. aus Käfern, Insektengiften, Schlangengiften, etc.) hergestellt werden, so handelt es ich um Substanzgemische, die in Art und Quantität der einzelnen Komponenten stark schwanken. Eine für "konventionelle" Arzneistoffe unabdingbare Qualitätskontrolle, ist bei Homöopathika obsolet. In der EU ist nur wichtig, "dass Homöopathika nach den im Europäischen Arzneibuch oder nach einem in den offiziell gebräuchlichen Pharmakopöen der Mitgliedsstaaten der EU beschriebenen homöopathischen Zubereitungsverfahren hergestellt worden sind."
Traurig sieht es auch mit einem Nachweis der Wirksamkeit aus, wenn man eine Evidenz-basierte Medizin zugrunde legt:
Homöopathische Mittel werden nach anderen Kriterien zugelassen
als herkömmliche Arzneimittel. Um für die Zulassung eines neuen Arzneimittels einreichen zu können, müssen normalerweise dessen Wirksamkeit und Sicherheit in mehreren streng regulierten klinischen Studien am Patienten erwiesen sein. Der Nachweis erfolgt in sogenannten randomisierten Doppelblind-Studien, in denen eine statistisch ausreichend große Kontrollgruppe, die ein Placebo erhält, einer vergleichbar zusammengesetzten, mit dem zu prüfenden Therapeutikum (Verum) behandelten Gruppe gegenüber gestellt wird. Doppelblind bedeutet dabei, dass vor Versuchsende weder die Versuchsleiter noch die Teilnehmer wissen wer Placebo und wer die Prüfsubstanz bekommen hat.
Bei Homöopathika ist dies anders. Es besteht hier schon die Möglichkeit einer bloßen Registrierung, wenn das Mittel ohne Angabe eines Anwendungsgebietes in den Verkehr gebracht werden soll, oral oder äußerlich angewendet wird und die Urtinktur (der Ausgangsstoff) mindestens 10 000 fach (D4) verdünnt ist (dass ein derartiges Mittel von einem Anwender bezogen und nach Gutdünken dann für verschiedene Indikationen eingesetzt werden kann, steht auf einem anderen Blatt).
Die Zulassung homöopathischer Mittel erfolgt für eine in den eingereichten Unterlagen angegebene Indikation. Handelt es sich dabei um eine lebensbedrohende Erkrankung, so muss die Wirksamkeit in relevanten klinischen Studien nachgewiesen werden. Allerdings, wie das Deutsche Bundesinstitut für Arzneimittel und Medizinprodukte (BfArM) schreibt, wurde "bislang jedoch noch kein homöopathisches Arzneimittel durch das BfArM zugelassen, bei dem sich der Antragssteller auf eine Studie berufen hätte". https://www.bfarm.de/DE/Aktuelles/Schwerpunktthemen/Homoeopathische-Arzneimittel/_node.html).
Zum Nachweis der Wirksamkeit reicht ansonsten auch anderes "wissenschaftliches Erkenntnismaterial" (d.i. nach wissenschaftlichen Methoden aufbereitetes medizinisches Erfahrungsmaterial). Was darunter zu verstehen ist, ist sehr schwammig: Das BfArM nennt hier den Long-Term-Use (das Mittel muss seit 1978 auf dem Markt sein), Expertenurteile beispielsweise von der Kommission D des BfArM (deren Mitglieder allesamt aus der homöopathischen Ecke kommen), eine bewertete präparatbezogene Literaturübersicht zur Indikations-bezogenen Anwendung des Präparats und auch die homöopathische Arzneimittelprüfung (dabei werden Stoffe in Form von Ausgangsstoffen, Urtinkturen oder Verdünnungsgraden (Potenzen) gesunden Probanden verabreicht und diese berichten -subjektiv - mögliche Symptome). Neben dem wissenschaftlichen Erkenntnismaterial sind auch die medizinischen Erfahrungen zu berücksichtigen
Das Österreichische Bundesamt für Sicherheit im Gesundheitswesen (BASG) geht unisono mit dem deutschen BfArM und verweist auf die Experten der Kommission D. Es schreibt dazu: "Für eine Zulassung muss der Nachweis der spezifischen homöopathischen Wirksamkeit erbracht werden. Dies kann durch klinische Prüfungen oder durch allgemein anerkannte wissenschaftliche Literatur (z.B. Monographien der Kommission D des ehemaligen BGA in Deutschland) erbracht werden."
Dass die Beurteilung zur Zulassung neuer Homöopathika von einer aus Homöopathen bestehenden Kommission abhängt, erscheint zumindest fragwürdig.
Wie steht es nun um die Wirksamkeit homöopathischer Mittel?
Dazu Stellungnahmen von kompetenten Gremien:
Das Informationsnetzwerk Homöopathie
stellt fest: "Ein auf diesen Grundlagen aufbauendes Therapiesystem kann keine auf das eingesetzte Mittel zurückzuführende und über Placeboeffekte hinausgehende Wirksamkeit entfalten." Das Netzwerk ist eine unabhängige offene Arbeitsgruppe von über 60 Experten unterschiedlicher fachlicher Ausrichtung, Biografie und Weltanschauung, die es sich zum Ziel gesetzt haben, die Öffentlichkeit über die Homöopathie zu informieren und so den einseitig positiv überzeichneten Informationen der Anwender und Hersteller zutreffende Informationen entgegenzusetzen.https://netzwerk-homoeopathie.info/
Der unabhängige Online-Service "Medizin-Transparent",
ein Projekt von Cochrane Österreich an der Donau-Universität Krems hat zu den bislang durchgeführten Studien an Homöopathika recherchiert: es geht daraus deutlich hervor, dass in gut gemachten Studien die Homöopathie-Mittel nicht besser wirken als Wirkstoff-freie Scheinmedikamente (Placebos). Dies ist gut belegt u.a. für Asthma, Angsterkrankungen, Durchfall bei Kindern, Kopfschmerzen und Migräne, Erkältungskrankheiten oder etwa dem prämenstruellen Syndrom (PMS) bei Frauen. In vielen anderen Fällen liegen nur minderwertige , nicht nach strengen wissenschaftlichen Kriterien durchgeführte Studien vor. Insgesamt gibt es in bisherigen Studien keinen Hinweis, dass Homöopathie bei irgendeiner Krankheit oder Beschwerde besser helfen könnte als Placebo-Medikamente (https://medizin-transparent.at/streitthema-homoopathie/)
Was die die anfänglich von PharmaSGP gemachten Behauptungen "Unsere Arzneimittel basieren auf natürlichen pharmazeutischen Wirkstoffen mit dokumentierter Wirksamkeit und haben nahezu keine bekannten Nebenwirkungen." betrifft, so hat "Medizin-Transparent" auf Leser- Anfragen zu einigen dieser Behauptungen recherchiert, dazu Forschungs-Datenbanken durchsucht, um alle diesbezüglichen Studien dazu zu finden. Dazu zwei Beispiele
- Taumea, das gegen Schwindel helfen soll, enthält einen Extrakt aus den Früchten der Scheinmyrte (Anamirta cocculus) in homöopathischen Dosierungen (D4). Aussagekräftige wissenschaftliche Belege zur Wirkung gibt es bislang nicht.
- Neradin, ein Extrakt aus der Damiana-Pflanze (Turnera diffusa) wird als Mittel gegen Erektionsstörungen für den Mann beworben. Belegt ist diese behauptete Wirkung von Damiana auf die Potenz jedoch keineswegs. Denn an Menschen wurde sie bisher offenbar nie untersucht.
( Zur Wirksamkeit von Neradin gibt es übrigens einen niederschmetternde Bewertung von Kunden der Internet Apotheke shop-apotheke.at. Von insgesamt 104 Bewertungen haben nur 18 das Produkt als sehr gut befunden, 58 haben sich sehr negativ dazu geäußert.)
Eine neue internationale Studie
unter Leitung von G. Gartlehner hat sich mit den bis 2021 registrierten und publizierten klinischen Studien über Homöopathika befasst und einen besorgniserregenden Mangel an wissenschaftlichen und ethischen Standards auf dem Gebiet festgestellt. "Mehr als ein Drittel der registrierten Homöopathiestudien blieb unveröffentlicht, mehr als die Hälfte der veröffentlichten Studien war nicht registriert worden, und 25 % der primären Ergebnisse wurden verändert oder abgeändert." Die Schlussfolgerung der Forscher: "Homöopathieforscher dürften sich als Rosinenpicker betätigen. Studien mit den erhofften Ergebnissen werden veröffentlicht, um die Behauptungen über die Wirksamkeit der Homöopathie zu untermauern, während diejenigen, die weniger günstig ausfallen, bequem in einer Aktenschublade verschwinden. Fundierte Aussagen über die Wirksamkeit und die Risiken homöopathischer Mittel bleiben so unmöglich. Infolgedessen sind die Beweise für die Wirksamkeit der Homöopathie nach wie vor so dünn wie eine Prise Salz in einem riesigen Meer von Forschungsergebnissen schlechter Qualität." (Gartlehner G, et al., BMJ Evidence-Based Medicine 2022; 27:345–351. doi 10.1136/bmjebm-2021-111846)
Der Wissenschaftsbeirat der Europäischen Akademien (EASAC)
hat - basierend auf den neuesten Arbeiten seiner Mitgliedsakademien - eine Erklärung veröffentlicht, welche die Kritik an den gesundheitlichen und wissenschaftlichen Behauptungen über homöopathische Produkte verstärkt:
- ad Wissenschaftliche Wirkmechanismen: "Wir kommen zu dem Schluss, dass die Behauptungen über Homöopathie unplausibel sind und nicht mit etablierten wissenschaftlichen Konzepten übereinstimmen".
- ad Klinische Wirksamkeit: "Wir bestätigen, dass bei einzelnen Patienten ein Placebo-Effekt auftreten kann. Wir stimmen aber mit früheren umfassenden Bewertungen überein, dass es keine bekannten Krankheiten gibt, für die robuste, reproduzierbare Beweise vorliegen, dass Homöopathie über den Placeboeffekt hinaus wirksam ist. Damit verbunden sind Bedenken hinsichtlich der Informations-getragenen Einwilligung der Patienten und der Sicherheit, wobei letztere mit der schlechten Qualitätskontrolle bei der Herstellung von homöopathischen Arzneimitteln zusammenhängt."
- ad Werbung für die Homöopathie: "Wir weisen darauf hin, dass es dem Patienten erheblichen Schaden zufügen kann, wenn er die Inanspruchnahme einer evidenzbasierten medizinischen Versorgung hinauszögert, und dass generell die Gefahr besteht, das Vertrauen der Öffentlichkeit in die Natur und den Wert wissenschaftlicher Erkenntnisse zu untergraben."
European Academies Science Advisory Council, September 2017: Homeopathic products and practices: assessing the evidence and ensuring consistency in regulating medical claims in the EU https://easac.eu/fileadmin/PDF_s/reports_statements/EASAC_Homepathy_statement_web_final.pdf
Wie soll es nun weiter gehen?
Dazu die Empfehlungen des EASAC aus der oben zitierten Erklärung:
- Es sollte einheitliche regulatorische Anforderungen geben, um die Wirksamkeit, Sicherheit und Qualität aller Produkte für die Human- und Veterinärmedizin aufzuzeigen; diese müssen auf überprüfbaren und objektiven Nachweisen beruhen, welche der Art der Behauptungen entsprechen. In Ermangelung eines solchen Nachweises, sollte ein Produkt von den nationalen Regulierungsbehörden als Arzneimittel weder zugelassen noch registriert werden.
- Evidenz-basierte öffentliche Gesundheitssysteme sollten die Kosten für homöopathische Produkte und Praktiken nicht übernehmen, wenn deren Wirksamkeit und Sicherheit durch strenge Tests nicht nachgewiesen ist.
- Die Zusammensetzung homöopathischer Mittel sollte in ähnlicher Weise angegeben werden wie die anderer Gesundheitsprodukte, d. h. es sollte eine genaue, klare und einfache eine Beschreibung der Inhaltsstoffe und ihrer Mengen in der Rezeptur vorhanden sein.
- Werbung und Marketing für homöopathische Produkte und Dienstleistungen müssen festgelegten Standards von Genauigkeit und Klarheit entsprechen. Werbeaussagen zu Wirksamkeit, Sicherheit und Qualität sollten nicht ohne nachweisbare und reproduzierbare Belege gemacht werden.
Hoffen wir, dass diese Empfehlungen nicht auf taube Ohren stoßen!
Mit CapScan® beginnt eine neue Ära der Darmforschung
Mit CapScan® beginnt eine neue Ära der DarmforschungDo 18.05.2023 — Inge Schuster
> Kenntnisse über das Mikrobiom des Verdauungstrakts und über Stoffwechselprodukte in diesem System beruhen bislang hauptsächlich auf Daten, die aus Stuhlproben erhoben wurden. Mit Hilfe der CapScan®-Kapsel können erstmals auf nicht-invasive Weise Proben aus den Regionen des Dünndarms gesammelt und analysiert werden. Damit werden Aufschlüsse über lebenswichtige Darmaktivitäten möglich, die bisher nicht messbar waren und die zum Verstehen der Stoffwechselwege des Menschen und ihrer komplexen Interaktionen mit dem Mikrobiom in Gesundheit und Krankheit beitragen.
In den letzten Jahrzehnten ist der menschliche Darm und hier vor allem das dort ansässige Mikrobiom mehr und mehr in den Fokus der Forschung gerückt. Es wurde klar, dass die riesige Zahl und Vielfalt der Mikroorganismen eine essentielle Rolle in Gesundheit und Krankheit spielen - dies beginnt mit dem Metabolismus von Nahrung und Fremdstoffen und erstreckt sich über die Regulierung des Immunsystems, den Schutz vor Pathogenen bis hin zum Einfluss auf die Steuerung des Nervensystems und damit auch auf den Gemütszustand.
Der Dünndarm ist noch weitgehend eine terra incognita
Auf Grund des schwierigen Zugangs zu den oberen Teilen des Verdauungstrakts, d.i. dem 5 - 7 Meter langen Dünndarm, stammen Informationen über die Zusammensetzung des Mikrobioms und die im Darm generierten und diesen beeinflussenden Stoffwechselprodukte bislang weitestgehend aus Analysen des Stuhls. Tatsächlich geben solche Proben aber nur ein sehr beschränktes Bild der Vorgänge im Gesamtdarm: beim Eintritt in den aufsteigenden Teil des Dickdarms ist die Verdauung der Nahrung ja nahezu vollständig (zu 90 %) abgeschlossen und die entstandenen Stoffwechselprodukte (Metabolite) wurden bereits durch die Dünndarmwand in den Organismus aufgenommen (absorbiert), teilweise weiter umgewandelt und als Endprodukte in das Darmlumen ausgeschieden. Was im Colon noch ankommt, sind im Wesentlichen unverdauliche Nahrungsbestandteile, die von den dort ansässigen Bakterienkolonien aufgeschlossen und zu Nährstoffen der Darmwand umgebaut werden.
Der Zugang zu den Dünndarmabschnitten Jejunum und Ileum ist eine Herausforderung. Diesbezügliche Proben werden ja nicht unter physiologischen Bedingungen entnommen: sie stammen aus Operationsmaterial und von klinisch toten Organspendern, die mit verschiedenen Arzneimitteln, darunter Antibiotika behandelt worden waren und bestenfalls aus mehrstündigen Endoskopien, die an nüchternen, betäubten oder sedierten Probanden erfolgen.
Wie auch für unterschiedlichste bioaktive Produkte beruhen unsere Kenntnisse beispielsweise zur Vielfalt, Dynamik und Rolle der Gallensäuren bei der Verdauung auf den wenig repräsentativen Messungen der wenigen Prozent der Gallensäuren, die im Stuhl ankommen oder dem Bruchteil eines Prozents im Blut. Fazit: Stuhlproben sind ein unzulängliches Surrogat für die Vorgänge in den oberen Darmabschnitten.
CapScan® ermöglicht erstmals eine nicht-invasive Reise durch den Dünndarm
Ein Forscherteam von den kalifornischen Universitäten Stanford und Davis und dem Spin-off Envivo Bio Inc. hat eine spezielle Kapsel CapScan® entwickelt, die geschluckt wird und in bestimmten Abschnitten des Darms ein kleines Volumen des Darmlumens inklusive der dort vorhandenen Mikroorganismen, Proteine und Metabolite aufnimmt. Zwei zeitgleich am 10. Mai 2023 in den Fachjournalen Nature und Nature Metabolism erschienene Arbeiten beschreiben diese Kapsel beschrieben und berichten über die die damit erhobenen Veränderungen im oberen Darmtrakt während der normalen täglichen Verdauung bei 15 gesunden Menschen [1,2].
Das Prinzip von CapScan®: Die Kapsel - etwa in der Größe einer Vitamin-Pille - ist mit einer magensaftresistenten, pH-sensitiven Polymer-Beschichtung versehen und enthält darin eine kollabierte Blase mit einem Einwegventil. Normalerweise steigt der pH-Wert im Darmlumen von 4-6 im Zwölffingerdarm auf 7-8 im Ileum an und sinkt im anschließenden aufsteigenden Colon etwas ab. Sobald eine Kapsel mit voreingestelltem pH-Wert auf diesen Wert im Lumen trifft, löst sich die Beschichtung mit vorbestimmter Geschwindigkeit auf, die Blase entfaltet sich und kann bis zu 400 µl Lumenflüssigkeit ansaugen. Ist die Blase voll, so verhindert das Ventil, dass auf dem Weiterweg durch den Verdauungstrakt noch Flüssigkeit eintreten kann. Die Kapsel wird schließlich mit dem Stuhl ausgeschieden, daraus hervor geholt und ihr Inhalt analysiert
Die Untersuchungen erfolgten an 15 gesunden Personen im Rahmen einer klinischen Untersuchung. Um Proben aus vier verschiedenen Regionen des Darmtrakts nehmen zu können, nahmen die Versuchspersonen jeweils vier Kapsel-Typen mit unterschiedlichen pH-Sensitivitäten ein, nachdem sie Mahlzeiten ihrer Wahl gegessen hatten. Abbildung.
| Abbildung. 4 Kapsel-Typen, die Proben aus mehreren Regionen des menschlichen Darmtraktes während der normalen Verdauung sammeln. Die Kapseln öffnen sich bei unterschiedlichen, ansteigend höheren pH-Werten. Kapsel-Typ 4 enthält eine zeitverzögerte Beschichtung, um die Sammlung in Richtung des aufsteigenden Dickdarms zu lenken wo der pH-Wert typischerweise im Vergleich zum terminalen Ileum abfällt. (Bild: Ausschnitt aus Dari Shalon et al, 2023 [1]., deutsch beschriftet; Lizenz cc-by). |
Die Analyse: Ein sogenannter "Multiomics"-Ansatz wurde angewandt, um die Proben einerseits auf Bakterien, Viren, Wirtsproteine und andererseits auf Stoffwechselprodukte aus der Nahrung zu analysieren [1, 2]. In den verschiedenen Regionen des menschlichen Darms zeigten sich dabei signifikante, zum Teil dramatische Unterschiede in der Zusammensetzung des Mikrobioms, des Wirtsproteoms und in der Induktion von Prophagen. Auch hinsichtlich der Zusammensetzung der Stoffwechselprodukte - die Forscher identifizierten insgesamt nahezu 2000 Metaboliten - gab es massive Unterschiede vom oberen (proximalen) zum unteren (distalen) Dünndarm und zu Stuhlproben und (am Beispiel von Obst und Alkohol) interessante Zusammenhänge zwischen Ernährung und Metaboliten.
Fazit
Mit CapScan® kann nicht nur eine neue Ära in der Forschung des Darmmikrobioms beginnen, die Kapsel kann auch breite Anwendung in der Forschung und Entwicklung von Arzneistoffen finden. Ein erstes Beispiel ist die auf der US-Plattform für Klinische Studien (ClinicalTrials.gov) gelistete Phase 1 Studie: "Evaluierung der CapScan-Vorrichtung zur Messung des Sulfasalazin-Stoffwechsels." (Sulfasalazin wird zur Behandlung von chronisch-entzündlichen Darmerkrankungen eingesetzt.) Dabei geht es darum die regionale Verteilung von Sulfasalazin, die metabolischen Abbauprodukte von Sulfasalazin und die Darmmikrobiota im Verdauungstrakt gesunder Freiwilliger zu charakterisieren. Die von Envivo Bio Inc. geleitete Studie an 10 gesunden Freiwilligen ist vor Kurzem zu Ende gegangen; sie wurde von den NIH unterstützt, die auch weiterhin die Entwicklung und klinische Evaluierung von CapScan fördern wollen. Für ein anderes Projekt, eine klinische Studie über die "Auswirkungen des Darmmikrobioms auf die Darmgesundheit von Müttern und Kindern in Ländern mit niedrigem und mittlerem Einkommen" die von Envivo in Zusammenarbeit mit Forschern von Stanford Medicine durchgeführt wird, hat die Bill and Melinda Gates Foundation Mittel für zur Verfügung gestellt.
[1] Dari Shalon et al, Profiling the human intestinal environment under physiological conditions, Nature (2023). DOI: 10.1038/s41586-023-05989-7
[2] Jacob Folz et al, Human metabolome variation along the upper intestinal tract, Nature Metabolism (2023). DOI: 10.1038/s42255-023-00777-z
Mit CapScan könnten auch wesentliche Fragen zum Überleben von Probioika während der Darmpassage und zu ihrer Wirksamkeit gelöst werden. Dazu:
Inge Schuster, 15.01.2023: Probiotika - Übertriebene Erwartungen?
Über die unterirdischen Verbindungen zwischen den Bäumen
Über die unterirdischen Verbindungen zwischen den BäumenDo. 11.05.2023 — IIASA
Pilze bilden Netzwerke, welche die Bäume in einem Wald miteinander verbinden, diese Pilzgeflechte sind ein zentrales Element, das für den Zustand der Wälder ausschlaggebend ist und dafür, wie diese auf den Klimawandel reagieren. Laut der zunehmend populärer werdenden "Mutterbaum-Hypothese" werden solche Netze auch als Mittel betrachtet, mit dem Bäume ihren Jungbäumen und anderen "befreundeten" Bäumen nützen. In einer kürzlich erschienenen Studie hat eine internationale Forschergruppe die Hinweise, die für und gegen diese Hypothese sprechen, erneut überprüft.*
Die Bäume in einem Wald sind durch fadenförmige Strukturen, den so genannten Hyphen, von symbiotischen Pilzen miteinander verbunden; zusammen bilden diese ein unterirdisches Netzwerk, das so genannte Mykorrhiza-Netzwerk. Wie allgemein bekannt, können Mykorrhizapilze den Bäumen Nährstoffe im Austausch für den von den Bäumen gelieferten Kohlenstoff liefern.
Die Mutterbaum-Hypothese .............
Die so genannte Mutterbaum-Hypothese geht darüber hinaus und impliziert einen vollkommen neuen Zweck dieser Netzwerke. Über das Netzwerk teilen die größten und ältesten Bäume, die auch als Mutterbäume bezeichnet werden, Kohlenstoff und Nährstoffe mit Jungbäumen, die in besonders schattigen Bereichen wachsen und selbst nicht genügend Sonnenlicht für eine ausreichende Photosynthese erhalten. Die Netzwerkstruktur soll es den Mutterbäumen auch möglich machen, den schlechten Gesundheitszustand ihrer Nachbarn durch deren Notsignale zu erkennen und gemahnt zu werden diesen Bäumen die Nährstoffe zu schicken, die diese benötigen, um sich wieder zu erholen. Auf diese Weise - so die Hypothese - sollen die Mutterbäume als zentrale Knotenpunkte fungieren, die sowohl mit jungen Setzlingen als auch mit anderen großen Bäumen in ihrer Umgebung kommunizieren, um deren Überlebenschancen zu erhöhen.
Dies ist ein sehr ansprechendes Konzept, das nicht nur das Interesse der Wissenschaftler anspricht, sondern auch die Medien, in denen diese Hypothese dann oft als Tatsache dargestellt wird. In der soeben in der Zeitschrift New Phytologist veröffentlichten Studie [1] ist - nach Meinung der Autoren - die Hypothese jedoch nur schwer mit einer Theorie in Einklang zu bringen; dies hat die Forscher dazu veranlasst Daten und Schlussfolgerungen aus wissenschaftlichen Untersuchungen, die für und gegen die Mutterbaum-Hypothese sprechen, nochmals einer Prüfung zu unterziehen.
...............und was gegen diese Hypothese spricht
| Abbildung 1. Die Mutterbaum-Hypothese und Feldstudien in borealen Wäldern .(a) Stammvolumen von Waldkiefern (Pinus sylvestris) in der Umgebung von alten Bäumen, die bei der vor 20 und mehr Jahren erfolgten Schlägerung erhalten geblieben waren: im Umkreis von weniger als 5 m von den alten Bäumen wiesen die neuen Bäume ein um 24 % geringeres Wachstum als weiter entfernte Bäume. Das hypothetische Wachstumsmuster des jüngeren Bestands im Falle von Konkurrenz bzw. Förderung durch den großen Baum ist orangefarben bzw. blau markiert. (Daten: Jakobsson (2005) [2]). , (b) Kartierung der Waldkiefern in einem Bestand in Südfinnland,(100 x 100 m). Die älteren Bäume sind durch geschlossene Kreise gekennzeichnet (Größen entsprechen den Stammdurchmesser-Klassen), die jüngeren Bäume durch offene Kreise. Der Bereich innerhalb eines Radius von 5-6 m um die älteren Bäume wurde blau schraffiert, um die geringe Dichte der jüngeren Bäume im Vergleich zu den Bereichen ohne alte Bäume hervorzuheben. (Bild aus Aaltonen (1926) [3] mit Genehmigung von Oxford Academic Press nachgedruckt). , (c) Nach Südwesten ausgerichteter Rand eines borealen Kiefernbestands und der verjüngte Bestand daneben (Schweden, 64°N). Man beachte das schwache Wachstum in der Nähe der größeren Bäume. (Abbildung: Fig. 1 aus N. Henriksson et al., 2023, [1] von Redn. deutsch beschriftet. Lizenz: cc-by-nc.) |
Die unter der Leitung von Nils Henriksson von der Schwedischen Universität für Agrarwissenschaften ausgeführte Studie ergab, dass tatsächlich nur sehr limitierte empirische Belege für die Mutterbaum-Hypothese vorliegen, und theoretische Beweisführungen für die Mechanismen weitgehend fehlen [1].
Während große Bäume und deren Verbindungen zu ihren Nachbarn für das Ökosystem Wald nach wie vor essentiell sinnd, agiert das Pilznetzwerk nicht als eine einfache Pipeline, um Ressourcen unter den Bäumen zu aufzuteilen. In anderen Worten: der offensichtliche Austausch von Ressourcen zwischen den Bäumen ist eher das Ergebnis eines Handels zwischen Pilzen und Bäumen, als ein gezielter Austausch von einem Baum zum anderen. Sehr oft führt dies eher zu einer Verschärfung des Wettbewerbs zwischen den Bäumen, als zur Unterstützung der Stecklinge. Abbildung 1.
"Wir haben herausgefunden, dass Mykorrhiza-Netzwerke in der Tat für die Stabilität vieler Waldökosysteme unabdingbar sind, aber nur selten für ein Teilen und Hilfeleisten zwischen den Bäumen. Vielmehr funktioniert es wie ein Handelsplatz für einzelne Bäume und Pilze, die jeweils versuchen, das beste Geschäft zu machen, um zu überleben", erklärt Oskar Franklin, einer der Studienautoren und Forscher in der Forschungsgruppe Landwirtschaft, Forstwirtschaft und Ökosystem-Dienstleistungen des IIASA-Biodiversity and Natural Resources Program. "Der Wald ist kein Superorganismus oder eine Familie von Bäumen, die sich gegenseitig unterstützen. Er ist ein komplexes Ökosystem mit Bäumen, Pilzen und anderen Organismen, die alle voneinander abhängig sind, aber nicht von einem gemeinsamen Ziel geleitet werden." Abbildung 2.
| Abbildung 2. Alternative Wege des Kohlenstoff-Transfers von einem hypothetischen Donor-Baum zu einer Empfänger-Jungpflanze. EcM (Ectomycorrhiza): symbiotische Beziehung zwischen Pilzsymbionten und den Wurzeln von Pflanzen. Die blauen Pfeile stellen Mechanismen für die potenzielle Verteilung von Kohlenstoff aus dem Donor-Baum in den Boden dar, die keine gemeinsamen Mykorrhiza-Netzwerke (CMN) zwischen den Pflanzen erfordern. Zelluläre Atmung des Spenderbaums und seiner Mykorrhiza-Pilze geben in beiden Fällen Kohlenstoff (C) in Form von CO2 in den Boden ab, das in den Wurzeln des aufnehmenden Keimlings oder über die Photosynthese fixiert wird. Die Ausscheidungen der Feinwurzeln der Spenderbäume und des Mykorrhiza-Myzels setzen organische Verbindungen wie Kohlenhydrate, organische Säuren und Sekundärmetaboliten im Boden frei. Der Umsatz von Spenderbaum-C in Wurzeln und Mykorrhiza-Myzelien, Assimilation und Umsatz von Spender-C durch saprotrophe (d.i. vom Abbau organischer Verbindungen toter Organismen lebende) Pilze, Bakterien und Archaeen, sowie die Umverteilung durch diese und andere Lebensformen im Boden können dem Mykorrhiza-Myzel des aufnehmenden Setzlings ebenfalls C zur Verfügung stellen. Die Kettenräder-Symbole stehen an kritischen Kontrollpunkten des C-Transfers über CMN -Verbindungen. (Bild: Fig. 4. aus N. Henriksson et al., 2023, [1] von Redn. deutsch beschriftet. Lizenz: cc-by-nc.) |
"Wenn auch die Mutterbaum-Hypothese wissenschaftlich kaum belegt und in der wissenschaftlichen Gemeinschaft umstritten ist, hat sie sowohl die Forschung als auch das öffentliche Interesse an der Komplexität der Wälder geweckt. Es ist von entscheidender Bedeutung, dass die künftige Bewirtschaftung und Untersuchung der Wälder die tatsächliche Komplexität dieser wichtigen Ökosysteme berücksichtigt", so Franklin abschließend.
[1] Henriksson, N., Marshall, J., Högberg, M.N., Högberg, P., Polle, A., Franklin, O., Näsholm, T. (2023). Re-examining the evidence for the mother tree hypothesis - resource sharing among trees via ectomycorrhizal networks. New Phytologist DOI: 10.1111/nph.18935
[2] Jakobsson R. 2005. Effect of retained trees on the development of young Scots pine stands in Southern Finland. Doctoral thesis, Acta Universitatis Agriculturae Sueciae.
[3] Aaltonen VT. 1926. On the space and arrangement of trees and root competition. Journal of Forestry 24: 627–644.
* Der am 9. Mai 2023 als Presseaussendung auf der IIASA-Webseite unter dem Titel " Exploring the underground connections between trees" erschienene Artikel Artikel https://iiasa.ac.at/news/may-2023/exploring-underground-connections-between-trees wurde von der Redaktion möglichst wortgetreu übersetzt und durch 2 Abbildungen plus Legenden aus der zugrundeliegenden Veröffentlichung [1] ergänzt. IIASA hat freundlicherweise der Veröffentlichung der von uns übersetzten Inhalte seiner Website und Presseaussendungen in unserem Blog zugestimmt.
Ähnliche Inhalte im ScienceBlog
Gerd Gleixner, 05.08.2021: /Erdoberfläche - die bedrohte Haut auf der wir leben
Christian Körner, 29.07.2016: Warum mehr CO₂ in der Atmosphäre (meistens) nicht zu einem Mehr an Pflanzenwachstum führt
Knut Ehlers, 01.04.2016: Der Boden ein unsichtbares Ökosystem
Gerhard Glatzel, 04.04.2013: Rückkehr zur Energie aus dem Wald — mehr als ein Holzweg? (Teil 2)
Überfischen hat das natürliche Gleichgewicht der marinen Lebensformen aus dem Lot geraten lassen
Überfischen hat das natürliche Gleichgewicht der marinen Lebensformen aus dem Lot geraten lassenDo, 04.05.2023 — Tim Kalvelage
Fischereiprodukte leisten einen wichtigen Beitrag zur globalen Ernährungssicherheit. Derzeit stammen 17 Prozent des tierischen Eiweißes, das weltweit verzehrt wird, aus dem Ozean oder aus Süßgewässern und der Fischkonsum nimmt weiter zu. Meere sind aber keine unendlichen Speisekammern. Die Nutzung der marinen Ressourcen durch den Menschen hat die Grenzen der Nachhaltigkeit längst überschritten; wie Berechnungen zeigen, wurde die Biomasse der Fische und kleineren Meeressäuger bereits um 60 % dezimiert, die von Walen sogar um 90 %. Der Meeresmikrobiologe und Wissenschaftsjournalist Dr. Tim Kalvelage gibt einen Überblick über die Auswirkungen der industriellen Fischerei auf das marine Ökosystem.*
Vor der ostkanadischen Insel Neufundland, dort wo der warme Golfstrom auf den kalten Labradorstrom trifft, liegt einer der reichsten Fischgründe der Erde: die Grand Banks, eine Reihe flacher Unterwasserplateaus auf dem nordamerikanischen Kontinentalschelf. Berühmt wurde die Region einst für ihre riesigen Kabeljaubestände. Schon bevor Kolumbus Amerika entdeckte, segelten baskische Fischer wegen des Kabeljaus quer über den Atlantik. Ab Mitte des letzten Jahrhunderts plünderten immer größere Trawler – vor allem aus Europa und der Sowjetunion – die Fischreichtümer. Auf dem Höhepunkt wurden an den Grand Banks im Jahr 1968 mehr als 800.000 Tonnen Kabeljau gefangen. In den Folgejahren kollabierten die Bestände, sodass Kanadas Regierung 1992 ein Fangverbot verhängte. 40.000 Menschen in den Provinzen Neufundland und Labrador verloren ihre Jobs in der Fischerei.
Trotz mehrjährigen Fangverbots und noch immer drastisch reduzierter Fangquoten haben sich die Kabeljaubestände an den Grand Banks bis heute nicht erholt. Stattdessen ist der Zusammenbruch der dortigen Kabeljaufischerei zum Symbol geworden für die Ausbeutung der Ozeane durch den Menschen und den Rückgang vieler Fischpopulationen. In den vergangenen Jahrzehnten sind die weltweiten Fangflotten in immer entlegenere Gebiete vorgedrungen und haben ihre Netze und Leinen in immer größeren Tiefen ausgebracht, um die Nachfrage nach Fisch und anderen Meerestieren zu bedienen.
Nahrungslieferant Ozean
Fischfang und das Sammeln von Meeresfrüchten wie Muscheln spielen seit Tausenden von Jahren eine wichtige Rolle für die Ernährung der Küstenbewohner der Erde. Im Laufe der Zeit wurden ihre Boote besser und die Netze größer und sie wagten sich immer weiter aufs Meer hinaus. Im Mittelalter florierte der Handel mit getrocknetem und gesalzenem Fisch, etwa rund um die Ostsee oder zwischen Westeuropa und Nordamerika. Angesichts der endlosen Weiten der Ozeane und der riesigen Fischschwärme schienen die lebenden marinen Ressourcen einst unerschöpflich.
Dann kam die Industrielle Revolution: Dampfschiffe, die schneller waren als Segelschiffe und weniger abhängig von Wind oder Gezeiten, eroberten die Weltmeere. Mitte des 19. Jahrhunderts wurde die Harpunenkanone erfunden und das große Schlachten der Wale begann. Manche Arten waren binnen weniger Jahrzehnte nahezu ausgerottet.
Nach dem 2. Weltkrieg schließlich ermöglichten hochseetaugliche Kühlschiffe, elektronische Navigationssysteme und Echolote den Aufstieg der industriellen Fischerei. Heute werden laut der Welternährungsorganisation (Food and Agriculture Organization – FAO) von den rund 30.000 Fischarten im Ozean mehr als 1.700 kommerziell genutzt. Sie landen auf unseren Tellern oder werden als Pellets in der Aquakultur und der Landwirtschaft verfüttert. Für Milliarden von Menschen sind sie eine essenzielle Proteinquelle: 17 Prozent des tierischen Eiweißes, das weltweit verzehrt wird, stammen aus dem Ozean oder aus Süßgewässern. Fischereiprodukte leisten damit einen wichtigen Beitrag zur globalen Ernährungssicherheit.
Seit der Mitte des 20. Jahrhunderts ist der Hunger nach Fisch und Meeresfrüchten jedoch stark gestiegen: Die Weltbevölkerung hat sich seither mehr als verdreifacht, und pro Kopf wird heute die doppelte Menge an Fisch gegessen – ca. 20 Kilogramm jährlich. Fangstatistiken und wissenschaftliche Studien zeigen eindringlich, dass die Nutzung der marinen Ressourcen durch den Menschen die Grenzen der Nachhaltigkeit längst überschritten hat. Vielerorts wird dem Ozean mehr Fisch entnommen, als nachwachsen kann.
Räuber und Beute
Für ein besseres Verständnis der Produktivität der Ozeane hilft ein Blick auf die marinen Nahrungsbeziehungen und den damit verbundenen Transfer von Kohlenstoff und Energie. Betrachtet man ausgewählte Organismen in einem Ökosystem – etwa in einem Korallenriff – dann lassen sich ihre Nahrungsbeziehungen als lineare Nahrungsketten darstellen. In Wirklichkeit jedoch sind Fische, Wirbellose und andere Meerestiere in komplexen Nahrungsnetzen miteinander verknüpft (Abbildung 1). Denn viele besitzen ein breites Beutespektrum und stehen selbst auf dem Speisezettel verschiedener Räuber, mit denen sie zum Teil um Nahrung konkurrieren. Je nach Futterquelle besetzen Organismen unterschiedliche Ernährungsstufen – oder trophische Ebenen.
|
Abbildung 1: Marines Nahrungsnetz © World Ocean Review Nr. 2, maribus gGmbH, Hamburg 2013 |
Auf der niedrigsten Stufe stehen die Primärproduzenten, die Kohlenstoffdioxid und Wasser mittels Photosynthese in Zucker und Sauerstoff umwandeln. Sie ernähren direkt oder indirekt alle Konsumenten. Die wichtigsten Produzenten im Ozean sind frei schwebende Mikroalgen und Cyanobakterien: das Phytoplankton. Es bildet die Nahrungsgrundlage für Zooplankton – kleine Krebstiere, Fischlarven und Quallen, die mit der Strömung treiben. Krill und anderes Zooplankton wiederum landen in den Mäulern von Heringen, Makrelen, Walhaien und Blauwalen. Höhere trophische Ebenen gehören schnellen Räubern wie Schwert- und Thunfischen oder Delfinen, die Schwarmfische erbeuten. Am Ende der Nahrungskette jagen Weiße Haie, Orcas und Pottwale – Spitzenprädatoren, die keine natürlichen Feinde haben.
Muster im Meer
Forschende um den Biologen Ian Hatton vom Leipziger Max-Planck-Institut für Mathematik in den Naturwissenschaften haben untersucht, wie zahlreich Plankton Fische und Meeressäuger im Ozean sind und welche Masse sie auf die Waage bringen. Dabei machten die Forschenden eine verblüffende Entdeckung. Ausgangspunkt der Studie waren Beobachtungen, die kanadische Wissenschaftler bereits im Jahr 1972 gemacht hatten. Diese hatten in Wasserproben aus dem Atlantik und Pazifik rund um Nord- und Südamerika Plankton gezählt und dessen Größe bestimmt. Dabei stellten sie fest, dass Organismen umso häufiger vorkommen, je kleiner sie sind. Viel erstaunlicher jedoch war: Wenn sie das Plankton in logarithmische Größenklassen einteilten (1-10 µm, 10-100 µm usw.), dann entfiel auf jede Größenklasse der gleiche Anteil der Planktonbiomasse. In anderen Worten: Größere Organismen machen ihre zahlenmäßige Unterlegenheit gegenüber kleineren durch ein höheres Gewicht wett. Auf Basis der Ergebnisse formulierten sie die kühne Hypothese, dass sich dieses Muster in den Weltmeeren von mikroskopischen Einzellern bis hin zu riesigen Walen erstreckt. „Allerdings war die Hypothese bisher nie getestet worden“, erklärt Hatton, „denn lange Zeit fehlte es dafür an Daten.“
Fünf Jahrzehnte später gab es genug Daten um zu überprüfen, ob die Verteilung der marinen Biomasse tatsächlich diesem Muster folgte. Hatton und sein Team berechneten zunächst die Biomasse für die Zeit vor 1850, als die Meere noch relativ unberührt waren. Die weltweite Menge an Phytoplankton schätzten sie anhand von Satellitendaten ab, die heute routinemäßig zur Bestimmung der Primärproduktion im Ozean genutzt werden. Hunderttausende Wasserproben, die über Jahrzehnte rund um den Globus gesammelt worden waren, lieferten Zahlen für Zooplankton und Bakterien. Beim Plankton nahmen die Forschenden an, dass die Menge seit der Industriellen Revolution konstant geblieben ist. „Die größte Herausforderung war eine Abschätzung der Fischbiomasse“, sagt Hatton. „Fische sind schwer zu erfassen, da sie wandern, Netzen entgehen und konzentriert in Schwärmen auftreten.“ Letztlich wurden die historischen Fischbestände anhand von weltweiten Fangdaten und mithilfe von Computermodellen ermittelt. Für Robben, Wale und andere Meeressäuger griff das Team auf regelmäßige Tierzählungen zurück sowie auf Schätzungen, die in die Vergangenheit extrapoliert wurden.
|
Abbildung 2: Ozean aus dem Gleichgewicht. Die 23 Gewichtsklassen mariner Organismen sind als Säulen dargestellt. Die Farbender Säulen entsprechen dem relativen Anteil der jeweiligen Gruppe. Der schraffierte Bereich (pink) zeigt, wie stark der Mensch inzwischen die Bestände der großen Meeresbewohner durch Fischerei und Walfang reduziert hat. © I. Hatton, MPI für Mathematik in den Naturwissenschaften / CC-BY-NC-SA 4.0 |
Nachdem die Forschenden die Anzahl der Lebewesen und deren Biomasse für den globalen Ozean beziffert hatten, teilten sie diese in logarithmische Gewichtsklassen (1-10 g, 10-100 g usw.) ein. Das Körpergewicht mariner Organismen umfasst 23 Größenordnungen, vom weniger als ein Billionstel Gramm schweren Bakterium bis hin zum mehr als 100 Tonnen schweren Blauwal. In der Tat bestätigte sich die 50 Jahre alte Hypothese:
Bevor der Mensch das Ökosystem Meer weitreichend veränderte, war die Biomasse über alle Größenklassen hinweg erstaunlich konstant – zumindest in den produktiven obersten 200 Metern der Wassersäule. Damals betrug die Biomasse je Gewichtsklasse rund eine Milliarde Tonnen; nur an den Enden des Größenspektrums, bei Bakterien und Walen, wichen die Werte nach oben bzw. unten ab (Abbildung 2). Worauf die Regelmäßigkeit beruht, sei noch nicht klar, so Hatton. „Möglicherweise hängt es mit dem Kohlenstoff- und Energietransfer entlang der Nahrungskette zusammen, wie viele Forschende annehmen.“ Auch der Stoffwechsel, das Wachstum und die Fortpflanzung mariner Organismen könnten eine Rolle spielen. Klar ist jedoch: „Der Mensch hat dieses Naturgesetz der Meere gebrochen.“ Das sagt Hatton beim Blick auf die heutige Verteilung der Biomasse im Ozean. Die Studie offenbart einen dramatischen Rückgang für das obere Drittel des Größenspektrums gegenüber dem vorindustriellen Zeitalter (Abbildung 2). Seit 1800 hat die Biomasse von Fischen und Meeressäugern um rund zwei Milliarden Tonnen abgenommen, das entspricht einem Verlust von 60 Prozent. Bei den größten Walen beträgt er sogar fast 90 Prozent. Man schätzt, dass allein im 20. Jahrhundert knapp drei Millionen Tiere getötet wurden, unter anderem zur Gewinnung von Lampenöl, Margarine oder Nitroglycerin für Munition. Zwar wird heute kein kommerzieller Walfang mehr praktiziert – mit Ausnahme von Island, Japan, und Norwegen. Doch viele Populationen sind weit entfernt von ihrer einstigen Biomasse. Auch die Auswirkungen der industriellen Fischerei auf das Ökosystem sind unübersehbar. Insbesondere große Räuber auf hohen trophischen Ebenen wie Schwert- und Thunfische oder Haie sind vielfach verschwunden.
Weiße Flecken in der Fangstatistik
In ihrem aktuellen Fischereibericht beziffert die FAO die weltweite Produktion von Fisch und Meeresfrüchten für das Jahr 2020 auf knapp 180 Millionen Tonnen (Abbildung 3). Nahezu die Hälfte davon waren Wildfische aus dem Meer. Fast jeder zweite Speisefisch stammt heute aus der Aquakultur und wächst in Zuchtteichen oder in Käfigen im Meer auf. Aquakultur ist der am schnellsten wachsende Sektor der Nahrungsmittelproduktion und hat in der Vergangenheit oft zu großflächigen Umweltzerstörungen geführt. Für die Garnelenzucht etwa werden in Südostasien Mangrovenwälder abgeholzt, die wichtige Kinderstuben für Fische sind und Küsten vor Erosion schützen. Auch der Bedarf an Futterfischen und die Gewässerbelastung durch Futterreste und Fischkot sind ein Problem.
Die Fangstatistik der FAO basiert auf den offiziellen Fischereidaten, die Staaten an die UN-Behörde übermitteln. Wie hoch die tatsächlichen Fangmengen sind, ist unklar. Forschende des Projekts „Sea Around Us“ schätzen, dass ein Viertel aller gefangenen Meeresfische nicht in der FAO-Statistik auftaucht, weil sie illegal angelandet, nicht berichtet oder als Beifang wieder über Bord geworfen wurden. Zudem fehlen in der FAO-Statistik die Fangmengen kleiner Schwarmfische wie Sardinen und Heringe, die als Futtermittel wie Fischmehl oder Fischöl enden. Dabei machen diese geschätzte 25 Prozent der globalen Fangmenge aus.
|
Abbildung 3: Entwicklung von Fischfang und Aquakultur. Im Jahr 2020 wurden weltweit 90 Millionen Tonnen Meerestiere gefangen und 88 Millionen Tonnen Fischereiprodukte in Aquakultur produziert. Von der Gesamtproduktion entfallen 63 Prozent auf die Meere und 37 Prozent auf Binnengewässer. © Verändert nach FAO: The State of World Fisheries and Aquaculture, 2022 |
Nachhaltige Nutzung der Meere
Welche Maßnahmen könnten den Rückgang der Fischbestände aufhalten und dennoch langfristig hohe Fischereierträge sichern, um die wachsende Weltbevölkerung zu ernähren? Experten fordern seit langem, rund ein Drittel der Meere als Schutzgebiete auszuweisen, besonders artenreiche Regionen sowie wichtige Laichgründe und Kinderstuben. Durch Abwanderung in benachbarte, nicht geschützte Gebiete könnten sich die dortigen Populationen erholen. Gleichzeitig sollten Bestände nicht bis an ihre Belastungsgrenze ausgebeutet werden, damit es nicht zum Kollaps kommt, wenn sich Umweltbedingungen verändern und die Reproduktionsrate sinkt. Nachhaltiges Fischereimanagement beinhaltet, Zielarten nicht isoliert zu betrachten, sondern auch ihre Rolle im Ökosystem zu berücksichtigen. Zum Beispiel verschwinden große Raubfische, wenn ihre Beute zu stark befischt wird. Mehr als 95 Prozent des globalen Fischfangs findet innerhalb von 200 Seemeilen vor den Küsten statt (in der sog. Ausschließlichen Wirtschaftszone). In vielen Ländern müssten Fischereigesetze verschärft und die illegale Fischerei stärker verfolgt werden.
Fortschritte gibt es beim Schutz der Hochsee, die sich an die 200-Seemeilen-Zone anschließt. Im Frühjahr 2023 haben sich die Vereinten Nationen nach jahrelangen Verhandlungen auf ein Abkommen geeinigt. Bisher war dieses riesige Gebiet, das fast 60% der Weltmeere umfasst, ein nahezu rechtsfreier Raum. Geplant ist, mindestens 30 Prozent der Weltmeere unter Schutz zu stellen. Wirtschaftliche Projekte, Expeditionen und andere Aktivitäten in den Meeren sollen zukünftig auf ihre Umweltverträglichkeit geprüft und die biologische Vielfalt der Hochsee unter international verbindlichen Schutz gestellt werden. Jetzt kommt auf es auf die schnelle und ernsthafte Umsetzung in den Mitgliedsländern an.
* Der Artikel von Tim Kalvelage ist unter dem Titel: "Überfischte Meere - Leben im Ozean aus der Balance" im Geomax 27-Heft der Max-Planck-Gesellschaft im Frühjahr 2023 erschienen (https://www.max-wissen.de/max-hefte/geomax-27-ueberfischte-meere/). Mit Ausnahme des Titels wurde der unter einer cc-by-nc-sa Lizenz stehende Artikel unverändert in den Blog übernommen.
Weiterführende Links:
World Ocean Review 2021 - Lebensgarant Ozean – nachhaltig nutzen, wirksam schützen. https://worldoceanreview.com/de/wor-7/
Meeresschutz. https://www.bmuv.de/themen/wasser-ressourcen-abfall/meeresschutz
Fischereibericht der FAO, 2022 (engl.) https://www.fao.org/publications/home/fao-flagship-publications/the-state-of-world-fisheries-and-aquaculture/en
NASA-Weltraummission erfasst Kohlendioxid-Emissionen von mehr als 100 Ländern
NASA-Weltraummission erfasst Kohlendioxid-Emissionen von mehr als 100 LändernDo, 27.04.2023 — Redaktion
Im Rahmen eines Pilotprojekts der NASA wurden die Emissionen von Kohlendioxid und seine Entfernung aus der Atmosphäre in mehr als 100 Ländern anhand von Satellitenmessungen bestimmt. Das Pilotprojekt OCO-2 bietet erstmals einen aussagekräftigen Einblick auf das in diesen Ländern emittierte Kohlendioxid, und darauf, wie viel davon durch Wälder und andere kohlenstoffabsorbierende "Senken" innerhalb ihrer Abgrenzungen aus der Atmosphäre entfernt wird. Die Länder arbeiten daran ihre Klimaziele zu erreichen - aus den Befunden der Studie geht klar hervor, wie Weltraum-basierte Instrumente die auf der Erde erhobenen Erkenntnisse unterstützen können.*
Top-Down-Ansätze
Die internationale Studie wurde von mehr als 60 Forschern durchgeführt und hat Messungen der NASA-Mission Orbiting Carbon Observatory-2 (OCO-2) sowie ein Netzwerk von Erdoberflächen-basierten Beobachtungen genutzt, um die Zu- und Abnahmen der atmosphärischen Kohlendioxidkonzentration von 2015 bis 2020 zu quantifizieren [1]. (Zum OCO-2-Projekt: siehe Box.)
Mithilfe dieses auf Messungen basierenden (oder "Top-Down"-) Ansatzes konnten die Forscher dann auf die Kohlendioxid-Bilanz von Emissionen und Entfernungen aus der Atmosphäre schließen. Abbildung 1.
Wenn auch die OCO-2-Mission nicht speziell auf die Bestimmung der Emissionen einzelner Länder ausgerichtet war, so kommen die Ergebnisse aus den über 100 Ländern zu einem recht günstigen Zeitpunkt: Die erste globale Bestandsaufnahme - ein Prozess zur Bewertung des kollektiven globalen Fortschritts bei der Begrenzung der im Pariser Abkommen von 2015 festgelegten globalen Erwärmung - findet 2023 statt.
| Abbildung 1. Datenvisualisierung der Emissionen (Quellen) und Entnahmen (Senken) von Kohlendioxid (CO₂) für mehr als 100 Länder auf der ganzen Welt. Die Karte rechts zeigt die mittleren Nettoemissionen und Nettoentnahmen von Kohlendioxid aus der Atmosphäre von 2015 bis 2020. Länder, in denen mehr Kohlendioxid entfernt als emittiert wurde, erscheinen als grüne Vertiefungen, während Länder mit höheren Emissionen hellbraun bis dunkelbraun sind und aus dem Bild herauszuragen scheinen. Diese Befunde beruhen auf Messungen des atmosphärischen CO₂, die von einem Netzwerk bodengestützter Standorte und dem Orbiting Carbon Observatory 2 (OCO-2) der NASA durchgeführt wurden, das CO₂ seit 2014 rund um den Globus kartiert. (Das Bild wurde von Redn. deutsch beschriftet; Quelle: NASA's Scientific Visualization Studio; Lizenz cc-by.) |
"Die NASA konzentriert sich auf die Bereitstellung geowissenschaftlicher Daten, die sich mit realen Klimaproblemen befassen - etwa indem weltweit Regierungen geholfen wird die Auswirkungen ihrer Bemühungen zur Verringerung des Kohlenstoffausstoßes zu messen", sagt Karen St. Germain, Direktorin der Earth Science Division der NASA im NASA-Hauptquartier in Washington. "Dies ist ein Beispiel dafür, wie die NASA bestrebt ist die Messung von Kohlenstoffemissionen in einer Weise zu entwickeln und verbessern, die den Bedürfnissen der Nutzer entspricht."
Herkömmliche Aktivitäten-bezogene Ansätze,
solche "Bottom-Up"-Ansätze zur Kohlenstoffbestimmung beruhen auf dem Aufrechnen und Abschätzen wie viel Kohlendioxid in allen Sektoren einer Volkswirtschaft, wie im Transportsektor und in der Landwirtschaft, emittiert werden. Bottom-Up-Bestandsaufnahmen von Kohlenstoff sind von entscheidender Bedeutung für die Bewertung von Fortschritten bei der Emissionsreduzierung; ihre Erstellung erfordert allerdings beträchtliche Ressourcen, Expertise und Kenntnisse über das Ausmaß der relevanten Aktivitäten.
Deshalb ist - nach Meinung der Autoren - die Entwicklung einer Datenbank über Emissionen und deren Entfernungen mittels eines Top-Down-Ansatzes als besonders hilfreich für Länder anzusehen, die nicht über die üblichen Ressourcen für die Erstellung von Bestandsaufnahmen verfügen [1]. Tatsächlich enthält die gegenwärtige Studie Daten für mehr als 50 Länder, die zumindest in den letzten 10 Jahren keine Emissionen gemeldet haben.
Die Studie bietet eine neue Perspektive, indem sie sowohl die Emissionen fossiler Brennstoffe als auch die Veränderungen des gesamten Kohlenstoffbestands in Ökosystemen, einschließlich Bäumen, Sträuchern und Böden erfasst. Abbildung 2.
| Abbildung 2. Bestandsaufnahme von CO2-Emissionen und Entfernungen aus der Atmosphäre. CO2 wird der Atmosphäre durch Photosynthese entzogen und dann durch eine Reihe von Prozessen wieder in die Atmosphäre abgegeben. Drei Prozesse verlagern den Kohlenstoff lateral auf der Erdoberfläche, so dass die CO2-Emissionen in einer anderen Region schlagend werden als die CO2-Entnahme: (1) Landwirtschaft: Die geernteten Feldfrüchte werden in städtische Gebiete und zu Nutztieren transportiert, die ihrerseits in städtische Gebiete exportiert werden.CO2 wird in Viehzucht oder in städtischen Gebieten über die Atmung in die Atmosphäre abgegeben. (2) Forstwirtschaft: Der abgeholzte Kohlenstoff wird in städtische und industrielle Gebiete transportiert und dann durch Zersetzung in Deponien oder durch Verbrennung als Biokraftstoff emittiert. (3) Wasserkreislauf: Kohlenstoff wird aus den Böden in Gewässer, wie z. B. Seen, ausgewaschen. Der Kohlenstoff wird dann entweder deponiert, oder in die Atmosphäre freigesetzt oder in den Ozean transportiert. Pfeile zeigen Kohlenstoffflüsse (F) an - grau: Emissionen fossiler Brennstoffe, dunkelgrün: Ökosystem-Stoffwechsel, rot: Biomasseverbrennung, hellgrün: Forstwirtschaft, gelb: Landwirtschaft, blau:Wasserkreislauf. Halbtransparente Pfeile: Flüsse zwischen Erdoberfläche und Atmosphäre, volle Pfeile: Flüsse zwischen Landregionen.Gestrichelte Pfeile: Emissionen von reduziertem Kohlenstoffspezies in die Atmosphäre. (Bild und Legende von Redn.deutsch beschriftet, aus B.Byrne et al., 2023 [1] eingefügt; Lizenz cc-by.) |
Die Daten sind besonders nützlich, um Kohlendioxidschwankungen im Zusammenhang mit der Veränderung der Bodenbedeckung zu verfolgen. Allein die Emissionen aus der Entwaldung machen einen unverhältnismäßig großen Teil der gesamten Kohlenstoffbilanz im globalen Süden aus, der Regionen in Lateinamerika, Asien, Afrika und Ozeanien umfasst. In anderen Teilen der Welt deuten die Ergebnisse auf eine gewisse Verringerung der atmosphärischen Kohlenstoffkonzentration durch verbesserte Landbewirtschaftung und Wiederaufforstung hin.
Nach Meinung der Autoren sind Bottom-up-Methoden zur Abschätzung von Kohlendioxidemissionen und -Entnahmen durch Ökosysteme unabdingbar. Diese Methoden sind jedoch anfällig für Unsicherheiten, wenn Daten fehlen oder die Nettoauswirkungen bestimmter Aktivitäten, wie z. B. der Abholzung, nicht vollständig bekannt sind.
"Unsere Top-Down-Messungen liefern eine unabhängige Bestimmung dieser Emissionen und der Entnahmen. Wenn sie auch das detaillierte Prozessverständnis der traditionellen Bottom-up-Methoden nicht ersetzen, können wir doch beide Ansätze auf ihre Konsistenz hin überprüfen", so Philippe Ciais, einer der Studienautoren und Forschungsdirektor am Laboratoire des Sciences du Climat et de l'Environnement in Frankreich.
Dem Kohlenstoff auf der Spur bleiben
Die Studie bietet ein komplexes Bild der Reise des Kohlenstoffs durch Land, Ozean und Atmosphäre der Erde [1].
Zusätzlich zu den direkten Beeinflussungen durch den Menschen, die in den nationalen Verzeichnissen erfasst sind, können unbewirtschaftete Ökosysteme wie einige tropische und boreale Wälder - wo der Mensch nur einen minimalen Fußabdruck hinterlässt - Kohlenstoff aus der Atmosphäre binden und so die potenzielle globale Erwärmung verringern.
"Nationale Verzeichnisse sollen aufzeigen, wie sich Bewirtschaftungsmaßnahmen auf die Emissionen und den Abbau von CO2 auswirken", so Studienautor Noel Cressie, Professor an der University of Wollongong in Australien. "Der Atmosphäre ist es jedoch egal, ob CO2 durch Abholzung im Amazonasgebiet oder durch Waldbrände in der kanadischen Arktis ausgestoßen wird. Beide Prozesse erhöhen die CO2-Konzentration in der Atmosphäre und treiben den Klimawandel voran. Daher ist es von entscheidender Bedeutung, die Kohlenstoffbilanz unbewirtschafteter Ökosysteme zu überwachen und Veränderungen bei der Kohlenstoffaufnahme (= Photosynthese) zu erkennen".
Mit Blick auf die Zukunft sagen die Forscher, dass ihr Pilotprojekt weiter verfeinert werden kann, um zu verstehen, wie sich die Emissionen der einzelnen Länder verändern.
"Nachhaltige, qualitativ hochwertige Beobachtungen sind für diese Top-Down-Schätzungen von entscheidender Bedeutung", sagt der Hauptautor Brendan Byrne, Wissenschaftler am Jet Propulsion Laboratory der NASA in Südkalifornien. "Mit kontinuierlichen Beobachtungen von OCO-2 und Oberflächenstandorten können wir verfolgen, wie sich die Emissionen und Entnahmen im Zuge der Umsetzung des Pariser Abkommens verändern. Künftige internationale Missionen, die eine erweiterte Kartierung der CO2-Konzentrationen auf der ganzen Welt liefern, werden es uns ermöglichen, diese Top-Down-Schätzungen zu verfeinern und genauere Bestimmungen von Emissionen und Entfernungen in den einzelnen Ländern zu liefern."
[1] Brendan Byrne et al., National CO2 budgets (2015–2020) inferred from atmospheric CO2 observations in support of the global stocktake. Earth Syst. Sci. Data, 15, 963–1004, 2023. https://doi.org/10.5194/essd-15-963-2023
* Der vorliegende Artikel von Sally Younger (NASA's Jet Propulsion Laboratory) ist unter dem Titel "NASA Space Mission Takes Stock of Carbon Dioxide Emissions by Countries" https://www.jpl.nasa.gov/news/nasa-space-mission-takes-stock-of-carbon-dioxide-emissions-by-countries am 7. März 2023 auf der Web-Seite der NASA erschienen. Der unter einer cc-by-Lizenz stehende Artikel wurde von der Redaktion möglichst wortgetreu übersetzt und durch Abbildungen vom NASA’s Scientific Visualization Studio (Abbildung 1) und von der dem Artikel zugrunde liegenden Veröffentlichung von B.Byrne et al., 2023 [1] (Abbildung 2) ergänzt.
Weiterführende Links
Das NASA OCO-2 Projekt: https://ocov2.jpl.nasa.gov
Animation: National Carbon Dioxide Budgets Inferred from Atmospheric Observations: Fossil Fuel Emissions. Video 0:12 min. . https://www.youtube.com/watch?v=wr327cqtXfs
Klimawandel - Schwerpunktsthema im ScienceBlog. Bis jetzt behandeln mehr als 40 Artikel den Klimawandel - von Modellierungen über Folgen des Klimawandels bis hin zu Strategien seiner Eindämmung; chronologisch gelistet in Klima & Klimawandel
Die Zukunft der evolutionären Medizin
Die Zukunft der evolutionären MedizinDo 20.04.2023 — Redaktion
![]() Evolutionäre Medizin - d. h. die Anwendung von Erkenntnissen aus Evolution und Ökologie auf die Biomedizin - birgt ein enormes bislang ungenutztes Potenzial für Innovationen in der biomedizinischen Forschung, der klinischen Versorgung und der öffentlichen Gesundheit. Evolutionsprozesse sind die treibende Kraft hinter vielen Gefährdungen unserer Gesundheit; diese reichen von der Resistenz gegen Antibiotika und Tumortherapeutika über Pandemien bis hin zu "anthropozänbedingten" Krankheiten wie Adipositas, Typ 2 Diabetes, Allergien, etc. . Ein neuer in Frontiers in Science veröffentlichter Artikel zeigt, wie die Anwendung evolutionärer Prinzipien auf die Medizin neue Wege zur Vorbeugung und Behandlung von Krankheiten eröffnen kann.*
Evolutionäre Medizin - d. h. die Anwendung von Erkenntnissen aus Evolution und Ökologie auf die Biomedizin - birgt ein enormes bislang ungenutztes Potenzial für Innovationen in der biomedizinischen Forschung, der klinischen Versorgung und der öffentlichen Gesundheit. Evolutionsprozesse sind die treibende Kraft hinter vielen Gefährdungen unserer Gesundheit; diese reichen von der Resistenz gegen Antibiotika und Tumortherapeutika über Pandemien bis hin zu "anthropozänbedingten" Krankheiten wie Adipositas, Typ 2 Diabetes, Allergien, etc. . Ein neuer in Frontiers in Science veröffentlichter Artikel zeigt, wie die Anwendung evolutionärer Prinzipien auf die Medizin neue Wege zur Vorbeugung und Behandlung von Krankheiten eröffnen kann.*
Mit evolutionären Prinzipien die Biomedizin und das Gesundheitswesen verändern
Die evolutionäre Medizin verfügt über ein enormes - und noch nicht verwirklichtes - Potenzial zur Verbesserung von Gesundheit und Wohlbefinden der Menschen, indem sie neue biomedizinische Therapien und wirksame Maßnahmen im Bereich der öffentlichen Gesundheit ermöglicht. In einer neuen Veröffentlichung im Fachjournal Frontiers in Science [1] skizziert ein breit aufgestelltes internationales Team aus den US, dem UK und Neuseeland eine Forschungsagenda für die künftige evolutionäre Medizin, wobei der Fokus auf folgenden Anwendungen liegt:
- Tierarten zu finden, die von Natur aus resistent gegen menschliche Krankheiten sind, und diese Mechanismen in neue klinische Behandlungen umzusetzen
- Arzneimittelresistenzen bei Infektionskrankheiten und Krebs zu überwinden
- die Gesundheitspolitik darüber zu instruieren, wie Krankheiten wirksam bekämpft werden können, die aus der Diskrepanz zwischen den Ökologien, in denen wir uns entwickelt haben, und der heutigen Welt resultieren
- das Management von Pandemien zu verbessern
- menschlichen Widerstand gegen gesundheitsfördernde Verhaltensweisen zu überwinden.
| Abbildung 1. Evolutionäre Medizin basierend auf den Prinzipien der Evolution und der Ökologie. (Bild von Redn. leicht modifiziert. Quelle: B. Natterson-Horowitz et al.,2023, [1]; Lizenz cc-by) |
Was ist evolutionäre Medizin?
Die evolutionäre Medizin wendet die Erkenntnisse aus Ökologie und Evolution an, um biomedizinische Forschung, Maßnahmen des öffentlichen Gesundheitswesens und klinische Versorgung zu instruieren, zu steuern und zu verbessern. Abbildung 1.
Mit der Anwendung einer evolutionären Perspektive könnten viele kritische gesundheitliche Herausforderungen bewältigt werden. Dazu zählen die Krise der Antibiotikaresistenz, die Chemotherapieresistenz bei Krebserkrankungen, moderne Gesundheitsprobleme wie Fettleibigkeit, Diabetes und Herz-Kreislauf-Erkrankungen - und sogar die Resistenz von Menschen gegenüber Gesundheitsratschlägen. Dieser Ansatz wird auch für die Identifizierung neu auftretender Krankheitserreger und die Bewältigung künftiger Epidemien und Pandemien von entscheidender Bedeutung sein.
Wie kann die evolutionäre Vielfalt biomedizinische Therapien inspirieren?
Die traditionelle biomedizinische Forschung nutzt die Ähnlichkeiten zwischen Menschen und anderen Tieren, um Krankheitsmechanismen aufzudecken und Therapien zu entwickeln. Aber die Evolutionsmedizin lässt sich auch von unseren Unterschieden inspirieren. So erkranken Elefanten beispielsweise nur selten an Krebs, während Nacktmulle von Natur aus resistent gegen altersbedingte Krankheiten sind und daher ungewöhnlich lange und gesund leben. In der riesigen Vielfalt des Lebens auf der Erde gibt es wahrscheinlich noch unzählige andere Mechanismen zur Krankheitsresistenz. Wir müssen diese systematisch aufspüren, ihre physiologischen Grundlagen aufdecken und diese Erkenntnisse in neue klinische Behandlungen umsetzen. Abbildung 2.
| Abbildung 2. Die Identifizierung von Tierarten und ihrer besonderen Physiologie, welche die Anfälligkeit für Krankheiten einschränkt, kann die biomedizinische Innovation beschleunigen. Ein systematischer Ansatz ist erforderlich, um eine umfassende phylogenetische Karte der Anfälligkeit und Resistenz gegenüber Krankheiten zu erstellen.(Bild von Redn. leicht modifiziert. Quelle: B. Natterson-Horowitz et al.,2023, [1]; Lizenz cc-by) |
Wie können evolutionäre Prinzipien neue Krebsbehandlungen inspirieren?
Viele Krebstherapien zielen darauf ab, Tumore mit hochdosierten Krebsmedikamenten zu eliminieren. Wenn die Behandlung dann nicht mehr wirksam ist, wird sie durch eine andere und möglicherweise durch noch weitere Behandlungen ersetzt. Auch wenn dies erfolgreich sein kann, gibt es zwei entscheidende Nachteile: die Entwicklung von Arzneimittelresistenzen in Krebszellen und die Toxizität für normale Zellen. Die evolutionäre Medizin kann diese Hürden überwinden, indem sie Behandlungen entwickelt, welche die Entwicklung der Krebszellen berücksichtigen. Beispielsweise:
- Die Extinktionstherapie sieht eine hohe Dosis eines Medikaments vor, um den Tumor zu verkleinern, gefolgt von einem anderen Medikament, um die verbleibenden Krebszellen abzutöten, bevor sie eine Chemotherapieresistenz entwickeln.
- Die adaptive Therapie zielt darauf ab, die Tumorgröße stabil zu halten, während die Krebszellen für die medikamentöse Behandlung empfindlich bleiben. Dieser Ansatz ist vielversprechend für eine langfristige Kontrolle fortgeschrittener Krebserkrankungen.
Wie kann die Evolutionsmedizin dazu beitragen, die Krise der antimikrobiellen Resistenz zu bewältigen?
Die Resistenz von Bakterien und anderen infektiösen Mikroorganismen gegen antimikrobielle Mittel ist eine große globale Gesundheitsbedrohung. Zu den konventionellen Lösungen gehört die Entwicklung neuer antimikrobieller Mittel, was jedoch kostspielig ist und nur eine vorübergehende Lösung darstellt. Alternative Ansätze umfassen:
| Abbildung 3. Die Anti-Antibiotika-Strategie zur Verhinderung von Resistenzentwicklung und Weiterverbreitung bei kommensalen opportunistischen Krankheitserregern.(Bild von Redn. leicht modifiziert. Quelle: B. Natterson-Horowitz et al.,2023, [1]; Lizenz cc-by) |
- Medikamentenkombinationen, Zeitpläne und Dosierungen, die verhindern, dass Mikroben Resistenz entwickeln
- Eingriffe in den Evolutionsprozess selbst, z. B. durch Störung der Mechanismen, mit denen Bakterien ihre DNA austauschen und dadurch Resistenzen verbreiten (ein Prozess, der als horizontaler Gentransfer bezeichnet wird)
- orale "Anti-Antibiotika", die verhindern könnten, dass intravenös verabreichte Antikörper bei Darmbakterien Resistenzen auslösen (Abbildung 3)
- Einsatz von Viren, die Bakterien infizieren, sogenannte Phagen, um antibiotikaresistente Varianten abzutöten und/oder evolutionäre Kompromisse zu erzwingen, die die Empfänglichkeit für Antibiotika wiederherstellen.
Wie kann die evolutionäre Medizin in Maßnahmen des öffentlichen Gesundheitswesens einfließen?
Viele menschliche Krankheiten sind das Ergebnis eines Missverhältnisses zwischen den Ökosystemen, in denen wir uns entwickelt haben, und unserer modernen Umwelt. Abbildung 4. Ein Beispiel für diese Diskrepanz ist unsere Fähigkeit, Energie als Fett zu speichern. Dies war in der Vergangenheit ein Vorteil, als die Nahrung knapp war, führt aber in Gesellschaften, in denen es heute reichlich Nahrung gibt, zu einem Anstieg von Adipositas, Typ-2-Diabetes und Herz-Kreislauf-Erkrankungen. Weitere Probleme können entstehen, wenn unser Körper als Reaktion auf unser ökologisches und soziales Umfeld seine Prioritäten zwischen den drei wichtigsten evolutionären Zielen (Wachstum, Fortpflanzung und allgemeine Überlebensfähigkeit) verschiebt. Männliche Fruchtbarkeitsprobleme könnten ein Beispiel dafür sein.
| Abbildung 4. Gesundheitspolitische Massnahmen auf Basis der evolutionären Medizin. (Bild von Redn. leicht modifiziert. Quelle: B. Natterson-Horowitz et al.,2023, [1]; Lizenz cc-by) |
Eine evolutionäre Perspektive kann diese Probleme angehen, indem die ökologischen Bedingungen mit den Bedürfnissen der menschlichen Biologie in Einklang gebracht werden. Im Falle einer evolutionären Fehlanpassung könnte dies bedeuten, dass der Schwerpunkt von der individuellen Verantwortung, die sich auf Ernährung und Bewegung konzentriert, auf Maßnahmen des öffentlichen Gesundheitswesens wie Lebensmittelsteuern und Beschränkungen in der Vermarktung von Lebensmitteln an Kinder verlagert wird.
Welche Rolle spielt die evolutionäre Medizin bei der Bekämpfung von COVID-19 und künftigen Pandemien?
Evolutionäre Prinzipien sind für die Vorhersage von Krankheitsausbrüchen, die Beherrschung der Infektionen und die Modellierung von Zukunftsszenarien unerlässlich. Die COVID-19-Pandemie ist ein perfektes Beispiel dafür. Modelle, die auf Darwins Erkenntnissen über Anpassung und natürliche Selektion beruhen,haben die Eigenschaften der erfolgreichen SARS-CoV-2-Varianten korrekt voraus gesagt. Diese Erkenntnisse ermöglichten auch die schnelle Identifizierung und das Aufspüren neuer Varianten sowie der Orte, an denen das Virus übertragen worden war.
Wie kann die evolutionäre Medizin gesunde Verhaltensweisen fördern?
Die Evolutionsmedizin kann helfen zu erklären, warum wir ungesunde Entscheidungen treffen, wie z.B. übermäßiges Essen, Rauchen, einen sitzenden Lebensstil und die Ablehnung von Impfungen. So können beispielsweise Begebenheiten im Leben eines Menschen den Ausschlag für solche Verhaltensweisen geben, während Entscheidungen, die oft als "problematisch" bezeichnet werden, im Rahmen einer eigenen, oft unbewussten Kosten-Nutzen-Rechnung des Einzelnen durchaus Sinn machen können.
Betrachtet man das Verhalten der Menschen durch diese evolutionäre Brille, so ließen sich wirksamere Maßnahmen im Bereich der öffentlichen Gesundheit entwickeln. Politische Maßnahmen, die sich auf die Verbesserung der Lebensperspektiven der Menschen konzentrieren - indem sie allen Menschen Zugang zu Bildung und Arbeitsplätzen verschaffen und gleichzeitig Ernährungsunsicherheit, Diskriminierung und Kriminalität beseitigen - würden insbesondere dazu beitragen, gesunde Verhaltensweisen in der gesamten Gesellschaft zu fördern.
[1] Natterson-Horowitz B et al., The future of evolutionary medicine: sparking innovation in biomedicine and public health. Front Sci (2023) 1:997136. doi: 10.3389/fsci.2023.997136. (Lizenz: cc-by)
*Zur Veröffentlichung von Natterson-Horowitz B et al., [1] gibt es auch eine Zusammenfassung für Nicht-Wissenschafter (Lay summary 1doi: 10.3389/fsci.2023.997136). Diese wurde von der Redn. möglichst wortgetreu übersetzt und durch 2 Abbildungen aus dem Originalartikel ergänzt.
The future of evolutionary medicine. Video 0:40 min. https://www.youtube.com/watch?v=iuhWfoHaINs&t=4s
Mitochondrien - mobile Energielieferanten in Neuronen
Mitochondrien - mobile Energielieferanten in NeuronenFr, 14.04.2023 — Susanne Donner
Archaisch, mächtig, flexibel: Mitochondrien sind weit mehr als Kraftwerke. Sie treiben die Evolution, bestimmen, was das Gehirn leistet und sind mitverantwortlich, wenn wir bei Sauerstoffmangel in Ohnmacht fallen. Die Chemikerin und Wissenschaftsjournalistin Susanne Donner gibt eine Überblick über die Rolle der Mitochondrien in unserem enorm energieverbrauchenden Nervensystem, wie Neuronen über die ganze Länge ihrer Dendriten und Axone die Energieversorgung sicherstellen, und welche Folgen eine Unterversorgung haben kann.*
Die folgenreiche Vereinnahmung hat sich vor mindestens einer Milliarde Jahren ereignet. Lange bevor Tiere und Menschen die Erde bevölkerten. Im Wasser, das den Planeten bedeckte, trafen ein Bakterium und ein weiterer Einzeller aufeinander. Der Einzeller verleibte sich das Bakterium ein – eine Fusion zweier Lebensformen, die letztlich für beide von Nutzen war: So entstanden die Organelle, die sich heute in jeder Zelle von Tieren, Pflanzen und Menschen befinden.
Relikt der geschichtsträchtigen Verschmelzung, die als Endosymbiontentheorie in die Lehrbücher einging, sind beispielsweise die Mitochondrien, gern als „Kraftwerke der Zelle“ bezeichnet. Ihre Erbinformation ist bis heute „nackt“ und ringförmig als sogenanntes Plasmid angeordnet – so wie man es von Bakterien kennt. Dagegen ist die DNA des Zellkerns auf Histone aufgerollt, spezielle Proteine, die die DNA verpacken. Zudem sind die Mitochondrien von einer Doppelmembran umhüllt, die entfernt an die Zellwand von Bakterien erinnert. Abbildung 1. Auch, dass die mitochondriale DNA viel schneller Mutationen anhäuft – wohl weil die Reparatursysteme weniger effizient arbeiten – könnte auf Bakterien zurückgehen, die aufgrund ihrer kurzen Lebensdauer auf derartige Mechanismen weniger angewiesen sind. Die Kern-DNA ist dagegen robuster gegenüber dem Zahn der Zeit.
| Abbildung 1. Mitochondrien: zugereiste Kraftwerke in eukaryotischen Zellen (Bild von Redn. eingefügt aus: Wikipedia; gemeinfrei) |
Die Macht der Mitochondriengene
Gleichwohl wurde der Beitrag der Mitochondrien zur Erbmasse lange vernachlässigt, weil ihre Gene nur einen Anteil von 0,1 Prozent beisteuern. Sie zählen gar nur 37 Gene, gegenüber rund 25.500 auf der Doppelhelix im Zellkern. Die Mitochondrien stammen in der Regel von der Mutter. Die Kraftwerke des Vaters werden in der befruchteten Eizelle eliminiert. Es gibt allerdings Ausnahmen „Toyota-Prinzip: Nichts ist unmöglich“, kommentiert die Neurowissenschaftlerin Petra Wahle von der Universität Bochum. „Dann gibt der Vater seine Mitochondrien weiter. Das ist aber selten.“
So oder so – lange dachten Forscher, dass die Vererbungswege für die Mitochondrien-DNA und die Kern-DNA unabhängig nebeneinander existieren. Doch neuere Studien unterstreichen, wie eng Zellkern und Mitochondrien sich aufeinander abstimmen und miteinander austauschen.
Der Genetikspezialist Wei Wei von der Universität Cambridge und seine Kollegen untersuchten dafür das Erbgut von 1.526 Müttern und ihren Kindern. Und zwar sequenzierten sie sowohl das Kern- als auch das mitochondriale Genom. Dabei fiel ihnen auf, dass Mutationen im Mitochondriengenom nicht beliebig an den Nachwuchs weitergegeben werden. Vielmehr setzten sich eher solche Varianten durch, die bereits in der Vergangenheit aufgetreten waren und die mit der Kern-DNA harmonierten. Dies ergab ein Abgleich mit einer Gendatenbank, in der die Geninformationen zu 40.325 weiteren Personen hinterlegt waren. Wei schließt daraus, dass die Kern-DNA kontrolliert, welche Mitochondrien-Erbinformationen sich durchsetzen. Beide Vererbungswege sind keineswegs isoliert voneinander.
„Mitochondrien ko-evolvieren mit der Kern-DNA“, sagt Wahle. Das habe damit zu tun, dass sie längst nicht nur Energielieferanten sind. Sie haben vielfältige basale Funktionen. So steuern sie beispielsweise die Produktion wichtiger Signalstoffe und Zellbausteine. Nur mit ihrer Hilfe kann die Zelle komplexe Proteine und andere Stoffe erzeugen. Diese Baustoffe brauchen wiederum die Mitochondrien dauernd, um sich permanent rundzuerneuern. Es besteht eine wechselseitige Abhängigkeit. Diese bedingt, dass die Erbmasse von Mitochondrien und Kern fein aufeinander abgestimmt sein muss.
Wenn die Mitochondriengene nicht passen, droht das Aussterben
Das geht so weit, dass beliebig mutierte Mitochondrien die Fitness des Organismus kompromittieren würden. Wenn Forschende etwa die Mitochondrien einer anderen Art in die Keimbahn einschleusen, sind die Nachkommen dieser Bastarde zwar lebensfähig, aber weniger fruchtbar, legt Wahle dar. „Sie entwickeln sich schlechter und sterben unter dem Strich wieder aus.“
Mitochondriengene weisen aber, wie erwähnt, enorm hohe Mutationsraten auf. Auf diese Weise helfen sie Lebewesen dabei, sich zu Lebzeiten an sich rasch verändernde Umweltbedingungen anzupassen. Sie modulieren beispielsweise die Menge bereitgestellter Energie.
Über Mitochondrienmutationen entstehen auch in kurzer Zeit neue Arten, wie der israelische Evolutionsforscher Dan Mishmar herausarbeitete: etwa, wenn sich die Lebensräume zweier Populationen trennen. Dann verändert sich deren mitochondriale DNA derart rasch, dass beide schon nach kurzer Zeit nicht mehr kompatibel miteinander sind. Sie können zusammen keine ausreichend lebensfähigen Nachkommen mehr zeugen.
Mehr als kleine Punkte im Neuron
Neben der Fruchtbarkeit sind es kognitive Fähigkeiten, die als Erstes leiden, wenn die Mitochondrien schwächeln. Nach derzeitigem Wissensstand stehen alle neurodegenerativen Krankheiten mit maroden Zellkraftwerken in Verbindung. Die Nervenzellen im Gehirn, aber auch die Zellen von Muskeln und Auge haben einen besonders hohen Energiebedarf.
Das liegt an zwei Besonderheiten. Damit kognitive Prozesse, das Denken und Handeln, ablaufen können, muss der Ionenhaushalt im Gehirn in einem Gleichgewicht sein. Vor allem der Calciumspiegel darf weder in den Zellen noch außerhalb zu sehr abfallen. Das entscheidet mitunter über Leben und Tod einer Zelle. Und, wenn Nervenzellen feuern, müssen sie Aktionspotentiale weiterleiten. Auch das kostet viel Energie.
„In Lehrbüchern sind Mitochondrien immer so diskret hingezeichnet. Aber wir haben pro Neuron mehrere hundert von diesen Mitochondrien“, sagt der Neurowissenschaftler Oliver Kann von der Medizinischen Fakultät der Universität Heidelberg. Das sind viel mehr als beispielsweise in weniger energiehungrigen Geweben wie die Haut.
Mit Blick auf die Mitochondrien sei Neuron allerdings nicht gleich Neuron, wie Kann betont. In seinen Forschungen ergründet er, wie sich verschiedene Hirnzelltypen in ihren Mitochondrien unterscheiden. Manches ist augenfällig, etwa wenn seine Mitarbeitenden die Mitochondrien in Hirnschnitten von Ratte oder Maus histologisch anfärben. In den Dendriten, den verästelten Zellfortsätzen, die die elektrische Erregung von Nachbarzellen aufnehmen, sehen Mitochondrien langgezogen aus. Wenn man aber entlang des Zellfortsatzes näher an die Zelle heranrückt, werden die Mitochondrien kompakt und punktförmig. Und auch in den Prinzipalneuronen selbst, zu denen etwa die Pyramidenzelle gehört, haben sie diese aus Lehrbüchern vertraute Form.
Mitochondrien sind mobil und teilbar wie ein Güterzugsystem
| Abbildung 2. Die Form der Mitochondrien hängt vom Kompartiment des Neurons ab. Im Dendriten sind die Mitochondrien langgezogen (0,5 -8,9 µm) und füllen den Großteil des dendritischen Raums aus (b) unten). Im Axon sind die Mitochondrien vergleichsweise kurz (0,3 - 1,1 µm) und besetzen nur rund 5 % der Axonlänge. a) Pyramidenzelle aus dem Kortex, Mitochondrien mittels eines Matrix-gerichteten fluoreszierenden Proteins (mt-DsRED - grün) sichtbar gemacht. b)Vergrößerter Ausschnitt aus a) (braune Box): Teil eines Dendriten, mt-DsRED : mitochondrialer Marker, GFP: Cytosol markiert mit fluoreszierendem Protein. c) Vergrößerter Ausschnitt aus a) (grüne Box): Teil eines Axons, Markierung wie in b). (Bild von Redn. eingefügt, Quelle: Lewis, T.L. et al.,. Nat Commun 9, 5008 (2018) https://doi.org/10.1038/s41467-018-07416-2 Lizenz: cc-by 4.0) |
Besonders viele Mitochondrien sitzen in der synaptischen Endigung, also an jenem Ort, an dem Information von einer Nervenzelle zur nächsten übertragen wird. Dort ist besonders viel Energie nötig, um das Signal über den synaptischen Spalt weiterzuleiten. Dafür schließen sich die Mitochondrien teilweise sogar lokal zusammen. Sie bilden hochdynamische Netzwerke, erklärt Kahn, ähnlich einem Güterverkehrssystem. Mitochondrien können sich tatsächlich auch fortbewegen. Sie wandern entlang des Axons und nutzen dafür Mikrotubuli, röhrenförmige Proteine, und molekulare Motoren.
Qua ihrer Mobilität können sich Mitochondrien sammeln. Noch dazu sind sie – wieder in Analogie zum Güterzugsystem in der Lage - miteinander zu verschmelzen, sodass aus mehreren Mitochondrien ein Mitochondrium wird, oder sich zu teilen. (Abbildung 2) Diese Eigenart wird als „fusion“ für Verschmelzung und „fission“ für Spaltung bezeichnet. Wie wichtig sie ist, zeigt auch die Entdeckung, dass eine Stammzelle sich genau dann zum Neuron entwickelt, wenn sich die Mitochondrien in ihr massiv teilen und damit in ihrer Zahl erhöhen.
Wenn Mitochondrien nicht abliefern, schwächeln wir beim Lernen und Autofahren
Besonders viele Mitochondrien benötigen aber auch bestimmte Nervenzellen, Interneurone genannt, die die Netzwerkaktivität im Gehirn synchronisieren. Sie sind quasi die Taktgeber des Neuronenfeuers im Gehirn. Sie sorgen dafür, dass beim Ableiten der Hirnströme über die Kopfhaut überhaupt rhythmische Signale von den alpha- bis zu den theta-Wellen auf dem Bildschirm zu sehen sind. Die Interneurone ermöglichen höhere kognitive Leistungen wie die Verarbeitung komplexer visueller Reize und die selektive Wahrnehmung, auch das Bewusstsein. Dafür müssen sie allerdings in schnellem Takt stark feuern.
Kanns Team stellte unter Beweis, dass diese Energiefresser im Gehirn als erstes an Funktion einbüßen, wenn es zu mildem Stress auf den Stoffwechsel kommt. Der kann darin bestehen, dass weniger Glucose oder Sauerstoff ins Gehirn kommen oder zu viele freie Radikale anfluten. In weiteren Experimenten an isolierten Hirnschnitten von Ratte und Maus erkannten die Forschenden schließlich auch, dass auch ein gestörter Calcium-Ionen-Haushalt die rhythmische Netzwerkaktivität, den Job der Interneurone, unterminiert. Wahrscheinlich, weil die Mitochondrien nicht richtig arbeiten können, wenn der Calciumeinstrom in die Zelle gestört ist.
Für den Neurowissenschaftler können all diese Befunde ein bemerkenswertes Verhalten des menschlichen Gehirns erklären: An Piloten testete man nach dem Zweiten Weltkrieg, was geschieht, wenn kurzzeitig zu wenig Blut ins Gehirn gelangt. Wie anekdotisch überliefert sei, berichtet Kann, legte man ihnen dafür eine enge Manschette um den Hals. Nach vier bis sechs Sekunden waren sie schon bewusstlos. Doch ihre Nervenzellen feuerten noch weiter. Erst nach einigen Minuten beginnen die Neuronen unterzugehen. Kann sagt: „Das Gehirn hat einfach nicht ausreichend Energiespeicher, um die Interneurone zu versorgen und damit die höheren kognitiven Fähigkeiten aufrechtzuerhalten.“
Frauen bekamen das im 19.Jahrhundert dank der damals vorherrschenden Mode am eigenen Leib zu spüren. Zu eng geschnürte Korsette drückte ihre Atemwegsorgane ab, sodass ihr Gehirn zu wenig Sauerstoff bekam. Mangels Mitochondrien-Power fielen die Frauen dann schon mal zu Boden. Nach der Ohnmacht aber war es, als sei nichts gewesen.
Zusammenfassung
Zum Weiterlesen
- Wei, W et al.: Germline selection shapes human mitochondrial DNA diversity. Science. 2019. 364(6442):eaau6520. doi: 10.1126/science.aau6520
- Bas-Orth, C et al. : The mitochondrial calcium uniporter is crucial for the generation of fast cortical network rhythms. J Cereb Blood Flow Metab. 2020, Nov;40(11):2225–2239. doi: 10.1177/0271678X19887777
- Kann, O. et al.: The interneuron energy hypothesis: Implications for brain disease. Neurobiol Dis. 2016, Jun;90:75¬–85. doi: 10.1016/j.nbd.2015.08.005
- Elzoheiry, S et al.: Mild metabolic stress is sufficient to disturb the formation of pyramidal cell ensembles during gamma osciallations. J Cereb Blood Flow Metab. 2020 Dec;40(12) :2401–2415. doi: 10.1177/0271678X19892657
- Iwata, R et al.: Mitochondrial dynamics in postmitotic cells regulate neurogenesis. Science. 2020 Aug 14;369(6505):858–862. doi: 10.1126/science.aba9760
- Rangaraju, V et al.: Pleiotropic Mitochondria: The Influence of Mitochondria on Neuronal Development and Disease. J Neurosci. 2019 Oct 16;39(42):8200–8208. . doi: 10.1177/0271678X19892657
*Der Artikel stammt von der Webseite www.dasGehirn.info, einer exzellenten Plattform mit dem Ziel "das Gehirn, seine Funktionen und seine Bedeutung für unser Fühlen, Denken und Handeln darzustellen – umfassend, verständlich, attraktiv und anschaulich in Wort, Bild und Ton." (Es ist ein Projekt der Gemeinnützigen Hertie-Stiftung und der Neurowissenschaftlichen Gesellschaft e.V. in Zusammenarbeit mit dem Zentrum für Kunst und Medientechnologie Karlsruhe). Der vorliegende Artikel ist am 15.3.2023 unter dem Titel: " Zugereiste Kraftwerke" https://www.dasgehirn.info/grundlagen/energie/zugereiste-kraftwerke erschienen. Der unter einer cc-by-nc-sa Lizenz stehende Artikel wurde unverändert in den Blog gestellt.
Verwandte Themen im ScienceBlog
- Nora Schulz, 23.03.2023: Neuronen sind Energiefresser
- Christina Beck, 05.04.2018: Endosymbiose - Wie und wann Eukaryonten entstanden
- Christina Beck, 29.3.2018: Ursprung des Lebens - Wie Einzeller kooperieren lernten
- Gottfried Schatz, 1.11.2012: Grenzen des Ichs - Warum Bakterien wichtige Teile meines Körpers sind
- Gottfried Schatz, 8.11.2013: Die Fremden in mir — Was die Kraftwerke meiner Zellen erzählen
Candida auris: ein neuer Pilz breitet sich aus
Candida auris: ein neuer Pilz breitet sich ausFr, 07.04.2023 — Ricki Lewis
Vor wenigen Tagen hat das US-Center of Disease Control (CDC) vor der zunehmenden Gefahr von gegen Antimykotika resistenten Pilzen gewarnt, die sich derzeit in Gesundheitseinrichtungen ausbreiten. In erster Linie geht es hier um den neu aufgetretenen Stamm Candida auris (C. auris), den ersten Pilz, von dem bereits multiresistente Formen gegen alle 3 Klassen von Antimykotika existieren. Infektionen mit diesem Pilz führen zu schwerer verlaufenden Erkrankungen als mit dem von alters her bekannten Verwandten Candida albicans, der für häufige Infektionen der Vagina und des Rachens verantwortlich ist. Die Genetikerin Ricki Lewis gibt einen Überblick über die gegenwärtige Situation.*
| Candida auris, ein multiresistenter Hefepilz (Quelle: Lundquist Institute) |
Candida-Hefepilze sind ganz normale Bewohner unserer Haut und anderer oberflächlicher Organe; sie werden aber gefährlich, wenn sie in den Blutkreislauf gelangen oder Organe wie das Herz oder die Nieren erreichen. "Das, wodurch Candida auris von anderen Candida-Arten verschieden und besonders beängstigend erscheint, ist dass der Keim bis zu zwei Wochen auf der Haut und auf Oberflächen im Gesundheitseinrichtungen überleben kann, und dadurch in diesen und in Pflegeheimen die Ausbreitung von Mensch zu Mensch ermöglicht. Dieser Pilz wird in der Regel nicht durch die üblichen klinisch angewandten Antimykotika abgetötet; dies erschwert die Behandlung der Infektion und kann oft zu einem tödlichen Ausgang führen. Erschwerend kommt dazu, dass der Keim mit Standard-Labormethoden nur schwer zu identifizieren ist." So fasst Mahmoud Ghannoum, Direktor des Zentrums für medizinische Mykologie am University Hospitals Cleveland Medical Center, die Situation zusammen.
Das US-Center for Disease Control (CDC) und andere Organisationen des öffentlichen Gesundheitswesens setzen die Ganzgenom-Sequenzierung ein, um die Verbreitung dieses Pilzes weltweit zu verfolgen. Die Behörde hat mit FungiNet im Jahr 2021 ein "Netzwerk für die molekulare Überwachung und genomische Epidemiologie für Pilzkrankheiten" gestartet, wobei der anfängliche Schwerpunkt auf C. auris liegt. Mit der Online-Ressource werden "landesweite Laborkapazitäten zur raschen Erkennung, Vorbeugung und Reaktion auf Arzneimittelresistenzen" gegen diese Infektion unterstützt.
Der Vergleich ganzer Genomsequenzen kann Aufschluss darüber geben, woher der Erreger kommt und wohin er wahrscheinlich geht (als COVID auftrat war die Überwachung in den USA im Vergleich zu anderen Ländern notorisch verspätet). Anhand der Sequenzen lässt sich die Evolution auf der Grundlage einer einzigen Annahme zurückverfolgen: Je ähnlicher die Nukleinsäuresequenzen zweier Arten, Stämme oder sogar Individuen sind, desto jünger ist ihr gemeinsamer Vorfahre. Aus diesen Daten lassen sich also Stammbäume ableiten - manchmal gibt es mehr als einen, um die Hinweise in der Sprache der Genetik zu erklären.
Dem neuen Erreger auf der Spur
Die erste klinische Identifizierung von Candida auris erfolgte 2009 in Japan, aufbewahrte Kulturen zeigen aber, dass der Erreger mindestens bis 1996 in Südkorea zurückdatiert. Das CDC bezeichnet Candida auris als "neu auftretendes Pathogen", weil es inzwischen in mehr als 30 Ländern aufgetaucht ist. Die Behörde verfolgt seit 2013 den Erreger, dessen Ausbreitung hat sich 2015 beschleunigt.
Ein 2018 in der Fachzeitschrift Lancet Infectious Diseases veröffentlichter Bericht des US-Candida auris Investigation Teams des CDC hat Gesamtgenom-Sequenzen des Hefepilzes von Patienten aus zehn US-Bundesstaaten sowie aus Indien, Kolumbien, Japan, Pakistan, Südafrika, Südkorea und Venezuela verglichen.
Die Studie hat sich dabei auf einzelne Basen-Positionen im Genom konzentriert, die von Land zu Land, von Patient zu Patient und sogar in ein und derselben Person variieren können (Single Nucleotide Polymorphisms, kurz SNPs). Das Team hat auch Reiseverhalten und Kontakte berücksichtigt, welche die Ausbreitung begünstigt haben könnten. Die US-Fälle "waren genetisch mit denen aus vier globalen Regionen verwandt, was darauf hindeutet, dass C. auris mehrmals in die USA eingeschleppt wurde. Die genetische Vielfalt der Isolate von denselben Patienten, Gesundheitseinrichtungen und Bundesstaaten deutet darauf hin, dass es eine lokale und kontinuierliche Übertragung gibt", so die Schlussfolgerung des Berichts.
Jetzt ist der Pilz also da.
Im Jahr 2022 meldete das CDC 2.377 Fälle von Patienten und weitere 5.754 bei deren Kontaktpersonen, vor allem in Kalifornien, Nevada, Texas, New York, Florida und Illinois. Dies könnte jedoch eine zu niedrige Zahl sein - einige Ärzte dürften die Fälle noch nicht melden (oder vielleicht nicht erkennen).
Wer ist gefährdet?
Wie viele andere Erreger ist C. auris besonders für Patienten in Krankenhäusern gefährlich; der Keim gelant dort in den Blutkreislauf und breitet sich in der betroffenen Person und auch auf andere Menschen aus. Er infiziert auch Ohren (daher das "auris") und Wunden und möglicherweise Lunge und Blase - zumindest wurden die Hefepilze aus Sputum und Urin isoliert.
Am stärksten gefährdet sind Patienten mit Kanülen, die in Körperteilen stecken (z.B. Venenkatheter, Harnwegskatheter, Beatmungstuben; Anmerkung Redn.), oder Patienten, die übermäßig bestimmte Breitband-Antimykotika oder Antibiotika verwendet haben. Die Sterblichkeitsrate kann 60 % erreichen - allerdings sind viele Patienten bereits schwer krank, bevor sie sich dann im Spital infizieren.
Candida auris verbreitet sich über die Luft und durch Kontakt mit kontaminierten, pilzhaltigen Oberflächen. Und Menschen jeden Alters können infiziert werden.
Für die Identifizierung von C. auris sind spezielle Labortests und Verfahren zur Kultivierung der Hefe erforderlich; der Erreger kann leicht mit anderen Candida-Arten verwechselt werden. Außerdem ist er gegen viele herkömmliche Antipilzmittel resistent (Resistenzen wurden gegen alle 3 Klassen von Antimykotika - Azole, Polyene und Echinocandine - festgestellt; Anmerkung Redn.) Die wirksamsten Medikamente sind noch die Echinocandine, aber es können auch Kombinationen von Medikamenten erforderlich sein, um die Infektion in den Griff zu bekommen.
Die Suche nach neuen Antimykotika
ist im Gange. Beispielsweise hat die Case Western Reserve University gerade eine Förderung in der Höhe von 3 Millionen Dollar von den National Institutes of Health (NIH) erhalten, um neue Therapeutika zu entwickeln.
Meiner Meinung nach steckt aber der Großteil der Forschung noch im präklinischen Stadium (auf der Stufe von Tiermodellen und Synthese von Substanzen), denn auf der Plattform Clinicaltrials.gov, die weltweit klinische Studien listet, sind nur drei solcher Studien an dem Hefepilz gelistet; diese finden in Indien, Pakistan und Südafrika statt (in zwei der Studien werden Substanzen mit neuen Wirkmechanismen untersucht; Sponsoren sind Scynexis, Inc und Pfizer. Die dritte Studie in Pakistan befasst sich mit epidemiologischen Daten. Anmerkung Redn.). Ein Impfstoff wird am Lundquist Institute der UCLA entwickelt - bislang funktioniert er bei Mäusen und kann mit Antimykotika kombiniert werden.
Einstweilen werden die Genomvergleiche fortgesetzt, um Informationen zu sammeln, den Erreger zu verfolgen und wenn möglich auch vorherzusagen, wo sich die Hefe ausbreitet, um Ausbrüche verhindern zu können. Durch die Berücksichtigung von Genomdaten zusammen mit anderen Informationen können die Forscher herausfinden, wie die Menschen mit dem Pilz in Kontakt gekommen sind, wohin sich der Pilz wahrscheinlich geografisch bewegt, und sogar die Einschleppung des Erregers in neue Gebiete aufdecken, bevor Ausbrüche auftreten oder entdeckt werden.
Der Artikel ist erstmals am 30. März 2023 in PLOS Blogs - DNA Science Blog unter dem Titel " The New Fungus Among Us, Candida auris" https://dnascience.plos.org/2023/03/30/the-new-fungus-among-us-candida-auris/ erschienen und steht unter einer cc-by Lizenz . Die Autorin hat sich freundlicherweise mit der Übersetzung ihrer Artikel durch ScienceBlog.at einverstanden erklärt. Der leicht gekürzte Artikel folgt so genau wie möglich der englischen Fassung.
Warnungen der Behörden
- US-Centers for Disease Control and Prevention (CDC): Increasing Threat of Spread of Antimicrobial-resistant Fungus in Healthcare Facilities (Press release, 20. March 2023) https://www.cdc.gov/media/releases/2023/p0320-cauris.html
- European Centre for Disease Prevention and Control (ECDC): Emerging spread of new fungal species poses risk for healthcare settings in the EU/EEA (23. April 2018). https://www.ecdc.europa.eu/en/news-events/emerging-spread-new-fungal-species-poses-risk-healthcare-settings-eueea
Neuronen sind Energiefresser
Neuronen sind EnergiefresserDo, 23.03.2023 — Nora Schultz
Das Gehirn ist der größte Energiefresser des Körpers und gleichzeitig ungemein komplex. Das Gehirn des Menschen besteht aus rund 86 Milliarden Neuronen und obwohl viel Energie in die Kommunikation über die elektrischen Impulse auf der Membran läuft, verbrauchen auch die Prozesse im Inneren der Zelle eine gehörige Menge davon. Geliefert wird sie von bestimmten Zellorganellen, den Mitochondrien – sie bilden das Hauptkraftwerk sämtlicher Körperzellen.*
„It’s the economy, stupid.“ Der Spruch, der Bill Clintons Wahlkampagne 1992 mit zum Sieg verhalf, taugt auch als Motto für das Gehirn. Denn im Kopf kommt es ebenfalls entscheidend auf die Wirtschaft an – genauer gesagt: auf die Energiewirtschaft. Ohne eine stets ausbalancierte Energiezufuhr und -verteilung gelingen keine Geistesblitze und drohen schlimmstenfalls Zelltod und bleibende Schäden im Gehirn.
| Abbildung 1. Die Energieversorgung der Neuronen. Das einzelne Neuron enthält verschiedene Bereiche mit unterschiedlichen Aufgaben, und sie verzweigt sich über weite Distanzen. Dadurch wird der Transport von Substanzen innerhalb der Zelle zur Herausforderung, auch der von Mitochondrien, den Kraftwerken der Zelle. (Grafik MW |
Knapp anderthalb Kilo gallertartige Masse im Kopf verschlingen bis zu 20 Prozent des gesamten menschlichen Energieumsatzes – allein dies zeigt, wie anspruchsvoll unser Oberstübchen ist. Unter Ruhebedingungen geht der größte Stromverbrauch– wenig überraschend – auf das Konto der Nervenzellen. Viel Rechnerei ist teuer. Das gilt nicht nur für das Schürfen von Kryptowährung, sondern auch für den Datenaustausch im Gehirn.
Neuronen verrechnen Informationen und leiten sie weiter, indem sie ständig Neurotransmitter und Ionen durch ihre Zellmembranen transportieren. Dafür müssen sie fortwährend Transportkanäle öffnen, und brauchen viel Energie, um molekulare Pumpen zu bedienen und die Kanäle wieder zu schließen.
Zwar verbrauchen auch andere Zelltypen im Gehirn viel Energie, wie etwa die ebenso zahlreichen Gliazellen. Die Nervenzellen aber ragen als Rechenkünstler hervor. Abbildung 1.
Schon ihre Gestalt regt die Fantasie an. Neurone sind halb Baumwesen, halb Alien: Der „Kopf“ eines Neurons – der Zellkörper, der den Zellkern enthält – vernetzt sich über die vielen Antennen seiner fein verästelten Krone mit unzähligen anderen Neuronen. Seine Krone ist beindruckend ausladend – ihre Arme, die Dendriten erstrecken sich beispielsweise in den besonders großen Pyramidenzellen der Großhirnrinde über mehrere hundert Mikrometer, während der Zellkörper nur 20 Mikrometer Durchmesser hat. Abbildung 2. Verglichen mit dem „Stamm“ der Zelle – dem Axon – wirken die Dendriten allerdings winzig. Gemessen an den Maßstäben anderer Zellen schlängelt sich das Axon über gigantische Distanzen von bis zu einem Meter durchs Nervensystem und dockt am Ziel über das ebenfalls stark verzweigte Wurzelwerk der Axonterminale erneut an viele Partnerzellen an.
| Abbildung 2. Schematische Darstellung einer Nervenzelle und einer Helferzelle (Gliazelle). Neuronen sind auf Helferzellen angewiesen, die Blutgefäße anzapfen und vorverdauten Treibstoff an Neurone weitergeben (Bild von Redn. eingefügt, leicht modifiziert: Blausen.com staff (2014). "Medical gallery of Blausen Medical 2014". WikiJournal of Medicine 1 (2). DOI:10.15347/wjm/2014.010. Lizenz: cc-by.) |
Der Bedarf
Eine durchschnittliche Nervenzelle kommt so auf zehntausende Kontaktstellen (Synapsen) mit anderen Neuronen. Jede dieser Synapsen besteht aus einem präsynaptischen Terminal – einem Knöpfchen am Ende eines Axons – das Signale weitergibt, und einem postsynaptischen Terminal in Gestalt eines „Knubbels“, das sie empfängt. Abbildung 3, Ausschnitt. Signale überqueren den Spalt zwischen den Terminalen immer in Form kleiner Moleküle, den Neurotransmittern. Am postsynaptischen Terminal werden sie von spezialisierten Empfängereiweißen (Rezeptoren) erkannt. Diese lösen dann eine Reaktion aus, die das Neuron mit vielen Signalen, die es über seine anderen Synapsen empfängt, verrechnet. Je nach Rechenergebnis feuert die Zelle dann ein Aktionspotential, bei dem sich ein elektrisches Signal in einer Kettenreaktion entlang des Axons bis in die präsynaptischen Endungen fortpflanzt und über die dort liegenden Synapsen weitere Neurone erreicht. Aktionspotentiale sind immer gleich stark, unterscheiden sich aber in ihrer Häufigkeit – von Zelle zu Zelle und Situation zu Situation. Abbildung 3.
| Abbildung 3. Signalübertragung zwischen zwei Neuronen. Detail: Übertragung an der Synapse. (Bild von Redn. eingefügt: Christy Krames, MA, CMI — https://web.archive.org/web/20070713113018/http://www.nia.nih.gov/Alzheimers/Publications. Lizenz: gemeinfrei) |
Die Weitläufigkeit und Gliederung der Neuronen in verschiedene Bereiche (Kompartimente) lässt erahnen, welche Herausforderungen die Energieversorgung darstellt. Es gilt, jede Menge Prozesse und Materialien im Gleichgewicht – auch Homöostase genannt – zu halten. Je nachdem, was in den einzelnen Kompartimenten eines Neurons geschieht – in den Dendriten, dem Zellkörper, dem Axon oder den Synapsen – müssen Signale und Stoffe in andere Kompartimente geschickt, nach außen abgeben oder aus der Umgebung aufgenommen werden. Sowohl die Ausschüttung und Aufnahme von Neurotransmittern als auch die Verarbeitung von Informationen und der Weitertransport von Signalen entlang des Axons gehen mit chemischen und elektrischen Veränderungen an der Zellmembran einher. Ionenkanäle öffnen oder schließen sich und lassen positiv geladene Natrium-, Calcium- oder Kaliumionen oder negativ geladene Chlorionen in die Zelle hinein- oder aus ihr herausströmen, bzw. pumpen sie aktiv von einer Seite zur andern.
Dieses dynamische Gleichgewicht in der lebenden Zelle zu beobachten, ist nicht einfach und wird erst mit neueren Methoden nach und nach möglich. Klar ist, dass der dabei fortwährende Transport von Ionen und Neurotransmittern viel Energie frisst. Im Vergleich zu menschgemachten Computern und anderen Maschinen arbeitet das Gehirn allerdings – zumindest nach aktuellem Stand der Technik – viel effizienter. Sein Energieumsatz ähnelt mit knapp 20 Watt dem einer Energiesparlampe. Ein Laptop braucht locker das Doppelte, ist aber viel weniger leistungsfähig. Der Supercomputer SpiNNaker , der circa ein Prozent des menschlichen Gehirns zu simulieren vermag, braucht dafür 100 Megawatt.
| Abbildung 4. Neuronale Mitochondrien in den unterschiedlichen Kompartimenten des Neurons. (A) Mitochondrien werden mit Hilfe eines fluoreszierenden mitochondrialen Proteins (rot) sichtbar gemacht. (B) Schematische Darstellung der Kompartimente eines Neurons, wobei das lange Axon und mehrere Dendriten vom Zellkörper (Soma), der den Zellkern enthält, ausgehen. Axonale Mitochondrien sind klein und spärlich, während dendritische Mitochondrien größer sind und ein größeres Volumen des Prozesses einnehmen. Die Mitochondrien sind im Soma dicht gepackt. Maßstab: 20 µm, 10 µm in Vergrößerungen i und ii.(Bild von Redn. eingefügt; leicht modifiziert nach Seager R, et al (2020), Doi: https://doi.org/10.1042/NS20200008. Lizenz: cc-by) |
Die Dienstleister
Diese Aufgabe übernehmen allen voran die Astrozyten, sternförmige Gliazellen die mit ihren Endfüßchen wie Zapfsäulen an den Blutgefäßen hängen. Die Astrozyten könne die Blutgefäße verengen und auch erweitern, und sind damit ein wichtiger Regler für den energiereichen Blutfluss. Weitere Ausläufer legen Astrozyten handschuhartig um die postsynaptischen Regionen von Nervenzellen, wo der Energiebedarf besonders hoch ist. Im Gegensatz zu Neuronen können Astrozyten Glukose gut speichern, und zwar – genau wie die Leber – in Form von Glykogen. Außerdem reichen Astrozyten die Glukose, die sie dem Blut oder aus ihren Glykogenvorräten entnehmen, nicht direkt als Treibstoff an die Neuronen weiter, sondern verstoffwechseln sie zunächst zu Milchsäure. Dieses Zwischenprodukt kann dann nach nur einem einzigen weiteren Umwandlungsschritt direkt in die Atmungskette der Mitochondrien eingespeist werden. Abbildung 4.
Andere Gliazellen, die Oligodendrozyten, spielen ebenfalls eine Rolle bei der Versorgung der Axone. Bekannter als Urheber der isolierenden Myelinschicht, die Axone umwickelt und ihre elektrische Leitfähigkeit erhöht, halten Oligodendrozyten ebenfalls als Energielieferanten her. Auch sie schütten Milchsäure aus, die Mitochondrien im Axon dann schnell verwenden können. Die Liefermenge wird dabei an den Energiebedarf angepasst: je häufiger ein Neuron feuert, desto mehr Glukosetransporter bauen die Oligodendrozyten in ihre Membran ein, und desto mehr Energie können sie aus dem Blut aufnehmen und an ihr Axon weitergeben. Dieser maßgeschneiderte Lieferdienst gelingt, weil die Oligodendrozyten über spezielle Empfangsmoleküle „hören“ können, wie aktiv ein Neuron ist. Diese NMDA-Rezeptoren (für N-Methyl-D-Asparaginsäure) erkennen nämlich eine Verbindung, die jedes Mal freigesetzt wird, wenn eine Nervenzelle ein Aktionspotential „abfeuert“.
Mit diesem komplexen Versorgungsnetzwerk aus Gliazellen, Transportkanälen und Mitochondrien schaffen Neurone es im Idealfall, ihre Energieversorgung dynamisch im Gleichgewicht zu halten. Das gelingt allerdings nicht immer. Wird der Nachschub von Glukose und Sauerstoff aus dem Blut unterbrochen oder eingeschränkt, zum Beispiel bei einem Schlaganfall, kommt es schnell zum Zelltod und zu neuronalen Funktionsstörungen. Auch Entzündungsprozesse können die sensible Balance stören oder wichtige Komponenten der Energieversorgung schädigen. Neurologische Erkrankungen wie Demenz, Multiple Sklerose und die Parkinson-Krankheit haben fast immer eine energetische Komponente. Insofern dürfte der erfolgreiche Wahlslogan der Clinton-Kampagne leicht abgewandelt auch in der Hirnforschung noch länger Bestand haben: It’s the Energieversorgung, stupid!
* Der Artikel stammt von der Webseite www.dasGehirn.info, einer exzellenten Plattform mit dem Ziel "das Gehirn, seine Funktionen und seine Bedeutung für unser Fühlen, Denken und Handeln darzustellen – umfassend, verständlich, attraktiv und anschaulich in Wort, Bild und Ton." (Es ist ein Projekt der Gemeinnützigen Hertie-Stiftung und der Neurowissenschaftlichen Gesellschaft e.V. in Zusammenarbeit mit dem Zentrum für Kunst und Medientechnologie Karlsruhe). Im Fokus des Monats März steht das Thema "Energie". Der vorliegende Artikel ist am 15.3.2023 unter dem Titel: "Energieversorgung der Neurone" https://www.dasgehirn.info/grundlagen/energie/energieversorgung-der-neurone erschienen. Der unter einer cc-by-nc-sa Lizenz stehende Artikel wurde unverändert in den Blog gestellt, die Abbildungen von der Redaktion eingefügt.
Zum Weiterlesen
• Deitmer JW, et al: Unser hungriges Gehirn: Welche Rolle spielen Gliazellen bei der Energieversorgung? e-Neuroforum 2017, 23(1):2.12. Doi: https://doi.org/10.1515/nf.2016-1102
• Saab AS et al.: Oligodendroglial NMDA Receptors Regulate Glucose Import and Axonal Energy Metabolism. Neuron 2016 Jul; 91:119–132. Doi: http://dx.doi.org/10.1016/j.neuron.2016.05.016
• Seager R, et al: Mechanisms and roles of mitochondrial localisation and dynamics in neuronal function. Neuronal Signal 2020 Jun; 4(2): NS20200008. Doi: https://doi.org/10.1042/NS20200008
Artikel im ScienceBlog
Rund 60 Artikel zu verschiedenen Aspekten des Gehirns - u.a. zu den zellulären Komponenten und zur Signalübertragung - finden sich im Themenschwerpunkt Gehirn.
Ökologie ist eine treibende Kraft in der Verbreitung von Resistenzen gegen Aminoglykoside
Ökologie ist eine treibende Kraft in der Verbreitung von Resistenzen gegen AminoglykosideFr. 17.03.2023 — Redaktion
![]()
Die weltweite Ausbreitung der Antibiotikaresistenz in Umwelt und Bevölkerung könnte nicht - wie bisher angenommen - nur auf den übermäßigen Einsatz von Antibiotika in Landwirtschaft und Medizin zurückzuführen sein. Eine neue, enorm umfangreiche Analyse über die Verbreitung von Resistenzgenen gegen die seit den 1940er Jahren angewandte Antibiotika-Klasse der Aminoglykoside bringt wichtige neue Erkenntnisse über eine wesentliche Rolle von Ökologie (Biome) und menschlich generiertem Austausch (Importe durch Einwanderung und Wareneinfuhr). Die Rolle der Ökologie erscheint nicht verwunderlich, beruhen doch die meisten Antibiotika auf von Bakterien und Pilzen produzierten Naturstoffen, gegen die andere Mikroorgannismen - auch ganz ohne menschliche Einflussnahme - Resistenzen entwickelt haben und weiter entwickeln.*
Nach wie vor stellt Antibiotikaresistenz - die Fähigkeit von Bakterien, selbst die härtesten klinischen Behandlungen zu überleben - in der ganzen Welt ein massives Problem für die Gesundheit der Bevölkerung dar. Häufig wird der übermäßige Einsatz von Antibiotika in Medizin und Landwirtschaft als treibende Kraft für die Entstehung und Ausbreitung von Bakterien angesehen, die gegen Antibiotika resistent sind. In Gebieten, die stark von den Tätigkeiten des Menschen geprägt sind, kann exzessive Antibiotika Anwendung sicherlich die Selektion von Bakterien mit resistenten Genen gegen Antibiotika erklären. Dass aber Resistenzgene gegen klinisch relevante Antibiotika in Gegenden weitab von Krankenhäusern und landwirtschaftlichen Betrieben weit verbreitet sind, ist so nicht erklärbar. Tatsächlich wurden solche Gene sogar in so entlegenen Umwelten wie dem arktischen Permafrost und der Antarktis gefunden.
Nun berichten Léa Pradier und Stéphanie Bedhomme von der Universität Montpellier im Fachjournal eLife über die Ergebnisse einer Studie, die Licht in diese Fragestellung bringt [1]. Die Forscherinnen haben eine der bisher umfangreichsten Studien über Resistenzgene gegen Antibiotika durchgeführt, wobei der Schwerpunkt auf der Resistenz gegen Aminoglykoside lag.
Aminoglykoside
sind eine altbekannte, häufig angewandte Familie von Antibiotika, welche die Proteinsynthese von Bakterien blockieren. Der erste Vertreter dieser Klasse - das aus dem Bodenbakterium Streptomyces griseus isolierte Streptomycin - wurde bereits 1944 erfolgreich gegen Tuberkulose eingesetzt und erwies sich als erstes wirksames Mittel gegen gram-negative Bakterien. Später landeten noch weitere Isolate wie Kanamycin, Neomycin, Apramycin und chemisch modifizierte weiere Aminoglykoside in der Klinik. Bald tauchten aber erste Befunde zu resistenten Bakterienstämmen auf; zusammen mit einer Injektion erfordernden Applikationsart und ernsten Nebenwirkungen auf Niere und Gehör kam es zu einer stark reduzierten Anwendung von Aminoglykosiden in der Humanmedizin, die nun im wesentlichen als Mittel der zweiten Wahl oder als letzte Möglichkeit in der Behandlung von Infektionen mit gram-negativen Bakterien eingesetzt werden. Nach wie vor haben diese Antibiotika hohe Bedeutung in der Veterinärmedizin.
Die häufigste Ursache für die Entstehung von Aminoglykosidresistenz in klinischen Isolaten sind inaktivierende Enzyme, die die Übertragung chemischer Gruppen an Aminoglykosidmolekülen katalysieren: ein so modifiziertes Medikament kann dann nur mehr schlecht an seine Zielstruktur binden - es wurde unwirksam. Solche Aminoglykosid-modifizierende Enzyme (AMEs) sind biochemisch gut charakterisiert, ihre Bezeichnung basiert auf der Gruppe, die sie übertragen Die klassische Nomenklatur der AMEs basiert auf der Gruppe, die sie übertragen (d. h. Acetyltransferasen, AACs; Nukleotidyltransferasen, ANTs; und Phosphotransferasen, APHs).
Die Studie
Mit dem Ziel die zeitlichen, räumlichen und ökologischen Verteilungsmuster der Aminoglykosid-Resistenz zu beschreiben, haben Pradier und Bedhomme mehr als 160.000 öffentlich zugängliche Genome von Bakterien auf 27 Cluster von homologen Genen (CHGs - Gene, deren Sequenz einen gemeinsamen Vorläufer wahrscheinlich macht) untersucht, die für Aminoglykosid-modifizierende Enzyme kodieren (AME-Gene). Die Sequenzierungsdaten stammten von Bakterien aus allen Kontinenten (außer der Antarktis) und terrestrischen Ökoregionen (Biomen) und betreffen mit insgesamt 54 Bakterienstämmen (Phyla) die weitaus überwiegende Mehrheit aller Bakterien-Phyla. Die zur Sequenzierung herangezogenen Bakterien waren im Zeitraum zwischen 1885 und 2019 gesammelt worden, die meisten davon erst nach 1990.
Zusätzlich zu Ort und Datum der Probenahme wurden in der Studie auch die Anzahl der in den einzelnen Ländern konsumierten Antibiotika, die Handelsrouten und die menschliche Migration berücksichtigt.
Die Ergebnisse
| Abbildung 1 Globale Verteilung von Aminoglykosid-resistenten Bakterien. Die Größe der Kreise ist ein Maß für die Zahl der untersuchten Genome. (Bild von Redn. eingefügt und leicht modifiziert nach L. Pradier and S. Bedhomme (2023), [1]; Lizenz cc-by.) |
Resistenzgene gegen Aminoglykoside
zeigen eine weltweite geographische Verbreitung, sie kommen in allen Ökoregionen (Biomen) vor und wurden in 23 der 54 sequenzierten Bakterienstämme entdeckt. In den insgesamt rund 160 000 bakteriellen Genomen wurden 46 053 AME-Gene - d.i. in rund 25 % aller untersuchten Bakterien-Genomen - gefunden. In den meisten Regionen liegt die Häufigkeit der AME-Gen-tragenden Bakterien zwischen 20 % und 40 %; sie reicht von 10 % in Japan, Osteuropa und Ostafrika bis zu 50% in Indonesien, Mexiko und der Türkei. Abbildung 1.
Die Verteilung der für AME-kodierenden Gencluster
weist geographisch eine hohe Heterogenität auf (Abbildung 2). Bestimmte (für Acetyltransferasen AACf1 kodierende) Resistenzgene dominierten in der südlichen Hemisphäre von Afrika, Asien und Australien , während in den US und Westeuropa eine ausgewogene Mischung von Resistenzgenen detektiert wurde.
| Abbildung 2 Wie sich die 27 für die Aminoglykosid-modifizierenden Enzyme (AME) kodierenden Gencluster global verteilen. Die Bezeichnung der Gencluster basiert auf der Gruppe, die die entsprechenden Enzyme übertragen: AACs = Acetyltransferasen; ANTs = Nukleotidyltransferasen, und APHs = Phosphotransferasen). (Bild von Redn. eingefügt und leicht modifiziert nach L. Pradier and S. Bedhomme (2023), [1]; Lizenz cc-by.) |
Die Forscherinnen fanden heraus, dass die Häufigkeit von Aminoglykosid-Resistenzgenen zwischen den 1940er und 1980er Jahren zugenommen hat, was wahrscheinlich auf den verstärkten Einsatz von Aminoglykosid-Antibiotika nach der Entdeckung von Streptomycin im Jahr 1943 zurückzuführen ist, dann aber - trotz eines allgemeinen Rückgangs des Verbrauchs - bei einer Prävalenz von etwa 30 % blieb. Entscheidend ist, dass sie auch entdeckten, dass etwa 40 % der Resistenzgene potenziell mobil sind, d. h. leicht zwischen Bakterien ausgetauscht werden können.
Vorkommen in den Biomen
Pradier und Bedhomme stellten außerdem fest, dass antibiotikaresistente Bakterien in den meisten Biomen vorkommen, nicht nur in Krankenhäusern und landwirtschaftlichen Betrieben. (Abbildung 3).
| Abbildung 3.Resistenzgene gegen Aminoglykoside wurden bereits 4 Jahrzehnte vor der erstmaligen klinischen Anwendung entdeckt (oben) und sie sind nun in allen untersuchten Biomen in einer Häufigkeit von knapp über 0 bis 30 % präsent (unten: blau bis rot; leere Kästchen bedeuten 0 Resistenzgene). Die Bezeichnung der Gencluster basiert auf der Gruppe, welche die entsprechenden Enzyme übertragen: AACs = Acetyltransferasen; ANTs = Nukleotidyltransferasen, und APHs = Phosphotransferasen. (Bild von Redn. eingefügt und leicht modifiziert nach L. Pradier and S. Bedhomme (2023), [1]; Lizenz cc-by) |
Darüber hinaus fanden sie, dass die Prävalenz von Aminoglykosid-Resistenzgenen von Biom zu Biom stärker variierte, als dies mit der geografischen Lage von Menschen oder der Menge der verwendeten Antibiotika der Fall war. Für das Biom Mensch bedeutet dies, dass die in einem Land festgestellte Antibiotikaresistenz viel wahrscheinlicher mit der Antibiotikaresistenz in einem entfernteren Land korreliert ist, als mit der in der näheren Umgebung im Biom Boden oder im Biom Tiere.
Außerdem entdeckten sie, dass Biome wie Boden und Abwasser wahrscheinlich eine Schlüsselrolle bei der Verbreitung der Gene für die Antibiotikaresistenz über verschiedene Biome hinweg spielen. Eine Analyse der in Europa zwischen 1997 und 2018 gesammelten Proben zeigt deutlich wovon die Häufigkeit des Vorkommens von 16 AME-Gencluster abhängt: in 80 % der Fälle ist die Ökologie treibende Kraft in der Verbreitung der Resistenzgene, in 13 % menschlich generierter Austausch (Importe durch Einwanderung und Wareneinfuhr) und nur in 7 % der Fälle (übermäßige ) Anwendung.
Fragen
Diese Ergebnisse werfen wichtige Fragen zu den Mechanismen auf, die der Ausbreitung der Antibiotikaresistenz zugrunde liegen.
- Welche Faktoren begünstigen die Ausbreitung der Antibiotikaresistenz in Umgebungen, die nicht durch menschliche Aktivitäten beeinflusst werden?
- Können wir die von den Aminoglykosiden erhaltenen Ergebnisse auf alle anderen Antibiotikaklassen übertragen?
- Ist es möglich, dass die Antibiotikaresistenz eher auf Interaktionen mit lokalen mikrobiellen Gemeinschaften zurückzuführen ist, als auf die Exposition gegenüber kommerziellen Antibiotika?
- Verbreiten sich die Gene für die Antibiotikaresistenz in den pathogenen Bakterien, die für Infektionen bei Mensch und Tier verantwortlich sind, auf die gleiche Weise wie in den nicht-pathogenen Bakterien?
- Was begrenzt angesichts des Ausmaßes des Selektionsdrucks durch die menschliche Verschmutzung die Ausbreitung von Antibiotikaresistenzgenen zwischen Biomen, insbesondere angesichts des großen Anteils von Genen, die auf mobilen Elementen sitzen?
Es kann gut sein, dass der Verbrauch immer noch eine überragende Rolle spielt, wenn es um die Resistenz gegen Antibiotika geht, die zur Behandlung von Infektionen eingesetzt werden, insbesondere beim Menschen und in klinischen Biomen. Dennoch ist klar, dass wir der Rolle der Umwelt bei der Formulierung von Plänen zur Bekämpfung der Antibiotikaresistenz auf globaler Ebene mehr Aufmerksamkeit schenken müssen.
[1] Pradier L, Bedhomme S. 2023. Ecology, more than antibiotics consumption, is the major predictor for the global distribution of aminoglycoside-modifying enzymes. eLife 12:e77015. doi: 10.7554/eLife.77015
*Eine Zusammenfassung des Artikels von Léa Pradier und Stéphanie Bedhomme, 2023, [1] verfasst von Carolina Oliveira de Santana et al., ist am 8.3.2023 unter dem Titel "Antibiotic Resistance: A mobile Target " im eLife Magazin erschienen: : https://elifesciences.org/articles/86697 . Der Text wurde von der Redaktion ins Deutsche übersetzt und mit einigen Textststellen und 4 Abbildungen aus dem Originaltext [1] plus Legenden ergänzt. eLife ist ein open access Journal, alle Inhalte stehen unter einer cc-by Lizenz.
Antibiotika-Resistenz im ScienceBlog
Redaktion, 11.12.2022: Die globale Krise der Antibiotikaresistenz und diesbezügliches Wissen und Verhaltensweisen der EU-Bürger (Spezial Eurobarometer 522)
08.07.2021, Redaktion: Phagen und Vakzinen im Kampf gegen Antibiotika-resistente Bakterien
Karin Moelling, 29.08.2019: Ein Comeback der Phagentherapie?
Karin Moelling, 4.07.2019: Viren gegen multiresistente Bakterien. Teil 1: Was sind Phagen?
Inge Schuster, 23.09.2016: Gehen wir auf eine Post-Antibiotika Ära zu?
Gottfried Schatz, 30.05.2013: Planet der Mikroben — Warum wir Infektionskrankheiten nie endgültig besiegen werden
Laser - Technologie aus dem Quantenland mit unzähligen Anwendungsmöglichkeiten
Laser - Technologie aus dem Quantenland mit unzähligen AnwendungsmöglichkeitenDo, 09.03.2023 — Roland Wengenmayr
 Laser sind heute überall. Sie befeuern die Glasfasernetze der Telekommunikation, machen dem Internet per Lichtpost Beine, stecken in Laserpointern oder -scannern. Starke Industrielaser bearbeiten Werkstoffe. Laser spüren in der Atmosphäre umweltschädliche Gase auf, in Satelliten erfassen sie kleinste Veränderungen auf der Erde. Jüngste Generationen „optischer“ Atomuhren messen mit Lasern die Zeit immer genauer, Laser steuern künftige Quantencomputer. Auch in der Medizin werden Laser vielfältig genutzt und können in Zukunft noch mehr leisten: Das BIRD-Team, darunter Forschende vom Max-Planck-Institut für Quantenoptik in Garching, will Blutproben auf winzigste molekulare Spuren zur Krebsfrüherkennung durchleuchten. Das erfordert Laser mit extrem kurzen Lichtpulsen, das Spezialgebiet von Ferenc Krausz, Direktor am Institut.*
Laser sind heute überall. Sie befeuern die Glasfasernetze der Telekommunikation, machen dem Internet per Lichtpost Beine, stecken in Laserpointern oder -scannern. Starke Industrielaser bearbeiten Werkstoffe. Laser spüren in der Atmosphäre umweltschädliche Gase auf, in Satelliten erfassen sie kleinste Veränderungen auf der Erde. Jüngste Generationen „optischer“ Atomuhren messen mit Lasern die Zeit immer genauer, Laser steuern künftige Quantencomputer. Auch in der Medizin werden Laser vielfältig genutzt und können in Zukunft noch mehr leisten: Das BIRD-Team, darunter Forschende vom Max-Planck-Institut für Quantenoptik in Garching, will Blutproben auf winzigste molekulare Spuren zur Krebsfrüherkennung durchleuchten. Das erfordert Laser mit extrem kurzen Lichtpulsen, das Spezialgebiet von Ferenc Krausz, Direktor am Institut.*
Ort des Geschehens: ein Labor der Hughes Aircraft Company in der kalifornischen Stadt Culver. Am 16. Mai 1960 lässt dort der amerikanische Physiker Theodore Maiman eine spiralförmige Gasentladungslampe aufblitzen. In ihr steckt ein stabförmiger Rubinkristall, dessen Enden verspiegelt sind. Das Blitzlicht löst in dem Rubin den ersten Laserpuls der Welt aus. Bald konnte Maiman der Presse mit seinem Rubinlaser kräftige rote Laserblitze vorführen. Von ihm hörten die Anwesenden auch zum ersten Mal das Kunstwort „Laser“, für „Light Amplification by Stimulated Emission of Radiation“, auf Deutsch „Lichtverstärkung durch stimulierte Emission von Strahlung”. Heute sind Laser nicht nur Schlüsseltechnologien, sondern auch in der Grundlagenforschung allgegenwärtig. Forschende schauen mit den stärksten Laseranlagen der Welt immer tiefer in die Materie hinein oder verschmelzen damit Atomkerne. Mit schwächerem Laserlicht bremsen sie die Wärmebewegung der umherflitzenden Atome ab. Sie kühlen so die Atome bis fast auf den absoluten Temperaturnullpunkt hinunter und machen mit ihnen Quantenexperimente. Oder sie entwickeln ganz neue medizinische Methoden. Abbildung 1.
|
Abbildung 1. Laser für Krebsfrüherkennung. Christina Hofer an einem der vom BIRD-Team entwickelten Geräte: Mit ultrakurzen Laserblitzen soll es Krebs im Frühstadium in Blutproben aufspüren. Die zukünftigen Geräte sollen Tausende von Blutproben vollautomatisch analysieren. (Bild © Thorsten Naeser, Attoworld). |
Der Laser bezieht seine Stärke aus reiner Quantenphysik. „Es ist mir ein prächtiges Licht über die Absorption und Emission von Strahlung aufgegangen, [es ist] alles ganz quantisch“, schrieb Albert Einstein 1916 begeistert seinem Freund Emile Besso. Einstein legte damals die theoretischen Grundlagen für den Laser, allerdings ohne diesen vorauszuahnen. Schon 1905 hatte er anhand des Fotoeffekts gezeigt, dass Licht und Materie ihre Energie nur in festen Paketen austauschen können. Beim Energieaustausch mit Materie verhält sich Licht also eher wie Teilchen in Form von Energiequanten, den Photonen. Sonst zeigt Licht oft den Charakter einer Welle. Dieser Welle-Teilchen-Dualismus zeichnet alle Objekte der Quantenwelt aus. Der Laser nutzt beide Eigenschaften.
Angeregte Elektronen
Atome können Licht nur als Energiequanten aufnehmen (absorbieren) oder abgeben (emittieren). Trifft ein Photon passender Energie auf ein Elektron eines Atoms, dann kann das Elektron das Photon absorbieren und in ein Orbital höherer Energie hüpfen. Umgekehrt kann das Elektron wieder auf seinen alten Platz herunterfallen, dabei emittiert es das überschüssige Energiequant erneut als Photon: Das Atom leuchtet.
Die Elektronen der Atomhülle lassen sich gruppenweise nach Energiestufen ordnen. Jeder Stufenabstand entspricht einem Quantensprung zwischen zwei benachbarten Energieniveaus in der Elektronenhülle. Eine „klassische“ Lichtquelle wie die Sonne oder eine Glühbirne besteht nun aus vielen verschiedenen Atomen, die wild durcheinander leuchten. Verschiedene Atomsorten mit unterschiedlichen Quantenübergängen emittieren Photonen vieler Frequenzen, also Energien oder Farben. Ihr unkoordiniertes Abstrahlen produziert ein optisches Chaos mit einer breiten Farbmischung, die ein nahezu weißes Licht ergibt.
In einem Laser geht es dagegen extrem geordnet zu. Er strahlt Licht in einer sehr reinen Farbe ab, das er kräftig verstärkt. Dazu füttert er es ständig mit neuen Energiepaketen, mit Photonen in der exakt passenden Lichtfarbe. Dieser Verstärkungsmechanismus benutzt nur wenige, aufeinander abgestimmte Atomsorten, die zusammenwirken. In diesem „Atomkollektiv“ trägt wiederum ein einziger Quantensprung zwischen zwei speziell geeigneten Energieniveaus zur Verstärkung bei. Das ist der Laserübergang. Zudem emittieren diese Atome ihre Lichtquanten auch noch im präzisen Gleichtakt mit der Lichtwelle, die sich im Laser wie in einem Resonanzkörper aufschwingt. Deshalb besteht Laserlicht normalerweise aus sehr langen Lichtwellenzügen, im Gegensatz zum Glühbirnenlicht.
Pingpong mit Quanten
Für die Verstärkung im Laser sorgt ein besonderer Effekt. Diese stimulierte Emission beschrieb Albert Einstein 1917, kurz nach seinem Brief an Besso. Er hatte erkannt, dass Atome nicht nur spontan Lichtquanten abstrahlen können, diese spontane Emission dominiert das Leuchten klassischer Lichtquellen. Wenn ein Photon an einem Atom vorbeifliegt, kann es dieses auch gezielt zur Lichtemission anregen, indem es ein Elektron von einem höheren Energieniveau sozusagen herunterschüttelt. Das Atom strahlt dabei ein Photon mit exakt der gleichen Energie – also Farbe – ab. Im Laser sind allerdings viele gleichartige Atome gemeinsam an der stimulierten Emission beteiligt. Damit die Lichtverstärkung funktioniert, müssen diese Atome zusammen zwei weitere Voraussetzungen erfüllen: Erstens müssen sich Elektronen auf dem höheren Energieniveau des Laserübergangs befinden, zweitens muss dieser Laserübergang genau der Energie der vorbeikommenden Photonen entsprechen (Abbildung 2).
|
Abbildung 2. Stimulierte Emission. Laserübergang zwischen Grundzustand und angeregtem Zustand der Atome im aktiven Lasermedium. Von links nach rechts: Ein Elektron wird in den oberen Zustand gepumpt, dann kommt ein Photon passender Energie vorbei und stimuliert das Elektron, im Gleichtakt ein zweites Photon gleicher Energie abzustrahlen, wobei es wieder in den Grundzustand zurückfällt. Danach wiederholt sich das Spiel immer weiter, solange der Laser läuft. © R. Wengenmayr / CC BY-NC-SA 4.0 |
Damit das alles im Laser zusammenpasst, benötigt er einen optischen Resonator aus zwei Spiegeln. Ihr Abstand ist exakt auf ein Vielfaches der zu verstärkenden Laserwellenlänge justiert. Zwischen ihnen laufen die Quanten des Laserlichts wie Pingpong-Bälle hin und her (Abbildung 3). Es gibt auch Laser mit noch mehr Resonatorspiegeln. Einer der Spiegel muss teilweise lichtdurchlässig sein. Durch ihn entwischt ein kleiner Anteil der Photonen und formt den Laserstrahl. Durch dieses gewollte Leck verliert der Laser allerdings permanent Energie. Deshalb benötigt er einen Lichtverstärker, der immer neue Elektronen im Laserübergang nachliefert. Das macht das aktive Lasermedium. Es steckt im Resonator und enthält die Atome, die im Laserübergang leuchten sollen. Wenn die Photonen beim Spiegel-Pingpong immer wieder das Lasermedium durchqueren, „schütteln“ sie die Elektronen dieser Atome so, dass sie in Lawinen den Laserübergang hinunterfallen. Diese stimulierte Emission wirft immer neue Photonen ins Pingpong-Spiel, und das Lichtfeld im Resonator schwillt an. Allerdings fehlt noch eine Zutat: Der Laser braucht noch eine „Pumpe“, die immer neue Elektronen in das sich entleerende obere Niveau des Laserübergangs nachliefert. Dieses permanente Pumpen versorgt den Laser auch mit Energie. Manche Laser strahlen in Pulsen, andere kontinuierlich. Zur ersten Kategorie gehört Maimans Rubinlaser, dessen Pumpe das Blitzlicht der Gasentladungslampe war. Neben diesen optisch gepumpten Lasern gibt es auch solche, in denen ein elektrischer Strom den Laserprozess pumpt. Dazu zählen die weit verbreiteten Laserdioden.
|
Abbildung 3. Aufbau eines Lasers. Beschreibung im Text. © R. Wengenmayr / CC BY-NC-SA 4.0 |
Pyramide auf den Kopf gestellt
Das Lasermedium muss die passende Atomsorte mit dem Quantensprung in der gewünschten Lichtfarbe enthalten. Überdies braucht es noch eine weitere wichtige Eigenschaft: Die Elektronen müssen lange genug auf dem oberen Energieniveau seines Laserübergangs verweilen. Sie dürfen nicht zu anfällig für die spontane Emission sein, die sich nicht völlig ausschalten lässt. Die spontane Emission sabotiert die koordinierte Lichtverstärkung, denn sie lässt die Elektronen außerhalb des Gleichtakts ins niedrigere Energieniveau fallen. Zudem muss das Pumpen das untere Energieniveau schnell genug leeren. Nur wenn die Elektronen in dem unteren Quantenzustand genug Platz finden, können sie ungehindert dort hineinfallen.
Hat das Lasermedium diese Eigenschaften, dann kann das Pumpen in ihm den richtigen Betriebszustand herstellen, die Besetzungsinversion. Normalerweise verteilen sich die Elektronen über die Energieniveaus der Atome wie in einer Pyramide: Die unteren Niveaus sind stark mit Elektronen bevölkert, die oberen immer dünner. Die Natur spart gerne Energie. Das Pumpen muss diese Besetzungspyramide nun auf den Kopf stellen (Abbildung 4): Im oberen Energieniveau des Laserübergangs müssen sich mehr Elektronen als im unteren sammeln. Erst dann finden die vorbeikommenden Photonen genügend Elektronen, die sich „herunterschütteln“ lassen
|
Abbildung 4. Besetzungsinversion. Normalerweise ist das untere Energieniveau der Atome im Lasermedium stärker mit Elektronen (blaue Kugeln) besetzt als das obere. Die Besetzungsinversion stellt diese Pyramide auf den Kopf. Im Laser schafft sie die Voraussetzung für Lichtverstärkung über induzierte Emission. © R. Wengenmayr / CC BY-NC-SA 4.0 |
Allerdings geschieht das Pumpen in den meisten Lasern nicht in einem einzigen Quantensprung, sondern mit Zwischen zuständen. Außerdem wird die Konstruktion eines Lasers umso anspruchsvoller, je kürzer die Wellenlänge seines Lichts sein soll. Mit schrumpfender Wellenlänge wächst nämlich die Energie der Photonen und damit auch der nötige Quantensprung im Laserübergang. Je höher das obere Energieniveau im Vergleich zum unteren liegt, desto anfälliger reagieren die Elektronen dort auf die sabotierende spontane Emission. Immer weniger von ihnen fallen im Rhythmus der induzierten Emission herunter. Die frühen Laser leuchteten aus diesem Grund im langwelligen roten oder infraroten Spektralbereich. Als der Japaner Shuji Nakamura 1995 die erste blau leuchtende Laserdiode vorstellte, war das eine Sensation. Noch viel kurzwelligere Röntgenlaser wie der European XFEL in Hamburg funktionieren wegen dieses Problems ganz anders. Sie beschleunigen freie Elektronen fast auf Lichtgeschwindigkeit und jagen sie dann durch ein wellenförmiges Magnetfeld: Das zwingt die Elektronen, nach vorne gerichtetes Röntgenlaserlicht abzustrahlen.
Mit Lasern gegen Krebs
Es gibt Laser, die kein „typisches“ Laserlicht erzeugen. Sie strahlen keine langen Lichtwellenzüge in einer reinen Farbe ab, sondern produzieren extrem kurze Pulse in einem Gemisch von Frequenzen. Besonders wichtig sind Femtosekundenpulse, denn auf dieser Zeitskala ändern sich chemische Bindungen und schwingen die Moleküle. Eine Femtosekunde ist der 1015-te Teil einer Sekunde (0,000000000000001 Sekunden). Selbst Licht kommt in einer Femtosekunde nur 300 Nanometer weit – das entspricht dem Durchmesser eines Herpes-Virus.
Ein Pionier der Ultrakurzpuls-Forschung ist Ferenc Krausz, Direktor am Max-Planck-Institut für Quantenoptik. Dieses Team, zu dem auch Forschende der Ludwig-Maximilians-Universität (LMU) in München gehören, will einen Traum verwirklichen: Ultrakurze Laserlichtblitze sollen es ermöglichen, bei Blut proben „gesund“ von „krank“ zu unterscheiden. Dazu nutzt es Infrarot-Laserpulse mit nur wenigen Femtosekunden Dauer. Im Messergebnis soll eine lernfähige Software nach Spuren von Molekülen suchen, die etwas über den Gesundheitszustand aussagen. „Zuerst wollen wir die häufigsten Krebsarten nachweisen können“, erklärt Mihaela Žigman, Leiterin der Breitband-Infrarotdiagnostik-Gruppe (BIRD): „Lungenkrebs, Brustkrebs, Prostatakrebs und Blasenkrebs.“ Die neue Technik soll Krebs schon früh am molekularen „Fingerabdruck“ im Blut entdecken, lange bevor sich gefährliche Tumore im Körper verbreiten können. Die Molekularbiologin ist zudem Direktorin am Center for Molecular Fingerprinting in Budapest. Von dort erhalten die Garchinger Tausende von Blutproben, die der Biologe Frank Fleischmann in einem speziellen Tiefkühlverfahren mit flüssigem Stickstoff für viele Jahre haltbar einlagert.
Die Entwicklung der nötigen komplexen Lasertechnologie leitet der Physiker Alexander Weigel. Er und Fleischmann führen im Garchinger Labor der LMU zu einem der gerade laufenden Geräte. „Dieser sogenannte Infrasampler (Abbildung 1) zieht automatisch Proben ein“, erklärt Fleischmann. Dann durchleuchtet das Gerät sie mit dem Infrarot-Laserlicht. Dessen kurzer Puls entspricht gerade mal einer einzigen Lichtschwingung und enthält viele verschiedene Infrarotfrequenzen. Solche Infrarot-Laserpulse regen dann unterschiedlichen Biomoleküle in den Blutproben zu charakteristischen Schwingungen an. „Der kurze, starke Infrarotpuls ist wie ein Gongschlag“, sagt Weigel. Jedes Biomolekül entspricht einem kleinen Gong, der nach dem Schlag in einer charakteristischen Frequenz nachschwingt. Dieses Nachschwingen macht die Moleküle selbst zu kleinen Sendern, die Infrarotlicht aussenden.
Das gesendete Antwortlicht ist extrem schwach, weshalb das Team es mit einer speziellen Empfängermethode aufzeichnet, elektro-optisches Sampling genannt. Nach dem kurzen, sehr hellen Infrarotlichtpuls, dem „Gongschlag“, wird es sofort dunkel. Nun lässt sich ungestört das Antwortlicht analysieren. Dazu tastet das elektro-optische Sampling die Wellenform des schwingenden elektrischen Feldes vom Antwortlicht ab. „Allerdings wäre eine Elektronik dafür viel zu langsam“, erklärt Weigel. Das Infrarotlicht, das von den Molekülen ausgesandt wird, schwingt nämlich mit einer Frequenz von bis zu mehreren zehn Terahertz. Ein Terahertz entspricht einer Billion, oder 1012 Schwingungen pro Sekunde. Entsprechend schnell muss der „Scanner“ sein, um die Wellenformen der Schwingungen zu erwischen. Die Garchinger nutzen dafür Femtosekunden-Laserpulse, die sie mit dem Antwortlicht überlagern. Diese kurzen Laserpulse tasten dann die elektrischen Feldschwingungen des Antwortlichtes ab, ungefähr wie ein Scannerstreifen, der über ein Blatt Papier wandert. Damit das funktioniert, muss das anregende Infrarotlicht echtes Laserlicht sein, eine starke Infrarotlampe wäre nicht genug. Nur dann lässt sich das Abtastlicht exakt auf die Form der abzutastenden Welle abstimmen. Das sich ergebende Lichtsignal kann dann aufgenommen und elektronisch verarbeitet werden, während das Instrument die nächste Probe vorbereitet. Das gemessene Signal gleicht einem „Fingerabdruck“ der Moleküle. Es zeigt so Änderungen im Blut an, die durch Krankheiten wie Krebs hervorgerufen werden. Mit Hilfe von Maschinenlernen wird das System an vielen Proben darauf trainiert, von selbst die gesuchten Änderungen der molekularen Fingerabdrücke zu erkennen.
Nicht nur die Signale der Moleküle selbst sind sehr schwach. Die gesuchten Moleküle sind auch in teils extrem geringen Konzentrationen in der Blutprobe vorhanden. Und zum Durchleuchten müssen diese Proben auch noch selbst wenige Mikroliter winzig sein. Ein Infrasampler muss darin die gesuchten Moleküle in Mengen von wenigen Nanogramm nachweisen können, erklärt Mihaela Žigman: „Noch ist das BIRD-Projekt Grundlagenforschung und frühestens in zehn bis 15 Jahren reif für den breiten Einsatz in der Medizin.“ Doch dann wäre es ein großer Fortschritt für die Gesundheitsvorsorge.
* Der Artikel ist unter dem Titel: "Wunderlampe aus dem Quantenland – wie der Laser den Alltag und die Medizin erobert" im TECHMAX 06-Heft der Max-Planck-Gesellschaft im Winter 2022/23 in aktualisierter Form erschienen ((https://www.max-wissen.de/max-hefte/techmax-06-laser/). Mit Ausnahme des Titels wurde der unter einer cc-by-nc-sa Lizenz stehende Artikel unverändert in den Blog übernommen.
Videos zum Thema
Max-Planck-Gesellschaft: Laser - Licht in Formation. Video 5:26 min. https://www.youtube.com/watch?v=xFy9DNN0j4M
Max-Planck-Gesellschaft: Laser - Der schnellste Blitz der Welt. Video 8:30 min. https://www.youtube.com/watch?v=6zxzJqvzZMY&t=8s
Die ökonomischen Kosten von Krebserkrankungen
Die ökonomischen Kosten von KrebserkrankungenFr, 24.02.2023 — IIASA
Ein internationales Team unter Beteiligung von IIASA-Forschern hat die globalen wirtschaftlichen Kosten, die auf Grund der aktuellen Verluste bei den Arbeits- und Behandlungskosten von Krebserkrankungen entstehen, für einen künftigen Zeitraum von drei Jahrzehnten ab 2020 errechnet. Der Schätzung zufolge werden sich diese Kosten zwischen 2020 und 2050 auf 25,2 Billionen Dollar (zu internationalen Preisen von 2017) belaufen, was 0,55 % des globalen jährlichen Bruttoinlandsprodukts entspricht. Die Krebsformen, die dazu am meisten beitragen (insgesamt 47 % der Gesamtkosten) wurden als Krebs von Lunge/Atmungstrakt, Kolon-und Rektumkarzinom, Brustkrebs, Leberkrebs und Leukämie identifiziert. Die Inzidenz der ersten 4 Krebsarten kann durch Präventivmaßnahmen reduziert werden - Investitionen in solche Maßnahmen erscheinen für den Schutz der globalen Gesundheit und des wirtschaftlichen Wohlergehens unerlässlich.*
Krebserkrankungen zählen weltweit zu den häufigsten Todesursachen und fordern jedes Jahr fast 10 Millionen Menschenleben. Die Häufigkeit der Krebserkrankungen nimmt zu; schuld daran sind das Altern der Bevölkerung, Rauchen, Alkoholkonsum, ungesunde Ernährung, Bewegungsmangel und Luftverschmutzung. Krebs bedeutet nicht nur Leid für das Leben der Menschen, sondern auch Schaden für die Wirtschaft - infolge von Einbußen in der Produktivität, Ausfall von Arbeitskräften und Investitionsrückgängen stellen Krebserkrankungen eine enorme finanzielle Belastung für die Länder dar.
Die durch Krebserkrankungen verursachten gesundheitlichen und wirtschaftlichen Belastungen werden klar als dringende Anliegen gesehen: dies geht hervor aus der von Präsident Biden erneut ins Leben gerufenen Cancer Moonshot Initiative, aus dem Globalen Aktionsplan zu Prävalenz und Kontrolle von nicht-übertragbaren Krankheiten der WHO sowie aus dem von den Vereinten Nationen definierten Ziel 3.4 für nachhaltige Entwicklung (SDG 3.4), das anstrebt mit Hilfe von Prävention und Therapie die durch nicht-übertragbaren Krankheiten bedingte vorzeitige Sterblichkeit bis 2030 um ein Drittel zu senken. Trotz dieser dringlichen Sachlage sind die globalen wirtschaftlichen Kosten von Krebserkrankungen bis jetzt noch nicht umfassend untersucht worden.
Um diese Lücke zu schließen und die politischen Entscheidungsträgern dabei zu unterstützen, die Zunahme von krebsbedingten Todesfällen und Invalidität einzudämmen, hat sich ein internationales Forscherteam daran gemacht, die wirtschaftlichen Kosten von 29 Krebsarten in 204 Ländern und Regionen abzuschätzen, die insgesamt die meisten Länder der Welt abdecken. Die Studie wurde in der Fachzeitschrift Journal of the American Medical Association (JAMA) - Oncology veröffentlicht [1]; darin wurde ein umfassendes Modellierungs-Framework angewandt, um die makroökonomischen Kosten von Krebs in Form von entgangenem BIP zu schätzen.
"Viele der aktuellen wirtschaftlichen Studien über Krebs sind rein statisch und lassen die künftigen Folgen der aktuellen Verluste bei den Arbeits- und Behandlungskosten außer Acht", sagt Michael Kuhn, Direktor des IIASA Economic Frontiers Program, der an der Studie mitgewirkt hat. "Unsere Arbeit ist insofern bahnbrechend, als sie die makroökonomischen Kosten von Krebs mit Hilfe eines Modells abschätzt, das viele der wirtschaftlichen Anpassungsmechanismen einbezieht und Veränderungen des Arbeitsangebots aufgrund von Krebsmortalität und -morbidität sowie den Verlust von Kapitalinvestitionen im Zusammenhang mit den Behandlungskosten berücksichtigt."
Die Studie schätzt die weltweiten wirtschaftlichen Kosten von Krebserkrankungen für den Zeitraum 2020-2050 auf rund 25,2 Billionen US-Dollar (INT$, zu konstanten Preisen von 2017), was einer jährlichen Steuer von 0,55 % auf das globale Bruttoinlandsprodukt entspricht. Die Forscher haben auch die Krebsarten identifiziert, welche die höchste wirtschaftliche Belastung verursachen, wobei Lungenkrebs(15,4 %) an erster Stelle steht, gefolgt von Colon- und Rektumkarzinom (10,9 %), Brustkrebs (7,7 %), Leberkrebs (6,5 %) und Leukämie (6,3). Für die einzelnen Länder und Regionen ist dies in der Abbildung unten dargestellt.
| Abbildung. Für die einzelnen Länder ist jeweils die Krebsart mit den höchsten wirtschaftlichen Kosten im Zeitraum 2020-2050 dargestellt. (TBL: Tracheal-, Bronchial- und Lungenkrebs). Beschriftung von Redn.verändert. |
Weiters zeigen die Ergebnisse, dass Gesundheitskosten und ökonomische Kosten von Krebserkrankungen ungleich zwischen den Ländern und Regionen verteilt sind. China und die Vereinigten Staaten sind in absoluten Zahlen mit 24,1 % bzw. 20,8 % der globalen ökonomischen Gesamtkosten von Krebserkrankungen am stärksten betroffen. Während die meisten Krebstodesfälle in Ländern mit niedrigem und mittlerem Einkommen auftreten, beträgt ihr Anteil an den wirtschaftlichen Kosten von Krebserkrankungen nur etwa die Hälfte.
"Die vier Krebsarten, welche die Wirtschaft am stärksten schädigen, lassen sich alle durch primäre und sekundäre Präventionsmaßnahmen betreffend Rauchen, Ernährung und Alkohol sowie verstärkte Vorsorgeuntersuchungen bekämpfen", so Kuhn. "Dies zeigt, dass es weltweit ein großes Potenzial für politische Maßnahmen gibt, die dazu beitragen können, die wechselseitige Beziehung von hoher Krankheitslast und starker wirtschaftlicher Belastung zu dämpfen."
Die Autoren betonen, dass Investitionen in wirksame Maßnahmen des öffentlichen Gesundheitswesens zur Verringerung der Krebsbelastung für den Schutz der globalen Gesundheit und des wirtschaftlichen Wohlergehens unerlässlich sind.
[1] Chen, S., Cao, Z., Prettner, K., Kuhn, M., Yang, J., Jiao, L., Wang, Z., Li, W., Geldsetzer, P., Bärnighausen, T., Bloom, D.E, Wang, C. (2022). The global economic cost of 29 cancers from 2020 to 2050: Estimates and projections for 204 countries and territories. Journal of the American Medical Association (JAMA) – Oncology DOI: 10.1001/jamaoncol.2022.7826
*Der am 24.Feber 2023 als Presseaussendung auf der IIASA-Webseite unter dem Titel "The Price of Cancer" erschienene Artikel https://iiasa.ac.at/news/feb-2023/price-of-cancerwurde von der Redaktion möglichst wortgetreu übersetzt. IIASA hat freundlicherweise der Veröffentlichung der von uns übersetzten Inhalte seiner Website und Presseaussendungen in unserem Blog zugestimmt.
Menschliche Avatare auf Silikonchips - ersetzen sie Tierversuche in der Arzneimittel-Entwicklung?
Menschliche Avatare auf Silikonchips - ersetzen sie Tierversuche in der Arzneimittel-Entwicklung?Sa 04.03.2023 — Inge Schuster

![]() Ende Dezember 2022 hat der US-amerikanische Präsident ein Gesetz unterzeichnet, das die Zulassung neuer Medikamente ohne die bislang erforderlichen, aufwändigen Tierversuche ermöglicht. An deren Stelle können human-relevante alternative Methoden, wie menschliche Miniorgane - sogenannte Organoide -, Multiorganchips menschlicher Zellen und Computermodelle treten, sofern diese validierte Verfahren zum Nachweis von Wirksamkeit und Toxizität sind.
Ende Dezember 2022 hat der US-amerikanische Präsident ein Gesetz unterzeichnet, das die Zulassung neuer Medikamente ohne die bislang erforderlichen, aufwändigen Tierversuche ermöglicht. An deren Stelle können human-relevante alternative Methoden, wie menschliche Miniorgane - sogenannte Organoide -, Multiorganchips menschlicher Zellen und Computermodelle treten, sofern diese validierte Verfahren zum Nachweis von Wirksamkeit und Toxizität sind.
Seit Beginn des 20. Jahrhunderts haben wir einen beispiellosen Boom von neuen wirksamen Medikamenten erlebt. Gab es anfangs erst wenige Arzneistoffe (das sind die aktiven Wirkstoffe in Medikamenten), die diese Bezeichnung heute noch verdienen - etwa die aus Heilpflanzen aufgereinigten Naturstoffe Chinin oder Morphin oder chemisch modifizierte Naturstoffe wie Aspirin oder Pyramidon -, so halten wir nun bereits bei rund 15 000 Wirkstoffen (https://go.drugbank.com/stats). Die neuen, gegen verschiedenste Krankheiten wirksamen Medikamente haben uns Menschen (zumindest in der westlichen Welt) zu einer erhöhten Lebensqualität und fast zu einer Verdoppelung der Lebenszeit seit 1900 verholfen. Ursprünglich aus Apotheken und Farbstoffchemie entstanden, hatte die Pharmaindustrie alleVoraussetzungen für interdisziplinäre Forschung an der Schnittstelle von Medizin, Biowissenschaften, Chemie und Gesundheitsfürsorge. Ihre spektakulären Erfolge machten sie zu einem zentralen Player im globalen Gesundheitssystem und zu einem der umsatzstärksten Industriezweige; aktuell wird der der globale Umsatz auf rund 1 200 Milliarden US $ (49,1 % davon in Nordamerika) geschätzt.
Die Entwicklung der Pharmasparte ist alledings nicht ohne größere Pannen und Katastrophen verlaufen und hat in Folge zu einem streng regulierten und kontrollierten Prozess der Arzneimittel-Forschung und Entwicklung geführt.
Versuchstiere werden in der Arzneimittelentwicklung vorgeschrieben
Bis in die 1930er Jahre brauchte man für den Kauf von Medikamenten meistens noch kein Rezept und die Formulierungen wurden zumeist in der Apotheke nach den Angaben in den Arzneibüchern (Pharmakopöen) zusammengemischt. Nur in wenigen Staaten gab es bereits Behörden für die Zulassung neuer Arzneimittel; diese hatten aber kaum Befugnisse, um übertriebene Werbung zu untersagen und in die Medikamentenentwicklung einzugreifen.
1937 änderte sich die Situation, als in den USA die Antibiotika-Bereitung Elixir-Sulfanilamid Massenvergiftungen hervorrief und mehr als hundert Menschen starben . Unter den Toten waren viele Kinder, die den nach Himbeeren schmeckenden aber das hochtoxische Lösungsmittel Diethylenglykol enthaltenden "Erkältungssaft" geschluckt hatten. In Folge erließ der US-Kongress 1938 Gesetze (Federal Food, Drug, and Cosmetic Act), welche die Qualität der pharmazeutischen Bereitungen garantieren sollten und positiv verlaufende Sicherheitstests an Tieren vorschrieben, bevor neue Medikamente auf den Markt kommen durften; darüber hinaus erhielt die Arzneimittelbehörde der Vereinigten Staaten (FDA) weitreichende Befugnisse in den Forschungs-und Entwicklungsprozess von Arzneimitteln einzugreifen. Andere Länder schlossen sich diesen Regulierungen an.
Noch immer gab es keine obligatorischen Untersuchungen zur Wirksamkeit eines neuen Medikaments. Dazu wurden die Pharmaunternehmen erst 1962, nach dem Contergan-Skandal verpflichtet. Das Medikament war zwischen 1957 und 1961 schwangeren Frauen als Beruhigungs- und Schlafmittel empfohlen worden und hatte bei vermutlich mehr als 10 000 Neugeborenen zu schweren Missbildungen von Gliedmaßen und Organen geführt. Das US-Gesetz von 1938 wurde nun erweitert: alle Arzneimittel mussten sowohl wirksam als auch (in vertretbarem Ausmaß) sicher sein; Zulassungsanträge an die FDA mussten die Wirksamkeit im Tierversuch nachweisen und alle bei den Tests aufgetretenen Nebenwirkungen offenlegen. Ein spezielles Programm untersuchte diesbezüglich auch alle vor 1962 zugelassenen Medikamente - von 3 443 Produkten haben sich fast ein Drittel (1051) als unwirksam erwiesen!
Seit den 1960er Jahren wurden die erforderlichen Protokolle für die präklinischen Wirksamkeits- und Toxizitätstest und für die folgenden klinischen Studien weiter entwickelt und haben zu einem immer länger (und damit teurer) werdenden und stärker regulierten Forschungs- und Entwicklungsprozess geführt. Der heute geltende, aus mehreren aufeinanderfolgenden Phasen zusammengesetzte Prozess soll hier kurz dargestellt werden:
Der Prozess beginnt im Labor mit der Forschung nach den möglichen Ursachen einer Erkrankung und der Suche nach Molekülen , mit denen diese Ursachen beeinflusst werden können. Gelingt es derartige Substanzen zu identifizieren und sind diese patentierbar, so kann die erfolgversprechendste davon in die präklinische Entwicklung eintreten und falls sie diese übersteht, zur Prüfung am Menschen in 3 Phasen zugelassen werden; in Phase 1 erfolgt die Prüfung an einer kleineren Gruppe (20 -100) junger, gesunder Freiwilliger, in Phase 2 sind bis zu mehreren Hundert Patienten involviert, in Phase 3 bis zu mehreren Tausend Patienten. Abbildung 1. Vom Beginn der präklinischen Entwicklung an dauert es im Mittel rund 10 Jahre bis für einen erfolgreichen Arzneimittel-Kandidaten die Unterlagen zur Registrierung eingereicht werden können (Abbildung 1).
| Abbildung 1. . Aktuelles Schema des Arzneimittel-Entwicklungsprozesses ab der Identifizierung eines patentierbaren Entwicklungskandidaten und Zeitdauer der präklinischen Phasen zur Prüfung von Toxizität und Wirksamkeit (Pharmakologie) und der 3 klinischen Phasen der Untersuchungen am Menschen. (Quelle: efpia: The Pharmaceutical Industry in Figures, Key data 2022. www.efpia.eu). |
Zur präklinischen Prüfung
der Wirksamkeit dienen Tiermodelle, in denen die zu behandelnde menschliche Krankheit nachgebildet wurde, d.i. ähnliche Symptome wie beim Erkrankten hervorgerufen werden (ein nicht immer erfolgversprechender Ansatz). Um eine Vorauswahl unter mehreren potentiellen Entwicklungskandidaten zu treffen, werden in verstärktem Ausmaß auch aus menschlichen Zellen erzeugte in vitro-Modelle eingesetzt.
der Sicherheit, d.i. fehlender/tolerierbarer Toxizität, sind Untersuchungen an jeweils zwei Spezies, einem Nager - meistens Ratte oder Maus - und einem Nichtnager - zumeist dem Hund - vorgeschrieben. Je nach Dauer der geplanten Anwendung einer neuen Substanz umfassen die Toxizitätstests Untersuchungen mit einmaliger (akute Toxizität) und wiederholter subchronischer und chronischer Verabreichung, weiters Reproduktionstests und Genotoxizitätstests bis hin zu lebenslangen (d.i. bei Ratten rund 2 Jahre dauernden) Cancerogenitätstests. Auch hier wird die Vorauswahl unter mehreren potentiellen Entwicklungskandidaten häufig an Hand von in vitro-Modellen getroffen.
Bei Verwendung statistisch ausreichend großer Gruppen in den verschiedenen Dosierungen kann die Anzahl der dazu erforderlichen männlichen und weiblichen Tiere bis auf über 3000 steigen. Dazu kommen noch Hunderte Tiere für Studien zur Pharmakokinetik (sogenannte ADME-Studien) - d.i. zur Quantifizierung der Aufnahme (A) eines Wirkstoffs in den Organismus, seiner Verteilung (D) und seinem Metabolismus (M) im Organismus und seiner Ausscheidung (E) - und zur Pharmakodynamik, d.i. zu den Effekten eines Wirkstoffs auf den geplanten Wirkort. Hinsichtlich pharmakokinetischer Eigenschaften haben in vitro Modelle aus menschlichen Zellen eine hohe Vorhersagekraft und können das Scheitern diesbezüglich ungeeigneter Verbindungen in der Klinik verhindern (siehe unten).
.. und der klinischen Prüfung
Wenngleich der Aufwand für Tierversuche und die damit verbundenen Kosten auch enorm hoch sind, ist es ein leider millionenfach bestätigtes Faktum, dass Versuchstiere zwar in ähnlicher, nicht jedoch in derselben Weise auf Wirkstoffe reagieren und damit umgehen wie wir Menschen. Wenn neue Wirkstoff-Kandidaten nach erfolgreicher Testung an Tieren in die klinische Prüfung am Menschen eintreten dürfen - d.i. rund 6 Jahre nach Beginn der Entwicklung , so scheitern dort bis zu 95 % - hauptsächlich wegen geringer/fehlender Wirksamkeit und/oder nicht tolerierbaren Nebenwirkungen. Die meisten fallen in der zweiten klinischen Phase durch, der Phase in der erstmals an Patienten geprüft wird - bis dahin sind 8 - 9 Jahre vergangen und rund ein Drittel der Gesamtkosten von bis zu mehreren Milliarden US $ angefallen.
Bis in die 90er-Jahre sind bis zu 40 % der Substanzen in der Klinik wegen unbefriedigenden pharmakokinetischen (ADME) Eigenschaften gescheitert: Viele oral verabreichte Arzneistoffe wurden aus dem Magen-Darmtrakt viel schlechter aufgenommen als aus dem Tierversuch zu erwarten war und erwiesen sich auf Grund der zu niedrigen Konzentrationen im Organismus dann als unwirksam. Viele Substanzen wurden im menschlichen Körper viel schneller/ viel langsamer abgebaut als im Tier und waren dann im ersten Fall unwirksam oder zeigten auf Grund zu hoher, langlebiger Konzentrationen im zweiten Fall schwerwiegende Nebenwirkungen. Mit der Verfügbarkeit von aus menschlichen Darmzellen bestehenden Resorptionsmodellen und aus menschlichen Leberzellen erzeugten Metabolismus-Modellen scheitern heute nur mehr wenige Prozent der Substanzen wegen ungünstigen ADME Eigenschaften.
Tierschützer wie auch einige Pharmaunternehmer sehen Tierversuche als eine Vergeudung von Leben, Kosten und Zeit und drängen darauf das veraltete System der wenig treffsicheren Tierversuche durch alternative humanbasierte Testverfahren zu ersetzen.
Reichen die derzeitig verfügbaren Moodelle aber dazu aus?
Die US-Arzneimittelbehörde FDA besteht nicht mehr auf Tierversuchen
Ende Dezember 2022 hat der amerikanische Präsident Biden den FDA Modernization Act 2.0 als Gesetz unterzeichnet. Mit diesem Gesetz, das auch den Reducing Animal Testing Act enthält, wird die überholte, aus den Jahren 1938 und 1962 stammende FDA-Vorschrift abgeschafft, wonach Arzneimittel-Kandidaten an Tieren getestet werden müssen, bevor sie in klinischen Studien am Menschen eingesetzt werden. In anderen Worten: Anträge zur Zulassung zu klinischen Studien können nun auf Basis tierversuchsfreier Verfahren akzeptiert werden.
Das Gesetz sieht allerdings kein vollständiges Verbot von Tierversuchen in der Entwicklung von Arzneimitteln vor; vielmehr ermöglicht es die Anwendung human-relevanter alternativer Methoden, sofern dies realisierbar ist. Als Beispiel dient hier ein von der Firma Hesperos Inc. (Florida) und Kollegen entwickeltes Neuropathie-Krankheitsmodell, das - aus induzierten pluripotenten Stammzellen und Schwannschen Zellen aufgebaut - in einem mikrofluiden System (siehe unten) eingesetzt wurde. In diesem Modell normalisierte die Behandlung mit speziellen Antikörpern (von Sanofi) die neuronale Funktion; die FDA akzeptierte dies als Nachweis der Wirksamkeit und gestattete, mit der klinischen Prüfung zu beginnen.
Auf der Humanbiologie basierende alternative Methoden
sind in den letzten 10 Jahren in der akademischen Forschung, in Biotech-Unternehmen und auch in behördlichen Forschungseinrichtungen in zunehmendem Maße entwickelt worden. Es sind dies zellbasierte in vitro Assays (Abbildung 2). Nach den 2-dimensionalen Zellkulturen, wie sie bereits seit Jahrzehnten angewandt werden, liegt nun der Fokus auf
- Organoiden - aus pluripotenten Stammzellen gezüchtete Gruppen unterschiedlicher Zelltypen eines Organs, die sich selbst zu 3-dimensionalen hohlen Mikrostrukturen organisieren, die dem Organ im Aufbau und Funktion ähneln (beispielsweise Mikrohirne) - und
- "Organ-on-a-Chip" - mikrophysiologischen Systemen, bei denen Organoid-ähnliche Kokulturen in mikrofluide Chips eingebettet sind.
Ausgangsmaterial dieser Assays sind aus verschiedenen Organen entnommene primäre menschliche Zellen und Biopsien, menschliche Zelllinien und adulte, embryonale und induzierte pluripotente Stammzellen.
| Abbildung 2. . Wesentliche Modelle in der Arzneimittelforschung und Entwicklung. Beschreibung: siehe Text. Abbildung modifiziert nach [1] Liancao Jiang et al., Bioengineering2022. https://www.mdpi.com/2306-5354/9/11/685 (Lizenz: cc-by) |
"Organ-on-a-Chip"
ist eine bereits fortgeschrittene Technologie mit einem ungeheuer breiten Spektrum an biologischen und biomedizinischen Anwendungsmöglichkeiten, die von Grundlagenforschung bis zu Personalisierter Medizin reichen [2].
Das Kernstück ist ein durchsichtiger, flexibler Chip aus einem Kunststoff auf Silikonbasis , der die Größe eines USB-Memory Sticks hat (Abbildung 2: ganz rechts) und ein Organ oder auch ein Organsystem nachahmen soll: Der Chip ist von hohlen mikrofluiden Kanälen durchzogen, die durch eine poröse Membran in zwei Kompartimente geteilt sind. Auf dieser Membran ordnen sich die Zellen eines bestimmten Organs organoid-ähnlich an, das angrenzende Kompartiment simuliert die funktionsbestimmende Umgebung des Organs - beispielsweise das Lumen des Darms. Das andere Kompartiment wird von Flüssigkeiten (Blutersatz) durchströmt; es kommen ihm - wie den Blutkapillaren im Gewebe - die mikrofluide Versorgung mit Nährstoffen und Entsorgung von Stoffwechselprodukten der Gewebezellen zu und es grenzt sich von diesen auch durch Endothelzellen ab.
Das durchsichtige Material ermöglicht morphologische Veränderungen kontinuierlich mikroskopisch zu verfolgen, in der ausströmenden Flüssigkeit können (biochemische) Indikatoren für diverse physiologische und pathologische Vorgänge (u.a. Wirkungsmechanismen und toxische Nebenwirkungen) gemessen werden.
Die Leber als zentrales Organ im Abbau von Fremdstoffen - u.a. den Medikamenten - ist häufig von deren toxischen Nebenwirkungen betroffen. Abbildung 3 illustriert das Design der "Leber auf dem Chip", das zu einem essentiellen Modell in der Pharmaentwicklung werden kann.
Eine kürzlich erschienene Untersuchung hat diesem Lebermodell ein hohes Potential für die korrekte Vorhersage von Leber-toxischen Substanzen bescheinigt. In einem verblindeten Set von 27 bekannten Leber-toxischen und untoxischen Verbindungen erkannten die Chips 87 % der toxischen Substanzen richtig und keine der untoxischen Substanzen als toxisch [3].
| Abbildung 3 . Schematische Darstellung eines "Leber auf dem Chip" Modells. Das Modell enthält alle wesentlichen Zelltypen der menschlichen Leber in der für das Organ charakteristischen Anordnung:. Die typischen Leberzellen (C; Hepatozyten) sitzen begrenzt von extrazellulärer Matrix (B) auf einer porösen Membran (D) innerhalb des oberen Kanals (A; blau). Der untere, dem Blutgefäß entsprechende (vaskuläre) Kanal (H; rot) wird von Endothelzellen ausgekleidet (G), die Fett und Vitamin A speichernden Sternzellen (E) sind im Raum zwischen Hepatozyten und Endothel angesiedelt, die Kupferzellen - Makrophagen oder Fresszellen - der Leber (F) im Gefäßraum. (Bild aus [1]: Lorna Ewart et al., Communications Medicine, 2022. https://doi.org/10.1038/s43856-022-00209-1 (Lizenz: cc-by) |
Das Tissue Chip for Drug Screening Programm
Initiative der NIH und FDA
Mit dem Ziel Sicherheit und Wirksamkeit von Arzneimittelkandidaten besser vorherzusagen zu können, haben die US-National Institutes of Health (NIH) gemeinsam mit der FDA bereits 2012 das Programm "Tissue Chip for Drug Screening" (Gewebechips für das Arzneistoffscreening) ins Leben gerufen [4]. Die Förderungen galten vorerst der Entwicklung von 3-D-Chips aus menschlichem Gewebe, die die Struktur und Funktion menschlicher Organe genau nachahmten.
2014 schlossen sich Forscher zusammen, um einzelne "Organs-on-a-Chip" zu einem Multiorgan Modell zu entwickeln, das nun auch die Wechselwirkungen zwischen den Organen berücksichtigen sollte (beispielsweise wurden Pharmakokinetik-Modelle entwickelt, die alle Aspekte von Aufnahme, Verteilung, Abbau und Eliminierung eine Arzneimittels erfassen konnten) .2016/2017 begannen die NIH Projekte zu fördern, die Chips zur Modellierung von Krankheiten und Testung der Wirksamkeit von Behandlungen designten. Es folgte die Förderung von Modellen für Schmerz und Opioidabhängigkeit und von Ansätzen zur Modellierung des Immunsystems.
Schlussendlich sollen alle wesentlichen Organe zu einem "Human-Body-on- a Chip" also einem menschlichen Avatar vernetzt werden. Abbildung 4.
| Abbildung 4. . Zukunftsmusik: Wesentliche "Organs-on-a-Chip" werden zu einem System zusammengeschlossen, das den "Human Body on a Chip simuliert.(Bild: NIH, National Center for Advancing Translational Sciences; Lizenz: gemeinfrei). |
Das Budget der FDA unterstützt ein FDA-weites Programm, um diese alternativen Methoden zu evaluieren. Die Forscher wollen damit einerseits die humanspezifische Physiologie von Geweben oder Organen modellieren, anderseits Methoden für regulatorische Zwecke vorantreiben, welche die Effizienz der Arzneimittelentwicklung erhöhen und Tierversuche ersetzen, reduzieren und verbessern ("replace, reduce, refine") können.
Können die derzeitigen alternativen Methoden bereits Tierversuche ersetzen?
Ein beträchtlicher Teil der bis jetzt entwickelten Modelle kann sicherlich in frühen Entwicklungsphase von Arzneistoffen zur sogenannten Leadoptimierung eingesetzt werden, d.i. wenn aus mehreren möglichen Kandidaten die erfolgversprechendste Verbindung in puncto Sicherheit und möglicherweise auch Wirksamkeit ausgewählt werden soll.
Das Lebermodell kann frühzeitig Lebertoxizität feststellen, das Multiorgan-Modell der Pharmakokinetik erlaubt konkrete Prognosen über Verfügbarkeit, Spiegel , Metabolisierung und Verweildauer neuer Substanzen im Organismus. Das reduziert die Entwicklungszeit, die Zahl der Versuchstiere und die Kosten.
Allerdings spiegeln die bis jetzt entwickelten alternativen Modelle die komplexe pharmakologische Situation unseres Körpers nur bedingt wider. Toxizitäten können auch in Organen auftreten, die von den Testsystemen nicht erfasst werden, die Rolle des Immunsystems wird in den Modellen noch unbefriedigend abgebildet. Am Kunststoff-Material der Chips können Substanzen hängen bleiben (adsorbiert werden) und damit niedrigere Konzentrationen (und damit geringere Nebenwirkungen) vortäuschen. Ein ganz wesentliches Problem der Multi-Organ Chips und damit schlussendlich des Body-on-a-Chip - eines menschlichen Avatars - ist das Skalieren der einzelnen Komponenten, d.i. der korrekten Verweilzeit der durchströmenden Flüssigkeit und damit der Testsubstanzen in den Organmodellen. Natürlich ist es auch entscheidend wie weit die Funktionsfähigkeit in den Zellen über die Versuchsdauer erhalten bleibt. Noch zahlreiche weitere Fragen sind offen.
Viele der bestehenden Probleme erscheinen lösbar und rücken uns dem Ziel näher immer verlässlichere Prognosen über die Auswirkungen von Fremdstoffen - darunter Medikamenten - auf den menschlichen Organismus zu erhalten, als es jetzt mit Versuchstieren der Fall ist.
Trotz der begrenzten Übertragbarkeit von Ergebnissen im Tierversuch auf den Menschen, dürften die Tierstudien in der präklinischen Entwicklungsphase noch nicht durch Alternativen ersetzbar sein. Die europäische Arzneimittelbehörde EMA hat eine Arbeitsgruppe eingerichtet, die die neuen Verfahren erst einmal evaluieren soll und sieht den Zeitpunkt für den Ersatz der Tierversuche noch nicht gekommen.
[1] Liancao Jiang et al., Bioengineering 2022. https://www.mdpi.com/2306-5354/9/11/685
[2] Ingber, D.E. Human organs-on-chips for disease modelling, drug development and personalized medicine. Nat Rev Genet 23, 467–491 (2022). https://doi.org/10.1038/s41576-022-00466-9
[3] Lorna Ewart et al., Performance assessment and economic analysis of a human Liver-Chip for predictive toxicology. Communications Medicine, 2022. https://doi.org/10.1038/s43856-022-00209-1
[4] Tissue Chip Initiatives & Projects:https://ncats.nih.gov/tissuechip/projects
Zum Arzneimittelforschungs-und Entwicklungsprozess im ScienceBlog
- Inge Schuster; 24.02.2019: Pharma im Umbruch
- Inge Schuster; 15.04.2016: Big Pharma - ist die Krise schon vorbei?
- Redaktion; 24.04.2015: Wie entstehen neue Medikamente? Pharmazeutische Wissenschaften
- Christian R. Noe; 01.01.2015: Neue Wege für neue Ideen – die „Innovative Medicines Initiative“(IMI)
- Peter Seeberger; 16.05.2014: Rezept für neue Medikamente
- Inge Schuster; 08.03.2012: Zur Krise der Pharmazeutischen Industrie
Was da kreucht und fleucht - Wie viele Gliederfüßer (Arthropoden) leben im und über dem Boden und wie hoch ist ihre globale Biomasse?
Was da kreucht und fleucht - Wie viele Gliederfüßer (Arthropoden) leben im und über dem Boden und wie hoch ist ihre globale Biomasse?Sa, 18.02.2023 — Redaktion

![]() Weltweit ist ein offensichtlich vom Menschen verursachter, besorgniserregender Rückgang von Arthropoden - und hier sind vorerst vor allem Insekten im Fokus - zu beobachten. Viele Fragen zu den, für den Erhalt unserer Ökosysteme unabdingbaren Arthropodenarten sind bislang noch ungeklärt, insbesondere was das Ausmaß der globalen Populationen und deren Zusammensetzung in verschiedenen Lebensräumen und Ökosystemen betrifft. Eine neue Studie aus der Gruppe von Ron Milo (Weizmann Institut, Israel) ermöglicht nun erstmals einen umfassenden, quantitativen Blick auf die Populationen der Arthropoden. Die neuen Befunde bieten eine wertvolle Grundlage zur Abschätzung, wie sich unsere ökologischen Systeme verändern und wie sich dies auf unsere Ökonomie, Ökologie und Lebensqualität auswirken könnte.
Weltweit ist ein offensichtlich vom Menschen verursachter, besorgniserregender Rückgang von Arthropoden - und hier sind vorerst vor allem Insekten im Fokus - zu beobachten. Viele Fragen zu den, für den Erhalt unserer Ökosysteme unabdingbaren Arthropodenarten sind bislang noch ungeklärt, insbesondere was das Ausmaß der globalen Populationen und deren Zusammensetzung in verschiedenen Lebensräumen und Ökosystemen betrifft. Eine neue Studie aus der Gruppe von Ron Milo (Weizmann Institut, Israel) ermöglicht nun erstmals einen umfassenden, quantitativen Blick auf die Populationen der Arthropoden. Die neuen Befunde bieten eine wertvolle Grundlage zur Abschätzung, wie sich unsere ökologischen Systeme verändern und wie sich dies auf unsere Ökonomie, Ökologie und Lebensqualität auswirken könnte.
Die zunehmende Dominanz des Menschen über den Erdball hat zu massiven ökologischen und geologischen Auswirkungen geführt. Das Artensterben hat sich im Anthropozän um Größenordnungen über die durch natürliche Evolutionsprozesse hervorgerufenen Veränderungen beschleunigt und die Populationen diverser Wirbeltierarten nehmen ab. Wie Untersuchungen aus jüngster Zeit zeigen, gehen auch die im Boden und auf/über dem Boden lebenden Populationen von Gliederfüßern (Arthropoden) - Insekten, Spinnen, Milben, Tausendfüßler und andere Vertreter der Taxa - deutlich zurück. Für den Rückgang werden menschliche Aktivitäten verantwortlich gemacht, welche die Zerstörung von Lebensräumen, die Intensivierung der Landwirtschaft, den verstärkten Einsatz von Pestiziden und das Einschleppen invasiver Arten miteinschließen. Dazu kommen die Folgeerscheinungen des anthropogen verursachten Klimawandels - ein hoher Stickstoffeintrag in der Atmosphäre, Hitzeperioden, Dürren, Waldbrände und veränderte Niederschlagsmuster.
Der Rückgang der Arthropoden
ist besorgniserregend, da ihnen eine zentrale Rolle in den terrestrischen Ökosystemen zukommt: sie sind ein enorm wichtiger Bestandteil vieler Nahrungsnetze und dienen diversen Tierarten - von Vögeln, Reptilien, Amphibien bis zum Vielfraß - als Nahrung und ernähren sich selbst von Pflanzen und Tieren. Arthropoden bestäuben unsere Pflanzen, verbreiten deren Samen und bauen unsere Böden auf, indem sie pflanzliche und tierische Abfälle abbauen/kompostieren.
Trotz ihrer ungeheuren ökologischen Bedeutung hat es bis jetzt kaum Informationen über die globale Verteilung der landlebenden Arthropoden gegeben. Die meisten bisherigen Studien beruhen auf Stichproben, die Trends in der Population oder Biomasse in einem Gebiet gemessen haben, aber nicht den gesamten dortigen Bestand quantifizieren konnten. Die Quantifizierung der Arthropoden in verschiedenen Lebensräumen und an verschiedenen Orten ist aber essentiell, um eine Basis zu schaffen, anhand derer künftige Veränderungen der Populationen gemessen werden können, wie sich diese auf globale Prozesse auswirken und welche Erfolge durch Maßnahmen erzielt werden könnten.
Eine erste Studie zur umfassenden Quantifizierung
In einer neuen Studie haben Forscher um den Biophysiker Ron Milo vom Weizmann Institut nun erstmals ein quantitatives Bild der Mengen an terrestrischen Arthropoden, ihrer Zusammensetzung und ihrer globalen Biomasse erzeugt [1]. Milo und sein Team sind weltweit anerkannt für ihre Studien zur Bestimmung der globalen Populationen und Biomassen verschiedener Artengruppen; ihre Arbeiten sind Tausende Male zitiert. Mit dem Ziel einen ganzheitlichen Überblick über die Zusammensetzung der Biosphäre zu ermöglichen, hat Ron Milo zusammen mit Rob Philipps (Professor am CalTech, USA) vor einigen Jahren eine Bionumbers Database (https://bionumbers.hms.harvard.edu/) entwickelt. Ebenfalls von Forscherteams um Ron Milo und Rob Philipps wurde kürzlich eine neue, separate Datenbank - die Human Impacts Database (www.anthroponumbers.org) - vorgestellt, welche die Auswirkungen menschlicher Aktivitäten auf Ökologie und Geologie unseres Planeten zum Thema hat. Über diese und frühere Aktivitäten wurde im ScienceBlog berichtet [2 - 4].
Um auf die gegenwärtige Studie über Arthropoden zurückzukommen, die sich zu einem gigantischen Projekt ausgewachsen hat: Die Forscher haben die Literatur nach Messungen der absoluten Populationen (Zahl der Individuen pro Fläche) und Biomassedichten (= Biomasse pro Fläche) von Arthropoden durchsucht und etwa 7000 solcher Auswertungen gesammelt, die weltweit an rund 500 Standorten und oft über viele Jahre hinweg erfolgt waren.
| Abbildung 1. Globale Standorte (A) der Probennahme - unterirdische oder oberirdische Proben - und Untersuchungszeitraum (B) . (Bild leicht modifiziert nach Fig 1.in Rosenberg et al., 2023 [1]. Lizenz: cc-by) |
Diese Standorte haben alle wichtigen Biome umfasst - von Regenwäldern bis hin zu borealen Wäldern, Grünland in tropischen und gemäßigten Zonen, Weiden, Ackerland, Tundra und Wüsten. An insgesamt etwa 440 Stellen wurden Proben des Lebensraums "Boden & abgestorbenes Pflanzenmaterial (Pflanzenstreu)" analysiert, an rund 60 Stellen Proben der oberirdischen Habitate "Bodenoberflächen und Pflanzen". Abbildung 1 gibt einen Eindruck von der globalen Verteilung der Sammelstellen mit den dort geprüften Habitaten - im Boden oder oberirdisch - und den Zeitraum der Untersuchungen.
Arthropoden sind ein riesengroßer, aus über einer Million Arten bestehender Stamm des Tierreichs. Um die an den verschiedenen Standorten erhaltenen Biomassedichten und Populationszahlen vergleichen und global integrieren zu können, wurden die landbewohnenden Arten auf Basis der Taxonomie in große Gruppen eingeteilt; für die im Boden lebenden Arthropoden sind diese Gruppen - unterschiedlich gefärbt - in Abbildung 2 dargestellt.
| Abbildung 2. Phylogenetischer Baum der im Boden lebenden Arthropoden. Jede Farbe steht für eine einzelne zusammengefasste taxonomische Gruppe. Die oberirdischen Arthropoden wurden separat analysiert.(Bild leicht modifiziert nach Fig 1.in Rosenberg et al., 2023 [1]. Lizenz: cc-by)., |
Die Analyse
der gesammelten 7000 Daten zeigt, dass der größte Teil der Biomasse der terrestrischen Arthropoden auf Spezies zurückzuführen ist, die im Boden leben (Abbildung 3, oben). Die gesamte globale Biomasse dieser unterirdischen Tiere wird auf rund 200 Millionen Tonnen (200 Mt) Trockengewicht geschätzt, die der oberirdisch lebenden Tiere auf rund 100 Mt. Insgesamt kann man von einem gesamten globalen Trockengewicht von 300 Mt Tieren ausgehen, entsprechend einem Lebendgewicht von etwa 1000 Mt. Umgerechnet mit den Lebendgewichten der einzelnen Arten ergibt dies eine Zahl von 1 x 1019 - 10 000 000 Billionen - Individuen (Unsicherheitsbereich: 0,5 x 1019- 2,0 x 1019).
|
Abbildung 3. Globale Biomasse der terrestrischen Arthropoden (oben) und taxonomische Gliederung der gesamten Biomasse der im Boden lebenden Arten (unten, links) und relative Biomasse der in Abbildung 2 aufgezeigten Unterstämme (unten, rechts). Die Fehlerbalken im Bild oben markieren den Unsicherheitsbereich, der sich aus der Summe der unteren und oberen Grenzen aller 95 %-Konfidenzintervalle auf Ebene aller untersuchten Lebensräume ergibt (dazu: Abbildung 4) . Das gestrichelte grüne Rechteck ist die zusätzliche Schätzung der oberen Grenze für die oberirdischen Arthropoden. (Bild modifiziert aus Figs 4 und 5 in: Rosenberg et al., 2023 [1]. Lizenz: cc-by). |
Bodenarthropoden
Von den im Boden lebenden Arten sind rund 40 % Termiten; auf Ameisen, Springschwänze und Milben fallen jeweils um die 10 %, (Abbildung 3, unten links), der Rest auf andere Spezies (Abbildung 3, unten rechts). Diese unterirdisch lebenden Tiere, insbesondere die winzigen Springschwänze und Milben, sind für Prozesse verantwortlich, die den Boden aufbauen, düngen und den globalen Kohlenstoffkreislauf beeinflussen und damit für Ökologie des Bodens von entscheidender Bedeutung sind.
Die Biomassen-Dichte (Trockengewicht) der Bodenarthropoden variiert in den einzelnen Habitaten und in der Zusammensetzung. Abbildung 4. Höchste Dichten von etwa 3 g/m2 - Termiten tragen am meisten zur Biomasse bei - gibt es in den tropischen und subtropischen Waldgebieten, niedrigste Werte bis unter 0,1 g/m2 in Wüsten und trockenem Buschland. Auf einzelne Individuen bezogen (beispielsweise wiegt eine Boden-Ameise im globalen Mittel rund 0,84 mg) dominieren in allen Habitaten die kleinen Milben und Springschwänze: ihre Dichte reicht von bis zu 200 000 Tieren pro m2 in den borealen Wäldern bis zu etwa 1000 Tieren pro m2 in Wüsten und trockenem Buschland.
Bemerkenswert dabei: das vom Menschen bearbeitete Ackerland weist eine wesentlich niedrigere Populationsdichte auf als Wälder und Grasland in vergleichbaren Zonen.
| Abbildung 4. Abbildung 4. Biomassendichte der Bodenarthropoden in den wesentlichen globalen Lebensräumen. Die Dichte der einzelnen farbig markierten Tiergruppen ist in logarithmischem Maßstab aufgetragen, die Farbstärke spiegelt die Zahl der Untersuchungen an der Meßstelle wider. Die globale Fläche der einzelnen Habitate ist in Klammern in Millionen km2 angegeben. (Bild modifiziert aus Fig 2 in: Rosenberg et al., 2023 [1]. Lizenz: cc-by). |
Oberirdische Arthropoden
Hier gibt es insgesamt bedeutend weniger Untersuchungen in den wesentlichen Lebensräumen (Abbildung 1) und die angewandten Methoden - Vernebelung von Baumkronen und Leeren von Fallen - können wichtige Entwicklungsformen - beispielweise Raupen - unterrepräsentieren. Studien haben in allen Waldarten stattgefunden und ein Drittel der auf Bäumen lebenden Tiere auf Ameisen zurückgeführt. In den tropischen Wäldern dürfte wohl der Großteil der Biomasse zu finden sein, allerdings basiert diese Aussage auf nur zwei Studien, die Proben der Waldbodenfläche und ganzer Baumgemeinschaften untersucht haben.
Da die oberirdischen Arthropoden - Schmetterlinge, Ameisen, Käfer, Heuschrecken und Spinnen - in der Regel viel größer als die hauptsächlich im Boden lebenden Milben und Springschwänze sind, ist ihre auf Basis der Biomassendichte geschätzte Populationsdichte und damit ihr Beitrag zur globalen Populationsdichte viel niedriger als die der Bodenanthropoden.
Fazit
Die Kenntnis der Zusammensetzung der Biosphäre ist von grundlegender Wichtigkeit, um wesentliche Veränderungen verfolgen, verstehen und mögliche Maßnahmen dagegen ergreifen zu können. Mit der Quantifizierung der terrestrischen Arthropoden über alle Lebensbereiche hat das Team um Ron Milo begonnen eine wichtige Lücke zu schließen. Laut Schätzungen der Forscher gibt es auf der Erde rund 10 Millionen Billionen terrestrische Arthropoden mit einer gesamten Biomasse von 300 Millionen Tonnen.
Wie viel ist das in Relation zu anderen Lebewesen und was bedeutet es?
Die Forscher geben die Antwort: Es ist dies eine Biomasse, die mit der von allen Menschen und ihren Nutztieren (400 Millionen Tonnen) vergleichbar und um eine Größenordnung höher als die der Wildtiere ist. Die Biomasse der Regenwürmer, Nematoden und Enchyträen ist etwa gleich hoch, die der Meeres-Arthropoden (dominiert von Krebstieren) um eine Größenordnung, die der Mikroorganismen im Boden um 2 Größenordnungen höher. Trotz der im Vergleich zu den Mikroorganismen viel niedrigeren Biomasse, tragen Boden-Arthropoden wesentlich zur Zersetzung von Pflanzenmaterial, zum Aufbau des Bodens und zur Homöostase der Kohlenstoffbilanz bei.
[1] Rosenberg et al., The global biomass and number of terrestrial arthropods. Sci. Adv. 9, eabq4049 (3 February 2023), https://www.science.org/doi/10.1126/sciadv.abq4049
[2] Redaktion, 22.12.2016: Kenne Dich selbst - aus wie vielen und welchen Körperzellen und Mikroben besteht unser Organismus?
[3] Redaktion, 29.12.2016: Wie groß, wie viel, wie stark, wie schnell,… ? Auf dem Weg zu einer quantitativen Biologie
[4] Redaktion, 10.02.2023: "Macht Euch die Erde untertan" - die Human Impacts Database quantifiziert die Folgen
"Macht Euch die Erde untertan" - die Human Impacts Database quantifiziert die Folgen
"Macht Euch die Erde untertan" - die Human Impacts Database quantifiziert die FolgenFr, 10.02.2023 — Redaktion

![]() Zu den verschiedenen anthropogenen Auswirkungen auf unserer Erde gibt es nun eine kuratierte, durchsuchbare Datenbank: die Human Impacts Database (HID) - www.anthroponumbers.org. Die HID enthält quantitative Daten (Werte und Zeitreihen von Werten) zu Schlüsselthemen, die ein möglichst umfassendes Bild menschlicher Aktivitäten und deren Folgen geben – vom Anstieg des Meeresspiegels über Viehbestände, Treibhausgasemissionen, den Einsatz von Düngemitteln zum Energiesektor und darüber hinaus. Damit bietet die HID eine einzigartige Ressource für Experten, politische Entscheidungsträger und die breite Öffentlichkeit, die verschiedenen Auswirkungen des Menschen auf den Planeten in ihren Ausmaßen und ihrer Vernetztheit besser zu verstehen.
Zu den verschiedenen anthropogenen Auswirkungen auf unserer Erde gibt es nun eine kuratierte, durchsuchbare Datenbank: die Human Impacts Database (HID) - www.anthroponumbers.org. Die HID enthält quantitative Daten (Werte und Zeitreihen von Werten) zu Schlüsselthemen, die ein möglichst umfassendes Bild menschlicher Aktivitäten und deren Folgen geben – vom Anstieg des Meeresspiegels über Viehbestände, Treibhausgasemissionen, den Einsatz von Düngemitteln zum Energiesektor und darüber hinaus. Damit bietet die HID eine einzigartige Ressource für Experten, politische Entscheidungsträger und die breite Öffentlichkeit, die verschiedenen Auswirkungen des Menschen auf den Planeten in ihren Ausmaßen und ihrer Vernetztheit besser zu verstehen.
In den letzten 10 000 Jahren hat der Mensch die Erde massiv verändert. Seine Aktivitäten in Landwirtschaft, Forstwirtschaft, Bergbau und Industrie haben in komplexer Weise Atmosphäre, Ozeane, Biosphäre und Geochemie der Erde beeinflusst. Die Auswirkungen auf den Planeten sind enorm vielfältig, betreffen nahezu jeden Aspekt des Erdsystems und jede wissenschaftliche Disziplin und werden nun mehr und mehr spürbar. Dennoch sind viele Menschen der Meinung, dass die Erde zu groß ist, um durch menschliche Aktivitäten nachhaltig geschädigt zu werden.
Bis jetzt gibt es nahezu unzählige Studien, die versucht haben den einen oder anderen Aspekt der Auswirkungen des Menschen auf den Planeten (beispielsweise die Treibhausgasemissionen oder den Energieverbrauch) zu quantifizieren. Da Wissenschaftler aus diversen Fächern auf Grund ihrer unterschiedlichen Ausbildung verschiedene Mess- und Analysemethoden anwenden, Daten in unterschiedlichen Einheiten und Formaten melden und verschiedenartige Definitionen verwenden, kann es für Forscher, politische Entscheidungsträger und die Öffentlichkeit sehr schwierig sein, diese Studien zu verstehen und miteinander in Beziehung zu setzen.
Die Human Impacts Database
Um eine Ressource zu schaffen, in der jeder Interessierte die vielfältigen Auswirkungen auf den Planeten schnell und einfach nachsehen, miteinander verflechten und quantifizieren kann, haben Forscherteams um die Biophysiker Rob Philipps, Professor am CalTech (USA) und Ron Milo, Professor am Weizmann Institut (Rehovot, Israel) die Datenbank Human Impacts Database - Anthroponumbers.org - entwickelt [1].
Beim Aufbau der Human Impacts Database haben sich die Forscher von den Erfahrungen mit dem Aufbau und der Nutzung Website BioNumbers Database - - https://bionumbers.hms.harvard.edu/ - leiten lassen, die Milo und Philipps einige Jahre zuvor herausgebracht hatten. Diese Datenbank enthält quantitative Daten zu verschiedenen Aspekten der (Molekular)Biologie – von Zellgröße zu Konzentration von Stoffwechselprodukten, von Reaktionsgeschwindigkeiten zu Generationszeiten, von Genomgröße zur Zahl der Mitochondrien in der Zelle – und hat sich zu einer weithin genutzten Ressource entwickelt (auch die diesbezüglichen Berichte im ScienceBlog [2],[3] wurden bereits viele Tausend mal aufgerufen ). Heute dient die BioNumbers Database Forschern zum Auffinden biologischer Kennzahlen, relevanter Primärliteratur, zum Erlernen von Messmethoden und zum Unterrichten grundlegender Konzepte in der Zellbiologie.
Die Zahlen
"Meiner Meinung nach liegt der Ursprung des Verstehens im Zahlenverständnis: Sobald man die Zahlen kennt, wird klar, was die Probleme sind, welche Dinge wichtig sind und welche weniger wichtig sind", sagt Philipps.
Die neue Human Impacts Database enthält momentan 307 eindeutige, manuell kuratierte Einträge, die ein breites Spektrum an Datenquellen abdecken, wissenschaftliche Primärliteratur, Regierungs- und NGO-Berichte und Industriemitteilungen einschließen. Bevor ein Eintrag in die Datenbank aufgenommen und veröffentlicht wird, wird er von den Administratoren (Experten in den einzelnen Disziplinen) eingehend geprüft (zum Kuratierungsverfahren: siehe Note S1 [1]). Was derzeit an Einträgen vorhanden ist, sind Schlüsseldaten, die ein erstes quantitatives Bild für die globalen Auswirkungen des Menschen auf die Erde vermitteln können; mit dem Fortschreiten relevanter Forschungsarbeiten, wird die Datenbank weiter wachsen und zu einem immer besseren Verstehen der komplexen Zusammenhänge und zu zielgerichteteren Maßnahmen führen.
Die Daten sind in fünf Hauptkategorien unterteilt: "Land", "Wasser", "Energie", "Flora & Fauna" und "atmosphärische & biogeochemische Zyklen". Da diese Kategorien sehr weit gefasst sind, und Einträge mehreren Kategorien zugeordnet werden können, gibt es dazu auch 20 Unterkategorien, wie beispielsweise "Landwirtschaft" oder "Kohlendioxid". Soweit verfügbar, enthält die Datenbank auch Zeitreihen, um zu veranschaulichen, wie sich diese Zahlen im Laufe der Jahre verändert haben.
| Abbildung 1. CO2-Konzentration in der Atmosphäre. Der Eintrag mit der HuID-Nummer 81043 findet sich in der Kategorie "atmosphärische & biogeochemische Zyklen" und Subkategorie CO2 (Quelle: Figure 1 aus Griffin Chure et al., (2022),[1]. Lizenz cc-by-nc-sa) |
Als Beispiel für einen solchen Eintrag ist die wohl bekannteste anthropogene Auswirkung auf den Planeten, die steigende CO2-Konzentration in der Atmosphäre, in Abbildung 1 aufgezeigt.
Der Eintrag findet sich in der in der Kategorie "atmosphärische & biogeochemische Zyklen", Subkategorie "CO2" (B) und ist mit einer fünfstelligen Zahl, "Human Impacts Database identifier"- HuID gekennzeichnet, die auch zitiert werden kann (D). Die aktuelle (d.i. Ende 2021) CO2-Konzentration von ungefähr 415 ppm (C) kann auch in anderen Messgrößen eingesehen werden. Die Zeitreihe im Zeitraum 1964 - 2021 (C) ist graphisch dargestellt (I), die Werte zu einzelnen Zeitpunkten sind interaktiv abrufbar. Es folgen eine Kurzbeschreibung des Inhalts (F) und der angewandten Methoden, einschließlich der Qualität der Messungen (G) und der Link zur Datenquelle ( H). Last, but not least gibt es auch die Information zur Lizenz der Originaldaten (K) und zum Administrator (L).
Von diesen Daten ausgehend kann man in weiterer Folge Einträge zu den CO2-verursachten steigenden Oberflächentemperaturen aufrufen, von hier u.a. Daten zu Gletscherschmelze und Ansteigen der Meeresspiegel, zum CO2-bedingten Absinken des pH-Wertsder Meere usw, usf. In kurzer Zeit erhält man so ein quantitatives Bild von dem Ausmaß der Treibhausgas-verursachten Auswirkungen beispielsweise auf die Meere.
Die Zahlen bieten die Grundlage, um Zusammenhänge in grafischer Darstellung besser erläutern zu können. Ein einfaches Beispiel ist in Abbildung 2 gezeigt.
| Abbildung 2. Wachstum der Weltbevölkerung und deren regionale Verteilung, unterschieden nach städtischen und ländlichen Wohnorten. (Quelle: Zeitreihe und Grafik zusammengestellt aus Griffin Chure et al., (2022),[1]. Lizenz cc-by-nc-sa) |
Fazit
Mit der Human Impacts Database ist ein großer Wurf gelungen!
Erstmals kann man nun die komplexen vielfältigen Wechselwirkungen zwischen dem Menschen und dem Land, den Ozeanen und der Atmosphäre in quantitativer Weise untersuchen und - auch, wenn die Einträge noch weiter wachsen werden - bereits jetzt besser verstehen. Abbildung 3 gibt einen groben Überblick über das Ausmaß dieser Auswirkungen. Das schnelle, einfache Auffinden relevanter Informationen hat ein enormes Suchtpotential zur Folge - man kann viele Stunden damit verbringen, um über immer neue Zusammenhänge zu spekulieren.
Die Datenbank bietet für Jeden etwas. Forscher haben leichten Zugriff auf quantitative Daten aus seriösen Quellen und den Link zur Primärliteratur. Lehrer und Schüler gewinnen ein besseres Verständnis für das Ausmaß und die komplexen Zusammenhänge der anthropogenen Auswirkungen auf unseren Planeten - entsprechende Themen könnten in den Unterricht aufgenommen werden und einseitigem Aktivismus vorbeugen. Interessierte Laien haben ein Nachschlagwerk, in dem die Informationen auf aktuellem Stand sind und sie diesen (zum Unterschied zu den online Plattformen) voll vertrauen können. Politische Entscheidungsträger schließlich können auf Basis der konkreten Daten Ansätze zur Behebung der humanen Auswirkungen andenken anstatt der üblichen allgemeinen Absichtserklärungen.
| Abbildung 3. Menschliche Auswirkungen auf den Planeten und ihre relevanten Ausmaße. Physikalische Einheiten und im täglichen Leben übliche Größen sind links oben dargestellt, die Kategorien oben in der Mitte. (Quelle: Figure 2 aus Griffin Chure et al., (2022),[1]. Lizenz cc-by-nc-sa) |
--------------------------------------------
[1] Griffin Chure, Rachel A. Banks, Avi I. Flamholz, Nicholas S. Sarai, Mason Kamb, Ignacio Lopez-Gomez, Yinon Bar-On, Ron Milo, Rob Phillips. Anthroponumbers.org: A quantitative database of human impacts on Planet Earth. Patterns, 2022; 100552, DOI: 10.1016/j.patter.2022.100552
[2] Redaktion, 22.12.2016: Kenne Dich selbst - aus wie vielen und welchen Körperzellen und Mikroben besteht unser Organismus?
[3] Redaktion, 29.12.2016: Wie groß, wie viel, wie stark, wie schnell,… ? Auf dem Weg zu einer quantitativen Biologie
Enorme weltweite Bildungsdefizite - alarmierende Zahlen auch in Europa
Enorme weltweite Bildungsdefizite - alarmierende Zahlen auch in EuropaSa, 04.02.2023— Inge Schuster

![]() Basierend auf den Daten aus internationalen und regionalen Leistungstests hat das Münchner ifo Zentrum für Bildungsökonomik kürzlich die bislang umfassendste Darstellung der Bildungsleistungen von Jugendlichen aus 159 Ländern (entsprechend 98 % der Weltbevölkerung) erarbeitet. Die Ergebnisse sind erschreckend: weltweit erreichen zwei Drittel der Jugendlichen keine grundlegenden Fähigkeiten, wie sie für die Teilhabe an modernen wettbewerbsfähigen und erfolgreichen Volkswirtschaften erforderlich sind. Das von allen Mitgliedsstaaten der UNO vereinbarte Entwicklungsziel SDG4 bis 2030 "sicher zu stellen, dass alle Kinder der Welt eine kostenlose, hochwertige Grund- und Sekundarbildung erhalten, die zu brauchbaren Lernergebnissen führt" ist in fernere Zukunft gerückt.
Basierend auf den Daten aus internationalen und regionalen Leistungstests hat das Münchner ifo Zentrum für Bildungsökonomik kürzlich die bislang umfassendste Darstellung der Bildungsleistungen von Jugendlichen aus 159 Ländern (entsprechend 98 % der Weltbevölkerung) erarbeitet. Die Ergebnisse sind erschreckend: weltweit erreichen zwei Drittel der Jugendlichen keine grundlegenden Fähigkeiten, wie sie für die Teilhabe an modernen wettbewerbsfähigen und erfolgreichen Volkswirtschaften erforderlich sind. Das von allen Mitgliedsstaaten der UNO vereinbarte Entwicklungsziel SDG4 bis 2030 "sicher zu stellen, dass alle Kinder der Welt eine kostenlose, hochwertige Grund- und Sekundarbildung erhalten, die zu brauchbaren Lernergebnissen führt" ist in fernere Zukunft gerückt.
Transformation unserer Welt: Die Agenda 2030 für nachhaltige Entwicklung
Mit dem Ziel weltweit eine nachhaltige Entwicklung auf ökonomischer, sozialer und ökologischer Ebene zu sichern und damit ein gutes, erfülltes Leben für alle Menschen zu gewährleisten, hat die Generalversammlung der Vereinten Nationen im September 2015 die Agenda 2030 für Nachhaltige Entwicklung verabschiedet [1]. Diese enthält 17 nachhaltige Entwicklungsziele (Sustainable Development Goals, SDGs), die vom Beenden von Armut und Hunger zu menschenwürdiger Arbeit, Wirtschaftswachstum und Schutz des Planeten vor Schädigungen reichen. Alle 193 Mitgliedstaaten haben sich verpflichtet auf nationaler, regionaler und internationaler Ebene auf die Umsetzung dieser Ziele bis zum Jahr 2030 hinzuarbeiten.
Ein sehr wichtiges Ziel ist die Gewährleistung einer hochwertigen, gerechten und inklusiven Bildung für alle (SDG4), das bedeutet: "frühkindliche, Grund-, Sekundar- und Hochschulbildung sowie Fach- und Berufsausbildung. ... damit alle sich das Wissen und die Fertigkeiten aneignen können, die sie benötigen, um Chancen zu nutzen und uneingeschränkt an der Gesellschaft teilhaben zu können"[1]. Die Teilhabe an modernen wettbewerbsfähigen, erfolgreichen Volkswirtschaften ist ihrerseits Voraussetzung zur Erreichung anderer SDGs - vor allem der Armut und dem Hunger ein Ende zu setzen.
Bis 2030 soll laut SDG4.1 sichergestellt sein," dass alle Mädchen und Jungen gleichberechtigt eine kostenlose und hochwertige Grund- und Sekundarschulbildung abschließen, die zu brauchbaren und effektiven Lernergebnissen führt" [1].
Bis 2030 liegen nur mehr acht Jahre vor uns. Wie aus einer kürzlich erschienenen Studie des Münchner ifo Instituts hervorgeht, ist die Welt noch extrem weit davon entfernt die vereinbarten Bildungsziele zu erreichen [2].
Eine umfassende Darstellung der globalen Bildungsdefizite
Ludger Wößmann, Leiter des ifo Zentrums für Bildungsökonomik (München) und ifo-Forscherin Sarah Gust haben zusammen mit dem Bildungsökonomen Eric A. Hanushek von der Stanford University die bisher umfassendste Darstellung der weltweiten Bildungsleistungen von Jugendlichen erarbeitet [2]. Basierend auf individuellen Schülerdaten aus internationalen (Programme for International Student Assessment -PISA - und Trends in International Mathematics and Science Study - TIMSS) und regionalen Leistungstests haben sie die Leistungen auf eine global vergleichbare Skala (die PISA-Skala) gebracht. Insgesamt haben sie dann für 159 Länder (98,1 % der Weltbevölkerung und 99,4 % des weltweiten BIP) den Anteil der Kinder abgeschätzt, die keine Grundfertigkeiten erreichen. Details wie Leistungen in Ländern ohne repräsentative Teilnahme an (internationalen) Tests abgeschätzt werden konnten, sind in [2] beschrieben.
Was ist unter Grundfertigkeiten ("Basic Skills") zu verstehen?
Die Autoren definieren diese als die Fertigkeiten, die für eine effektive Teilnahme an modernen Volkswirtschaften erforderlich sind und an Hand der Beherrschung zumindest der untersten PISA-Kompetenzstufe (1 von 6) gemessen werden. Der Fokus liegt dabei auf Mathematik und Naturwissenschaften (in diesen Fächern lassen sich die Leistungen besser zwischen Ländern vergleichen als - auf Grund der Sprachunterschiede - im Lesen). Laut OECD (2019) liegt die zu erreichende Grenze der PISA-Stufe 1 bei 420 Punkten für Mathematik und bei 410 für Naturwissenschaften.
Kompetenzstufe 1 der PISA-Skala bedeutet für Mathematik: " Schüler können Fragen zu vertrauten Kontexten beantworten, bei denen alle relevanten Informationen gegeben und die Fragen eindeutig definiert sind. Sie sind in der Lage, Informationen zu erfassen und in expliziten Situationen Routineverfahren gemäß direkten Instruktionen anzuwenden. Sie können Schritte ausführen, die fast immer offensichtlich sind und sich unmittelbar aus der jeweiligen Situation ergeben." (OECD [3]) . Was sie nicht können, ist - wie in Stufe 2 erforderlich - einfachste Formeln anwenden, Schlussfolgerungen ableiten oder Ergebnisse interpretieren. Es sind dies allerdings Fähigkeiten, die der Arbeitsmarkt nicht nur in entwickelten Ländern in zunehmendem Maße erfordert, und ohne die selbst der nur für Eigenbedarf produzierende Landwirt nicht auskommt.
Kompetenzstufe 1b der PISA-Skale bedeutet für Naturwissenschaften: "Schüler können auf allgemein bekanntes konzeptuelles Wissen zurückgreifen, um Aspekte einfacher Phänomene zu erkennen. Sie können einfache Muster in Daten und naturwissenschaftliche Grundbegriffe erkennen und expliziten Anweisungen folgen, um ein einfaches naturwissenschaftliches Verfahren anzuwenden". (OECD [3])
Wie hoch sind die globalen Bildungsdefizite?
Die ifo-Studie zeigt erschreckende Zahlen (Abbildung 1):
| Abbildung 1. Geschätzter Anteil der Jugendlichen (inklusive der nicht in der Schule erfassten), die nicht zumindest den untersten Level (PISA 1) der Grundfertigkeiten in Mathematik und Naturwissenschaften erreicht haben. (Bild leicht modifiziert aus [2]; Lizenz cc-by-nc.). |
Weltweit haben mindestens zwei Drittel bis zu drei Viertel der Jugendlichen nicht das Mindestmaß an Grundfertigkeiten (PISA-Stufe 1) erlangt. Am deutlichsten treten die Qualifikationsdefizite bei dem Drittel der Jugendlichen zutage, die keine weiterführende Schule besuchen; allerdings erlangen weltweit auch 61,7 Prozent der Sekundarschüler keine Grundkenntnisse. Tabelle 1.
In 101 der 159 untersuchten Länder liegt der Anteil der Kinder, die keine Grundfertigkeiten erworben haben, über 50 %, in 36 dieser Länder bei über 90 Prozent. Es sind dies Länder mit den niedrigsten Einkommen (< 1085 $/Kopf und Jahr), vor allem in Afrika südlich der Sahara (Bildungsdefizit 94 %). In Südasien liegt das Bildungsdefizit bei 89 %, im nahen Osten bei 68 % und in Lateinamerika bei 65 % (Abbildung 1).
| Spalte 1: Anteil der getesteten Schüler, die Grundfertigkeiten nicht erreichen. Spalte 2: Anteil der Schüler ohne Sekundarbildung (daher nicht getestet), Spalte 3: Alle (getestete und nichtgeteste) Kinder, die Grundfähigkeiten nicht erreichen. (Quelle: Table 2 in [2], (Lizenz cc-by-nc) ); Einkommen pro Kopf und Jahr: http://data.worldbank.org/about/country-classifications.) |
Niedriges Pro-Kopf Einkommen und Fehlen adäquater Schulen korrelieren mit dem Ausmaß der Bildungsdefizite. Allerdings gibt es einige sehr reiche Länder, wie beispielsweise Katar, in denen trotz enormer Investitionen in den Bildungssektor - vom Kindergarten bis zu Hochschulen - (noch) keine dramatische Verbesserung der Situation gelungen ist (in Katar erreichen 57 % der Jugendlichen nicht die unterste Stufe der Grundfertigkeiten).
Bedenklich erscheint, dass in den die meisten reichen Ländern - wie in den USA und den europäischen Staaten (s.u.) - im Mittel rund ein Viertel der Kinder nicht über das Mindestmaß an Grundfertigkeiten verfügt. Weltweit liegt nur in 19 Ländern deren Anteil unter 20 %; dazu gehören Canada, China (hier die Regionen Beijing, Shanghai, Jiangsu, Zhejiang und Guangdong99, dazu Hongkong und Macao, weiters Japan, Südkorea, Singapur, Taiwan und 11 europäische Staaten (diese sind aus Abbildung 2 ersichtlich). Offensichtlich kommt es aber nicht nur auf die Investitionen in Bildung an: In den 5 chinesischen Regionen ist das Einkommensniveau wesentlich niedriger als im OECD-Durchschnitt (3).
Wie hoch sind die Bildungsdefizite in Europa?
Die in Europa erhobenen Schülerleistungen stammen - mit Ausnahme von Zypern (TIMSS-Testung) -aus PISA-Testungen - ein Ländervergleich der Bildungsdefizite ist damit sehr verlässlich. Europaweit haben bis zu 99 % aller Schüler die Sekundarschulstufe besucht; nur in der Ukraine (64,0 %), Nord-Mazedonien (75,7 %), Moldawien (78,0%) und im Kosovo (78,1 %) liegt die Beteiligung deutlich niedriger.
| Abbildung 2. Anteil der Jugendlichen [in %] in europäischen Ländern, die das Mindestmaß an grundlegenden Fähigkeiten in Mathematik und Naturwissenschaften nicht erreichen. Nicht eingetragen sind Zypern (32 %), Malta (33,9 %), Luxemburg (31,3 %) und Liechtenstein (14,5 %). (Die Europakarte ist gemeinfrei, die eingefügten Zahlen sind Tabelle 4A in [2] entnommen (Lizenz cc-by-nc).) |
Europaweit bestehen erhebliche Unterschiede in den Bildungsdefiziten, die von 10,5 % (Estland) bis 79,9 % (Kosovo) reichen (Abbildung 2). Höchst alarmierend erscheint die Situation in Südosteuropa. In Ländern , die zusammen genommen 91 Millionen Menschen zählen, erreichen mehr als 40 % der Schüler nicht die unterste Stufe der Grundfertigkeiten; die höchsten Leistungsdefizite weisen dabei Länder des Westbalkans auf: der Kosovo (79,9 %), gefolgt von Nord-Mazedonien (61,1 %) , Bosnien-Herzegowina (59,7 %), Moldawien (52,3 %) und Montenegro (50,1 %). Auch in den EU-Ländern Rumänien und Bulgarien verfügt nahezu die Hälfte der Schüler nicht über die Grundfertigkeiten von PISA-Stufe 1.
Wovon hängen die Lernerfolge ab?
Wie bereits oben erwähnt, bedarf es primär ausreichend ausgestatteter Bildungssysteme, um gute Schülerleistungen zu erzielen. Der Schulbesuch ist aber nicht gleichbedeutend mit Lernen - dies sieht man an Hand der schlechten Leistungen in Südost-Europa.
Der Ruf nach mehr Geld, nach höheren Investitionen in den Bildungsbereich garantiert nicht unbedingt bessere Lernerfolge:
So wurden in Estland die besten schulischen Leistungen in Europa erzielt, obwohl die dortigen Bildungsausgaben rd. 30% unter dem OECD-Durchschnitt liegen [3].
Auf der anderen Seite ist das schwache Abschneiden von Luxemburg, des weltweit derzeit reichsten Staates, zu vermerken, in dem fast ein Drittel (31,3 %) der Schüler die Grundqualifikation nicht erreichen und dies trotz eines hohen Bildungsbudgets.
Im OECD-Bereich sind im letzten Jahrzehnt die Ausgaben für den Primär- und Sekundarbereich um mehr als 15 % gestiegen, die schulischen Leistungen haben sich aber in den meisten Ländern (72 von 79 Ländern) nicht verbessert [3].
Zweifellos kann auch Migration zum schulischen Abschneiden beitragen. In Ländern, die relativ wenige Zuwanderer aus Kulturkreisen mit niedrigem Bildungsniveau und anderen Wertvorstellungen haben, gibt es auch weniger Probleme in den Schulen und diese sind nicht nur mangelnder Sprachbeherrschung geschuldet.
Ganz wesentlich für den Lernerfolg ist es Schüler - innerhalb der Schule und ausserhalb in Elternhaus und Gesellschaft - zu motivieren, dass sie bereit sind zu lernen.
Fazit
Die Leistungsdaten aus 159 Ländern, die 98 % der Erdbevölkerung abdecken, zeigen ganz klar, dass die Welt noch enorm weit davon entfernt ist allen Kindern Grundfertigkeiten zu vermitteln, die für die Teilhabe an modernen Volkswirtschaften erforderlich sind. Damit erscheint das Entwicklungsziel SDG4 der Agenda 2030 in näherer Zukunft nicht erreichbar. Das Dilemma: Der Großteil der Jugendlichen trifft unvorbereitet auf eine in raschem Wandel begriffene Welt, die mehr und mehr von informationsbasierten Tätigkeiten geprägt wird. Auch in Berufen, die als anforderungsarm gegolten haben, wird es zunehmend wichtiger schriftliche Informationen zu verstehen, sich damit kritisch auseinander zu setzen, rechnerische Verfahren anzuwenden, wissenschaftlich zu denken und auf Evidenz basierende Schlussfolgerungen zu ziehen.
[1] Resolution der Generalversammlung, verabschiedet am 25. September 2015: Transformation unserer Welt: die Agenda 2030 für nachhaltige Entwicklung. . https://www.un.org/Depts/german/gv-70/band1/ar70001.pdf
[2] Sarah Gust, Eric A. Hanashek, Ludger Woessmann: Global Universal Basic Skills: Current Deficits and Implications for World Development. (October 2022). https://docs.iza.org/dp15648.pdf, DOI 10.3386/w30566 .
[3] OECD: PISA 2018 Ergebnisse. Was Schülerinnen und Schüler wissen und können, Band 1 (2019). https://doi.org/10.1787/1da50379-de
Artikel zum Thema Bildung im ScienceBlog
Inge Schuster, 30.10.2021: Eurobarometer 516: Umfrage zu Kenntnissen und Ansichten der Europäer über Wissenschaft und Technologie - blamable Ergebnisse für Österreich
Inge Schuster, 03.10.2021: Special Eurobarometer 516: Interesse der europäischen Bürger an Wissenschaft & Technologie und ihre Informiertheit
IIASA, 17.05.2018: Die Bevölkerungsentwicklung im 21. Jahrhundert - Szenarien, die Migration, Fertilität, Sterblichkeit, Bildung und Erwerbsbeteiligung berücksichtigen.
Inge Schuster, 10.08.2017: Migration und naturwissenschaftliche Bildung
Inge Schuster, 22.6.2017: Der naturwissenschaftliche Unterricht an unseren Schulen
Inge Schuster, 02.01.2015: Eurobarometer: Österreich gegenüber Wissenschaft*, Forschung und Innovation ignorant und misstrauisch
Inge Schuster, 28.02.2014: Was hält Österreich von Wissenschaft und Technologie? — Ergebnisse der neuen EU-Umfrage (Spezial Eurobarometer 401)
Auf der Suche nach einem Religiositätsgen
Auf der Suche nach einem ReligiositätsgenDo, 26.01.2023 — Ricki Lewis
Prädisponieren uns unsere Gene dazu, dass wir vielleicht asozial sind, die Einsamkeit suchen, politischen Ideologien anhängen und möglicherweise, dass wir religiös sind? Zu letzterer Frage hat die US-amerikanische Genetikerin Ricki Lewis unter den Stichworten "Vererbung" und "Religiosität" in Google Scholar nach entsprechenden Berichten gesucht. Was Google Scholar dazu an Untersuchungen aufzeigte, stammte hauptsächlich aus den Sozialwissenschaften. Im Folgenden referiert Lewis kritisch über fünf zwischen 1996 und 2021 veröffentlichte Studien, die der Frage nachgingen, ob Religiosität in unseren Genen steckt.*
| Der erste Funke: Der Garten Eden trifft auf die Doppelhelix. (Harishbabu Laguduva). Lizenz: cc-by-sa |
Ich habe etwas Naturwissenschaftliches gesucht - bringt religiös zu sein einen Vorteil für Überleben zur Reproduzierung, das die treibende Kraft der natürlichen Selektion eines adaptiven vererbten Merkmals ist?
Das normale Google und die Mainstream-Medien ließ ich beiseite, weil ich ja nach Daten und nicht nach Meinungen suchte, und gab als Stichworte "Vererbung" und "Religiosität" ein. Für mich bedeutet Vererbung Gene, die für Proteine kodieren, welche den Phänotyp (Merkmal oder Krankheit) beeinflussen. Vererben bedeutet aber auch, dass von den Eltern etwas an die Nachkommen weitergegeben wird, wie beispielsweise Geld, Hab und Gut oder Wertvorstellungen.
Sicherlich hatte irgendjemand schon eine Genomweite Assoziationsstudie (GWAS) für "Religiosität" durchgeführt. Eine "GWAS" ist eine Untersuchung der Einzelnukleotid-Polymorphismen (SNPs) in einem Genom, d.i. Variationen in einzelnen Basenpaaren bei unterschiedlichen Individuen. Derartige Studien gibt es bereits seit zwei Jahrzehnten; sie versuchen für Eigenschaften wie antisoziales Verhalten, Vereinsamung und sogar politische Ideologien eine genetische Basis zu finden.
Heute verwenden die Forscher einen abgekürzten "polygenen Risikoscore" (PRS), um sogenannte komplexe Merkmale zu beschreiben, d. h. solche, die sowohl von mehreren Genen als auch von Umweltfaktoren beeinflusst werden. Im Gegensatz zu einer Entweder-Oder-Diagnose wie im Fall der Mukoviszidose umfasst ein PRS die Varianten vieler Gene, die zu einem Merkmal oder einer Krankheit beitragen.
Was Google Scholar an Untersuchungen aufzeigte, stammte eher aus den Sozialwissenschaften und verwendete eine Sprache, mit der ich zugegebenermaßen nicht vertraut bin. Hier ist eine kurze Beschreibung von fünf Studien in chronologischer Reihenfolge, die der Frage nachgingen, ob Religiosität in unseren Genen steckt.
1996: "Ein interaktives Modell der Religiositätsvererbung: Die Bedeutung des familiären Kontextes"
In diesem Bericht im Journal American Sociological Review hat Scott M. Myers von der Penn State University "das Ausmaß der Vererbung von Religiosität anhand von Interviews mit 471 Eltern im Jahr 1980 und ihren erwachsenen Nachkommen im Jahr 1992" abgeschätzt [1]. Sein Befund, dass die Religiosität einer Person von der elterlichen Religiosität abhängt, ist wenig überraschend. Verblüffend ist aber die antiquierte Sprache, die ja aus dem noch nicht so lange zurückliegenden Jahr 1996 stammt:
"Eine glückliche Ehe der Eltern, Unterstützung des Kinds durch die Eltern, angemessene Strenge und ein berufstätiger Ehemann/nicht berufstätige Ehefrau erhöhen die Fähigkeit der Eltern, ihre religiösen Überzeugungen und Gebräuche weiterzugeben." Du lieber Himmel!
2004: "Das Gottes-Gen: Wie der Glaube in unseren Genen fest verdrahtet ist"
Mit dem Konzept eines Gottes-Gens hat sich der Genetiker Dean Hamer in seinem viel zitierten Buch "Das Gottes-Gen" auseinander gesetzt. Es handelt sich um den am National Cancer Institute (Bethesda) tätigen Forscher, der bereits 1993 mit seiner inzwischen widerlegten Entdeckung eines "Schwulen-Gens" Schlagzeilen gemacht hat [2].
Im "Das Gottes-Gen" führt Hamer die "Veranlagung von Menschen zu spirituellen oder mystischen Erfahrungen" auf Varianten eines Gens, nämlich des für den vesikulären Monoamintransporter 2 (VMAT2) kodierenden Gens, zurück (VMAT2 transportiert in der Nervenzelle Monoamine aus dem Cytosol in Vesikel; Anm. Redn.). Laut Online Mendelian Inheritance in Man (d.i. der "Bibel" der Humangenetik) ist die korrekte Funktion des Gens "unerlässlich für die einwandfreie Leistung der monoaminergen Systeme, die bei etlichen neuropsychiatrischen Störungen des Menschen eine Rolle spielen. Der Transporter ist ein Wirkort für wichtige Medikamente".
Zu den Monoamin-Neurotransmittern gehören Serotonin, Dopamin, Adrenalin und Noradrenalin, die starke Auswirkungen auf die Gemütslage haben. Können also Variationen im Monoamin-Feld tatsächlich für religiöse Gedanken und sogar Visionen verantwortlich sein?
Hamers Kandidat des Gens für ein höheres Wesen tauchte 2017 in einem Bericht von Linda A. Silveira (von der University of Redlands) in der Zeitschrift Life Sciences Education wieder auf: "Experimentieren mit Spiritualität: Analyse des Gottes-Gens in einem Laborkurs". Die Studenten haben Varianten in ihren VMAT2-Genen untersucht. Die Autorin bewertete die Übung als ein Lehrmittel. Und das war es auch, denn die Klasse widerlegte die Hypothese, dass Varianten des Gens etwas mit dem Bekenntnis zu einer Religion zu tun haben.
2011: "Religion, Fruchtbarkeit und Gene: Ein Modell der Dualen Vererbung"
In einer Veröffentlichung der Royal Society untersuchte der Wirtschaftswissenschaftler Robert Rowthorn von der University of Cambridge anhand eines Modells die evolutionären Auswirkungen der Tatsache, dass religiöse Menschen im Durchschnitt mehr Kinder haben als ihre säkularen Pendants" [4]. Diese Aussage ist nicht belegt und wird als Allgemeinwissen dargestellt.
Rowthorn legte seine Annahmen dar: (1) die Kultur bestimmt die Fruchtbarkeit und (2) die "genetische Veranlagung" beeinflusst die Neigung zur Religion. "Menschen, die ein bestimmtes 'Religiositäts'-Gen besitzen, werden überdurchschnittlich oft religiös oder bleiben religiös", erklärte er. Dies ist eine Abwandlung der allzu simplen "a-gene-for"-Mentalität, auch bekannt als genetischer Determinismus.
Ich konnte nicht umhin, mich zu fragen, was ein Religionsgen eigentlich sein könnte? Ein DNA-Abschnitt, der für eine Hämoglobinvariante kodiert? Eine Form von Kollagen? Ein Blutgerinnungsfaktor? Ein Verdauungsenzym? Ist es vielleicht doch die abgelehnte VMAT2-Variante von Hamer?
Rowthorn schreibt: "Die Arbeit betrachtet die Auswirkungen von religiösen Übertritten und Exogamie auf die religiöse und genetische Zusammensetzung der Gesellschaft."
Ich wusste, dass Endogamie in der Genetik die Paarung innerhalb einer Gruppe bedeutet. Daher war ich nicht überrascht, als ich entdeckte, dass Exogamie "die soziale Norm ist, außerhalb der eigenen sozialen Gruppe zu heiraten". In der Biologie bedeutet Exogamie Auskreuzung (Tiere) oder Fremdbestäubung (Pflanzen). Einzeller wie Bakterien, Amöben und bestimmte Schleimpilze, die keine sozialen Normen haben, wachsen einfach und teilen sich dann.
Der Wirtschaftswissenschaftler versuchte, die Wissenschaft hinter seiner Hypothese des "Religiositätsgens" zu erklären, wobei er unbewusst die Konzepte der natürlichen Selektion und der genetischen Verknüpfung berührte:
"Sich von der Religion Lossagende verringern den Anteil der Bevölkerung mit religiöser Zugehörigkeit und verlangsamen die Ausbreitung des Religiositätsgens. Wenn jedoch der Unterschied in der Fruchtbarkeit bestehen bleibt, und Menschen mit religiösem Bekenntnis sich hauptsächlich mit Gleichgesinnten paaren, wird das Religiositätsgen trotz einer hohen Lossagungsrate schließlich überwiegen. Dies ist ein Beispiel einer "Gen-Kultur Koevolution'', in der sich ein Gen ausbreitet, weil es in der Lage ist, sich an ein kulturelles Brauchtum mit hoher Fitness anzuhängen."
(Nach Darwin bedeutet Fitness explizit: lange genug zu überleben, um sich fortzupflanzen.)
Rechnerische Modellierungen, so Rowthorns Schlussfolgerung, stützen seine "theoretischen Argumente" über die Ausbreitung eines Religiositätsgens.
2017: "Religiöse Wahnvorstellungen bei Schizophrenie"
Eine im Journal European Neuropsychopharmacology veröffentlichte Studie stößt in den Bereich der diagnostischen Medizin vor [5]. Forscher aus Deutschland haben polygene Risikoscores verwendet, um einen Zusammenhang zwischen "starker religiöser Aktivität" und einem erhöhten Schweregrad der Schizophrenie aufzuzeigen.
"Von 271 Patienten (217 Christen, 9 Muslime, 45 ohne religiöses Bekenntnis) erlebten 102 (38 %) religiöse Wahnvorstellungen während der Krankheitsepisoden", so die Forscher. "Die bloße Zugehörigkeit zu einer Religion korrelierte nicht mit einer höheren Wahrscheinlichkeit von Wahnvorstellungen, wohl aber eine "starke religiöse Aktivität".
"Andere untersuchte Faktoren - Alter, Geschlecht, Bildungsstand, Familienstand und sogar die Frage, ob ein Patient während der Befragung mitten in einem Wahnzustand war - schienen keine Rolle zu spielen. Und je höher die angegebene Religiosität war, desto größer war das Risiko von Wahnvorstellungen", so die Schlussfolgerung der Forscher.
Aber auch das Vorhandensein von mehr prädisponierenden Genen (höhere PRS) korrelierte mit der Wahrscheinlichkeit von Wahnvorstellungen. "Unsere Ergebnisse legen nahe, dass das Auftreten religiöser Wahnvorstellungen bei Schizophrenie und schizoaffektiven Störungen sowohl mit Umwelteinflüssen als auch mit genetischen Faktoren zusammenhängt. Eine moderate religiöse Aktivität scheint jedoch keine negativen Auswirkungen zu haben und könnte sogar hilfreich sein, um mit diesen Störungen fertig zu werden", so die Forscher.
2021: "Die Ursprünge des religiösen Unglaubens: Ein Ansatz der Dualen Vererbung"
Eine Studie in der Fachzeitschrift Social Psychological and Personality Science befasst sich mit den Hintergründen des Nichtglaubens [6]. Der Artikel beginnt mit der Beschreibung von Atheisten, als wären wir Außerirdische.
In dieser sozialwissenschaftlichen Untersuchung werden "theoretische Rahmenbedingungen für die wichtigsten Prädiktoren des religiösen Unglaubens" erörtert. Wir Atheisten "erleben weniger glaubwürdige kulturelle Hinweise auf religiöses Engagement, ... gefolgt von einem reflektierenden kognitiven Stil, ... und einer weniger fortgeschrittenen Mentalisierung."
Da meine Mentalisierung anscheinend zurückgeblieben ist, dachte ich, ich würde das mal genauer untersuchen. Die Wikipedia-Definition ist stumpfsinnig: "Mentalisierung kann als eine Form der imaginativen mentalen Aktivität betrachtet werden, die es uns ermöglicht, menschliches Verhalten in Form von intentionalen mentalen Zuständen wahrzunehmen und zu interpretieren."
Die Definition eines Begriffs sollte den Begriff nicht verwenden, etwas, das ich in dieser kleinen Auswahl der Soziologie-Literatur zuhauf fand. Ich denke, dass meine mangelnde Mentalisierung bedeutet, dass ich nicht unvoreingenommen bin, was vielleicht ein Umweg ist, um zu sagen, dass ich wie ein Naturwissenschaftler denke - ich ziehe Beweise der Phantasie und den Gefühlen vor.
Diesem Bericht zufolge ist Atheismus eher bei Menschen anzutreffen, die "kulturell relativ wenig mit Religion in Berührung kommen". Eine verblüffende Erkenntnis!
-------------------------------------------------------------
Meine Eltern haben mich mit unserer Religion vertraut gemacht. In der dritten Klasse habe ich mittwochs nach der Schule den "Religionsunterricht" besucht.
In der ersten Stunde habe ich aufmerksam der Geschichte der Schöpfung zugehört und in meinem Kopf die Drittklässler-Version eines Stammbaums aufgebaut. Ich war konsterniert. Woher kamen die Frauen von Kain und Abel?
Ich stellte diese Frage und man sagte mir, ich solle still sein. Aber ich hob immer wieder meine Hand. Gehörten die Ehefrauen von Kain und Abel zur Gattung Australopithecus, waren sie Neandertaler oder Schimpansen? Ich hatte die faszinierende Ausstellung von Hominidenköpfen im American Museum of Natural History vor Augen.
Ich bin nicht mehr zum "Religionsunterricht" zurückgekehrt. Meine Mutter wollte die angeborene Neugierde eines zukünftigen Wissenschaftlers nicht unterdrücken.
Wenn es ein Gen für Religiosität gibt, dann ist meines sicherlich gelöscht. Aber das ist in Ordnung.
----------------------------------------------------------------------------------------
[1] Scott M. Myers: An Interactive Model of Religiosity Inheritance: The Importance of Family Context. American Sociological Review Vol. 61, No. 5 (Oct., 1996), pp. 858-866. https://doi.org/10.2307/2096457
[2] Ricki Lewis: Retiring the Single Gay Gene Hypothesis. 29.09.2019. https://dnascience.plos.org/2019/08/29/retiring-the-single-gay-gene-hypothesis/
[3] Linda A. Silveira: Experimenting with Spirituality: Analyzing The God Gene in a Nonmajors Laboratory Course. (2017) CBE—Life Sciences EducationVol. 7, No. 1. https://doi.org/10.1187/cbe.07-05-0029
[4] Robert Rowthorn: Religion, fertility and genes: a dual inheritance model.(2011). Proc. Royal Soc. https://doi.org/10.1098/rspb.2010.2504 . .
[5] Heike Anderson-Schmidt et al., T41 - Dissecting Religious Delusions In Schizophrenia: The Interplay Of Religious Activity And Polygenic Burden. . European Neuropsychopharmacology Volume 27, Supplement 3, , 2017.
[6] Will M.Gervais et al., The Origins of Religious Disbelief: A Dual Inheritance Approach. (2021)SAGE journals 12,7. https://doi.org/10.1177/1948550621994001
* Der Artikel ist erstmals am 22. Dezember 2022 in PLOS Blogs - DNA Science Blog unter dem Titel "In Search of a Religiosity Gene"https://dnascience.plos.org/2022/12/22/in-search-of-a-religiosity-gene/ erschienen und steht unter einer cc-by Lizenz . Die Autorin hat sich freundlicherweise mit der Übersetzung ihrer Artikel durch ScienceBlog.at einverstanden erklärt, welche so genau wie möglich der englischen Fassung folgen.
NASA-Analyse: 2022 war das fünftwärmste Jahr seit Beginn der Aufzeichnungen
NASA-Analyse: 2022 war das fünftwärmste Jahr seit Beginn der AufzeichnungenDo, 19.01.2023 — Redaktion
Nach einer Analyse der NASA war die durchschnittliche Oberflächentemperatur der Erde im Jahr 2022 gleichauf mit 2015 die fünftwärmste seit Beginn der Aufzeichnungen. Der langfristige Erwärmungstrend der Erde setzt sich fort; laut den Wissenschaftlern des Goddard Institute for Space Studies (GISS) der NASA lagen die globalen Temperaturen 2022 um 0,89 Grad Celsius über dem Durchschnitt des NASA-Bezugszeitraums (1951-1980).*
| Abbildung 1. Globale Temperaturanomalien im Jahr 2022. (NASA Earth Observatory images by Joshua Stevens, based on data from the NASA Goddard Institute for Space Studies) |
"Dieser Erwärmungstrend ist alarmierend", sagt NASA-Administrator Bill Nelson. "Unser sich erwärmendes Klima hinterlässt bereits seine Spuren: Waldbrände nehmen zu, Hurrikane werden stärker, Dürren richten Verwüstungen an und der Meeresspiegel steigt. Die NASA verstärkt ihr Engagement, um ihren Teil zur Bekämpfung des Klimawandels beizutragen. Unser Earth System Observatory wird hochaktuelle Daten zur Unterstützung unserer Klimamodellierung, -analyse und -vorhersage bereitstellen, um der Menschheit bei der Bewältigung des Klimawandels auf unserem Planeten zu helfen."
Die letzten neun Jahre waren die wärmsten Jahre seit Beginn der modernen Aufzeichnungen im Jahr 1880. Das bedeutet, dass die Erde im Jahr 2022 etwa 1,11°C wärmer war als der Durchschnitt des späten 19. Jahrhunderts.
Die Landkarte in Abbildung 1. zeigt die globalen Temperaturanomalien im Jahr 2022. Das sind nicht die absoluten Temperaturen, sondern um wie viel wärmer oder kühler die einzelnen Regionen der Erde im Vergleich zum Durchschnitt der Jahre 1951 bis 1980 waren.
Das Balkendiagramm in Abbildung 2. gibt das Jahr 2022 im Kontext mit den Temperaturanomalien seit 1880 wieder. Es sind dies über den gesamten Globus gemittelte Oberflächentemperaturen für die jeweiligen Jahre.
| Abbildung 2. Globale Temperaturanomalien seit 1880 bezogen auf den Temperatur-Durchschnitt im Zeitraum 1951 - 1980. (Abweichungen in o C.) NASA Earth Observatory images by Joshua Stevens, based on data from the NASA Goddard Institute for Space Studies |
"Der Grund für den Erwärmungstrend liegt darin, dass der Mensch fortfährt enorme Mengen an Treibhausgasen in die Atmosphäre zu pumpen, und die langfristigen Auswirkungen auf dem Planeten werden sich fortsetzen", sagt Gavin Schmidt, Direktor des GISS, dem führenden Zentrums der NASA für Klimamodellierung.
Nach einem kurzzeitigen Absinken aufgrund der COVID-19-Pandemie im Jahr 2020 sind die vom Menschen verursachten Treibhausgasemissionen wieder angestiegen. Kürzlich haben NASA-Wissenschaftler, ebenso so wie auch internationale Wissenschaftler festgestellt, dass die Emissionen von Kohlendioxid im Jahr 2022 so hoch waren wie nie zuvor. Mit Hilfe des EMIT-Instruments (Earth Surface Mineral Dust Source Investigation), das letztes Jahr zur Internationalen Raumstation gebracht wurde, hat die NASA außerdem einige Superemittenten von Methan - einem weiteren starken Treibhausgas - identifiziert (siehe dazu: [1]).
Die stärksten Erwärmungstendenzen weist nach wie vor die arktische Region auf: der Temperaturanstieg ist hier fast viermal so hoch wie im globalen Durchschnitt -, so die GISS-Untersuchung, die auf der Jahrestagung 2022 der American Geophysical Union vorgestellt wurde, sowie eine separate Studie.
Gemeinschaften auf der ganzen Welt leiden unter Auswirkungen, die Wissenschaftler in Zusammenhang mit der Erwärmung der Atmosphäre und der Ozeane sehen. Der Klimawandel hat Niederschläge und tropische Stürme verstärkt, Dürreperioden verschärft und die Auswirkungen von Sturmfluten verstärkt. Das vergangene Jahr brachte sintflutartige Monsunregenfälle, die Pakistan verwüsteten, und eine anhaltende Megadürre im Südwesten der USA. Im September wurde Hurrikan Ian zu einem der stärksten und kostspieligsten Wirbelstürme, die das US-amerikanische Festland heimsuchten.
Unser Planet im Wandel
Die globale Temperaturanalyse der NASA stützt sich auf Daten, die von Wetterstationen und Forschungsstationen in der Antarktis sowie von Instrumenten auf Schiffen und Bojen gesammelt wurden. NASA-Wissenschaftler analysieren diese Messungen, um Unsicherheiten in den Daten zu berücksichtigen und einheitliche Methoden für die Berechnung der globalen durchschnittlichen Oberflächentemperaturunterschiede für jedes Jahr beizubehalten. Diese Bodenmessungen der Oberflächentemperatur stimmen mit den Satellitendaten überein, die seit 2002 vom Atmospheric Infrared Sounder auf dem Aqua-Satelliten der NASA gesammelt wurden, sowie mit anderen Abschätzungen.
Um zu erfassen, wie sich die globalen Temperaturen im Laufe der Zeit verändern, verwendet die NASA den Zeitraum von 1951 bis 1980 als Bezugslinie. Diese Basislinie umfasst Klimaverläufe wie La Niña und El Niño, ebenso wie ungewöhnlich heiße oder kalte Jahre, die auf andere Faktoren zurückzuführen sind, und stellt sicher, dass die natürlichen Schwankungen der Erdtemperatur berücksichtigt werden.
Viele Faktoren können die Durchschnittstemperatur in einem bestimmten Jahr beeinflussen. So war beispielsweise das Jahr 2022 eines der wärmsten seit Beginn der Aufzeichnungen, obwohl im tropischen Pazifik das dritte Jahr in Folge La Niña herrschte. NASA-Wissenschaftler schätzen, dass der kühlende Einfluss von La Niña die globalen Temperaturen geringfügig (etwa 0,06 °C) gegenüber dem Durchschnitt gesenkt haben könnte, der unter typischeren Meeresbedingungen erreicht worden wäre.
Eine separate, unabhängige Analyse der National Oceanic and Atmospheric Administration (NOAA) ergab, dass die globale Oberflächentemperatur im Jahr 2022 die sechsthöchste seit 1880 war. Die NOAA-Wissenschaftler verwenden für ihre Analyse größtenteils dieselben Temperatur-Rohdaten, haben aber einen anderen Bezugszeitraum (1901-2000) und eine andere Methodik. Obwohl die Rangfolge für bestimmte Jahre zwischen den Aufzeichnungen leicht abweichen kann, stimmen sie weitgehend überein und spiegeln beide die anhaltende langfristige Erwärmung wider.
Der vollständige Datensatz der NASA zu den globalen Oberflächentemperaturen bis zum Jahr 2022 sowie alle Einzelheiten mit Code zur Durchführung der Analyse durch die NASA-Wissenschaftler sind beim GISS öffentlich zugänglich [2]
[1] Redaktion, 03.11.2022: NASA: neue Weltraummission kartiert weltweit "Super-Emitter" des starken Treibhausgases Methan
[2] Goddard Institute for Space Studies: GISS Surface Temperature Analysis (GISTEMP v4). https://data.giss.nasa.gov/gistemp
* Der vorliegende Artikel ist als Presseaussendung unter dem Titel " NASA Says 2022 Fifth Warmest Year on Record, Warming Trend Continues" am 12. Jänner 2023 auf der Web-Seite der NASA erschienen
https://www.nasa.gov/press-release/nasa-says-2022-fifth-warmest-year-on-record-warming-trend-continues.. Der unter einer cc-by stehende Artikel wurde von der Redaktion möglichst wortgetreu übersetzt und mit den Abbildungen aus https://earthobservatory.nasa.gov/images/150828/2022-tied-for-fifth-warmest-year-on-record ergänzt.
Weiterführende Links
NASA Goddard: A Look Back: 2022's Temperature Record (12.1.2023), Video 4:41. min. https://www.youtube.com/watch?v=GxXmIgcfFn4&t=4s (Credit: NASA's Goddard Space Flight Center Kathleen Gaeta (Lizenz: gemeinfrei))
Schwerpunktsthema im ScienceBlog. Bis jetzt behandeln mehr als 40 Artikel den Klimawandel - von Modellierungen über Folgen des Klimawandels bis hin zu Strategien seiner Eindämmung; chronologisch gelistet in Klima & Klimawandel
Probiotika - Übertriebene Erwartungen?
Probiotika - Übertriebene Erwartungen?So 15.01.2023 — Inge Schuster

![]() Innerhalb weniger Jahre ist ein milliardenschwerer Markt für Probiotika entstanden, d.i. für "lebende Mikroorganismen, die, wenn sie in angemessener Menge verabreicht werden, dem Wirt einen gesundheitlichen Nutzen bringen ". Die Werbung verspricht, dass solche "guten" Mikroorganismen eine "gestörte" Darmflora verbessern und in Folge diverseste Erkrankungen verhindern oder heilen können. Für diese Behauptungen fehlt größtenteils ein stichhaltiger, wissenschaftlicher Nachweis.
Innerhalb weniger Jahre ist ein milliardenschwerer Markt für Probiotika entstanden, d.i. für "lebende Mikroorganismen, die, wenn sie in angemessener Menge verabreicht werden, dem Wirt einen gesundheitlichen Nutzen bringen ". Die Werbung verspricht, dass solche "guten" Mikroorganismen eine "gestörte" Darmflora verbessern und in Folge diverseste Erkrankungen verhindern oder heilen können. Für diese Behauptungen fehlt größtenteils ein stichhaltiger, wissenschaftlicher Nachweis.
Als wir In den späten 1970er Jahren im ehemaligen Wiener Sandoz-Forschungsinstitut an neuen, oral verabreichbaren Wirkstoffen gegen Pilzinfektionen forschten, hat mein Labor dazu u.a. mit Untersuchungen zur Aufnahme (Absorption) der Substanzen aus dem Darm beigetragen. Dies war ein Gebiet, in dem der damalige Wissenstand und die experimentellen Möglichkeiten noch sehr bescheiden waren; insbesondere betraf dies auch das Thema, ob und welchen Einfluss Darmbakterien auf die Physiologie/Pathologie (nicht nur) des Verdauungstraktes haben könnten. Hat man auf Tagungen führende Experten danach befragt, kam stereotyp die Antwort: "Das wissen wir leider nicht."
Einige Jahrzehnte später erlebt nun der Markt für Probiotika einen riesigen Boom; laut Definition der International Scientific Association for Probiotics and Prebiotics (ISAPP) versteht man darunter "lebende Mikroorganismen, die, wenn sie in angemessener Menge verabreicht werden, dem Wirt einen gesundheitlichen Nutzen bringen". Die Werbung sieht diese als "gute" Bakterien, deren Anwendung zur Prävention und Therapie eines unglaublichen Spektrums an Krankheiten verhelfen soll. Beispielsweise schreibt das Portal https://probiotics.org/benefits/ "Um nur einige der Wirkungen zu nennen: Probiotische Bakterien bekämpfen Krebs, beugen Karies vor, reduzieren Allergien, senken den Blutdruck und helfen Ihnen, besser zu schlafen!" und listet dann im Detail mehr als 100 unterschiedliche Krankheitsbilder auf, gegen die Probiotika wirksam sein sollen. Solchen Heilsversprechen kann man nicht entrinnen, nicht in den Medien - vor allem in der TV-Werbung zur Hauptsendezeit -, nicht im Internet auf Webseiten und auf sozialen Plattformen.
Man fragt sich hier primär: Wissen wir überhaupt schon genug über unsere Darmbakterien und über gesundheitliche Auswirkungen, wenn deren Gemeinschaften gestört sind (d.i. bei sogenannten Dysbiosen)? Und in weiterer Folge: Welche Evidenz gibt es dafür, dass Probiotika die Darmflora "verbessern" können und damit den einen oder anderen gesundheitlichen Nutzen erbringen?
Was wissen wir über das menschliche Mikrobiom.....
Fakt ist: Wir leben in einer Symbiose mit komplexen Gemeinschaften von Mikroorganismen, die uns in der Regel nicht nur keinen Schaden zufügen, sondern für Wohlbefinden und Aufrechterhaltung der Gesundheit sogar unerlässlich sind. Lange Zeit schien es allerdings ziemlich aussichtslos die auf und in uns lebenden mikrobiellen Gemeinschaften in ihrer Gesamtheit - das, was man nun als das Mikrobiom versteht - charakterisieren zu wollen. Mit den etablierten mikrobiologischen Methoden hätte man ja die unterschiedlichen Stämme der Mikroorganismen isolieren und in Kultur bringen müssen, um sie dann sequenzieren zu können - in Anbetracht der im Dickdarm (Colon) hauptsächlich anaerob (ohne Sauerstoff) existierenden Keime allerdings ein heilloses Unterfangen.
Neue Techniken, vor allem in der Sequenzierung der DNA, wie sie im Human Genome Project erfolgreich angewandt wurden, haben seit der Mitte der 2000-Jahre eine umfassende Untersuchung von mikrobiellen Gemeinschaften ermöglicht ohne, dass man deren einzelne Vertreter vorher isolieren und kultivieren musste.
Initiiert von den US-National Institutes of Health (NIH) und mit 170 Mio $ unterstützt, startete 2007 das Human Microbiome Project (HMP - https://hmpdacc.org/), eine 10 Jahre laufende, bahnbrechende Initiative, an der sich mehr als 40 Forschungseinrichtungen vorwiegend aus den US aber auch aus Kanada und Belgien beteiligten.
In der ersten Phase hat sich das HMP auf die Identifizierung und Charakterisierung der mit dem Menschen assoziierten Mikroorganismen konzentriert und darauf, ob es so etwas, wie ein zentrales gesundes Mikrobiom - also gemeinsame Elemente im Mikrobiom gesunder Menschen - gibt. Dazu wurden von 242 gesunden Versuchspersonen Proben der mikrobiellen Gemeinschaften an verschiedenen Stellen des Körpers - vom Nasen-Mundraum über die Haut, den Magen-Darm-Trakt bis zum Urogenitaltrakt - mittels metagenomischer Whole Genome Shotgun Sequenzierung charakterisiert. In der zweiten, bis 2016 laufenden Phase sollte das Zusammenspiel von Wirtsorganismus und Mikrobiom, einschließlich Immunität, Stoffwechsel und dynamischer molekularer Aktivität, erforscht werden, um Einblicke in die Rolle der Mikroben für Gesundheit und Krankheit zu gewinnen. Dazu wurden Datensätze biologischer Eigenschaften sowohl des Mikrobioms als auch des Wirts aus drei verschiedenen Kohortenstudien zu Mikrobiom-assoziierten Erkrankungen erstellt: aus Studien i) zu Schwangerschaft und Frühgeburten, ii) dazu, wie entzündliche Darmerkrankungen ihren Ausgang nehmen und iii) wie Diabetes Typ2 anfängt und fortschreitet.
Während seiner Laufzeit sind aus dem Projekt bereits Hunderte wissenschaftliche Publikationen hervorgegangen. Das gesamte Datenmaterial ist auf dem HMP-Portal (https://portal.hmpdacc.org/) als Basis für viele weitere Untersuchungen, Analysen und Hypothesen öffentlich zugänglich.
...... und speziell über das Mikrobiom im Darm?
Die bisherigen Ergebnisse aus dem Human Microbiome Project aber auch aus vielen anderen, größeren Studien haben unsere Kenntnisse über die komplexen Gemeinschaften von Mikroorganismen im Darm erheblich erweitert und auch erste fundierte Informationen zur Rolle der Bakterien in Gesundheit und Krankheit beigetragen. Wir beginnen ein besseres Bild über die Diversität und ungeheure Variabilität der Gemeinschaften von Mikroorganismen zu bekommen, über den Einfluss, den Alter, Geschlecht, Ethnizität, Diät, Lebensstil, Krankheit und Arzneimittelkonsum auf diese Gemeinschaften haben und auch über deren enorme Fähigkeit der Adaptation auf geänderte Bedingungen.
Abweichend von früheren Schätzungen geht man nun davon aus , dass uns etwa gleich viele Bakterien (rund 38 Billionen Zellen) besiedeln, wie wir Körperzellen (rund 30 Billionen) besitzen. Bis zu 1000 unterschiedliche Bakterienspezies (plus deren Subspezies) wurden festgestellt, wobei der Darm - und hier speziell das Colon - um 2 Größenordnungen mehr Keime enthält als alle anderen Körperstellen zusammen. Im Colon machen diverse Stämme von Bacteroideten und Firmicuten bis zu 90 % der Besiedlung aus. Bis zu 100 Milliarden Keime/ml finden sich in der eindickenden Masse im Colon, bis zu 60 % der eingedickten und mit dem Kot ausgeschiedenen (Trocken)Masse bestehen aus Bakterien.
Wenn Bakterien auf Grund ihrer wesentlich geringeren Größe auch nur etwa 0,3 % (ca. 200 g) unseres Körpergewichts ausmachen, so tragen sie auf Grund ihres extensiven Metabolismus und ihrer enormen Vermehrungsrate (Verdoppelung innerhalb weniger Minuten) essentiell zur Homöostase unseres Verdauungssystems bei. Wie weit dies für die lange Strecke im über 5 m langen Jejunum (dem oberen Dünndarm) gilt, in dem zwar nur sehr niedrige und passager auftretende Mengen an Bakterien (1 000 - 10 000/ml) vorkommen, aber über 90 % der verwertbaren Stoffe des Nahrunsgsbreis aber auch Arzneistoffe absorbiert werden, ist nicht bekannt.
Was allerdings aus dem Ileum (dem unteren Dünndarm) an noch unverdauten und unverdaulichen Bestandteilen des Nahrungsbreis ins Colon gelangt, wird von den dort ansässigen Bakterien in absorbierbare, für uns verwertbare Produkte umgewandelt, u.a. in kurzkettige Fettsäuren (Essigsäure, Propionsäure. Buttersäure) und in zahlreiche andere essentielle Produkte (u.a. in die Vitamine Biotin, Vitamin K). Welche möglicherweise bioaktiven Stoffwechselprodukte hier von Bakterien freigesetzt werden (das sogenannte Metabolom), wie sie diese zu Kommunikation und Abwehr untereinander und mit dem Wirtsorganismus nutzen, ist Gegenstand intensiver Untersuchungen, insbesondere, ob und welche Auswirkungen derartige Substanzen auf Stoffwechsel, Immunsystem und Körperorgane bis hin zum CNS haben.
Die Darmflora ist einmalig, wie ein Fingerabdruck und zudem dynamisch - sie adaptiert sich laufend auf die Situation des jeweiligen Wirts und setzt einer Kolonisierung mit fremden pathogenen Organismen Resistenz entgegen.
Nach wie vor ist das Mikrobiom des Darms aber größtenteils eine "terra incognita".
Erst jetzt gibt es erste Ergebnisse zu Lokalisierung und Konzentration der verschiedenen Bakterien-Spezies in den einzelnen Darmabschnitten, erst jetzt Informationen darüber, wo sich die Bakterien dort befinden: assoziiert an das Darmepithel, an die darüber liegende Mucus-Schichte oder im Darmlumen diffundierend.
Über Ausmaß, Lokalisierung und mögliche Auswirkungen unserer Symbiose mit anderen Darmbewohnern wie Archaeen, Pilzen, Parasiten und Viren (vorzugsweise Bakteriophagen) ist noch viel zu wenig bekannt.
Der Probiotika Boom
Wahrscheinlich haben Menschen bereits vor Jahrtausenden begonnen vergorene Lebensmittel - wie Sauerkraut, Joghurt und Kefir -, die große Mengen an Lactobacillen (Milchsäure produzierende Bakterien) enthalten, zu konsumieren. Die Entdeckung der Lactobazillen in diesen Lebensmitteln und die überdurchschnittliche Langlebigkeit bulgarischer Bauern, deren Ernährung sehr viel Joghurt enthielt, ließen den russischen Physiologen und Nobelpreisträger Ilya Metschnikow 1907 erstmals die Hypothese von einer gesundheitssteigernden, lebensverlängernden Wirkung der Lactobazillen herleiten, für die er den Begriff "Probiotika" formulierte. Joghurt wurde in Folge in vielen Ländern populär und 1935 kam in Japan ein mit einem Lactobacillus casei-Stamm angereichertes Joghurt - Yakult - auf den Markt, das auch heute noch zu den globalen Blockbustern zählt. Dann geschah jahrzehntelang nichts.
Der Probiotika-Boom setzte erst viel später ein. An Hand der Veröffentlichungen in wissenschaftlichen Fachjournalen kann er etwa mit dem Ende der 1990er Jahre datiert werden. So findet man in der US-Datenbank PubMed unter dem Suchbegriff "probiotics" insgesamt rund 41 000 Publikationen. Vom Jahr 2000 (212 Publikationen) an gibt es eine nahezu exponentiell wachsende Zahl jährlicher Publikationen. Abbildung 1. Eine ähnliche Zeitkurve erhält man auch auf dem Portal Google Scholar, das allerdings wesentlich mehr Zeitschriften erfasst und auf insgesamt 684 000 Publikationen kommt. Sucht man auf auf Google selbst, das ja Webseiten von Firmen und Werbung erfasst, nach "probiotics" kommt man auf 138 Millionen Einträge.
| Abbildung 1. Ab 2000 findet ein rasanter Anstieg der Publikationen über Probiotika statt (Quelle: https://pubmed.ncbi.nlm.nih.gov/ abgerufen am 2.1.2023) . |
Wie Probiotika wirken können
Zu den in Probiotika verwendeten "guten" Bakterien zählen diverse Stämme von Michsäurebakterien (Lactobazilli) , Bifidobakterien und E. coli. (Daneben werden auch Hefepilze eingesetzt.)
Davon ausgehend, dass Probiotika eine "gestörte" Darmflora verbessern, gibt es verschiedene Mechanismen, wie dies bewirkt wird und diese können von Bakterienstamm zu Bakterienstamm unterschiedlicher Natur sein.
"Gute" Bakterien können das Wachstum pathogener Bakterien hemmen, indem sie u.a. mit den Pathogenen um die Besiedlung der Darmschleimhaut konkurrieren, den pH-Wert im Darmlumen absenken, Toxine abbauen, bioaktive Stoffwechselprodukte ausscheiden, die gegen die Pathogene gerichtet sind und/oder auch als Signalmoleküle auf das Immunsystem, das endokrine System und das Nervensystem wirken.
Die Voraussetzung dafür: Eine ausreichende Menge an "guten" Bakterien muss die Passage durch Magen und Dünndarm überleben und vermehrungsfähig ins Colon gelangen, um dort, in dem dicken Brei aus Nahrungsresten und bis zu 100 Milliarden Mikroorganismen im Milliliter eine Chance zu haben gegen pathogene Keime vorzugehen. Wie weit dies bei Probiotika in üblichen Dosierungen von 5 bis zu 50 Milliarden Keimen der Fall ist, bleibe dahingestellt.
Ein wachsender Markt..........
Gleichzeitig mit dem Interesse an Probiotika ist auch der Markt für probiotische Joghurts, Nahrungsergänzungsmittel, Hygieneprodukte etc. schnell gewachsen. Zahlreiche Firmen teilen sich den Markt; zu den wichtigsten Akteuren zählen Yakult Honsha, Danone, Morinaga, Nestle, PepsiCo Inc., Bio-K Plus International Inc. und Daflorn Ltd. Die Größe des globalen Markts wurde 2021 mit 61,1 Milliarden US $ beziffert, bei einem jährlichen Wachstum von 8,3 % soll er 2029 115,71 Milliarden US $ erreichen ((https://www.maximizemarketresearch.com/market-report/probiotics-market-size/542/). (Zum Vergleich: der globale Pharmamarkt lag 2022 bei 1 136 Milliarden US $.)
Vertrieben über Supermärkte, Apotheken und online verleiten gesundheitsbezogene Angaben (d.i. Angaben, mit denen ein Zusammenhang zwischen einem Produkt und Gesundheit suggeriert wird) zur Selbstmedikation und dies auch bei schweren Erkrankungen. Für Probiotika in Lebensmittel sind derartige gesundheitsbezogene Angaben nicht erlaubt. Medizinprodukte mit probiotischen Inhaltstoffen fallen mit dem Inkrafttreten der Europäischen Verordnung über Medizinprodukte (Mai 2021) in den Geltungsbereich der Richtlinie über Human-Arzneimittel. Wie für diese sind also sehr aufwändige, teure klinische Studien zum Nachweis von Wirksamkeit und Sicherheit erforderlich, um eine Zulassung zu erreichen.
...erforderliche klinische Studien......
Forschungsinstitutionen und Industrie haben bereits eine sehr große Zahl an klinischen Studien zur Wirksamkeit von Probiotika durchgeführt, viele laufen noch oder sind in Planung.
Auf der Webseite der von den NIH betriebenen größten Datenbank für klinische Studien https://clinicaltrials.gov/ finden sich unter dem Schlagwort "probiotic" insgesamt 2051 Studien (abgerufen am 2.1.2023). Davon wurden 1140 schon abgeschlossen, darunter 464 von der Industrie durchgeführte Studien. Die meisten der abgeschlossenen Studien untersuchten die Wirksamkeit gegen Infektionskrankheiten (238), Durchfall (94), Reizdarm (66), Entzündung (64), Adipositas (62), vaginale Erkrankungen (42) und Stress (42). Weitere Studien behandelten Depression, kognitive Fähigkeiten, Diabetes, atopische Dermatitis, Krebserkrankungen, ulcerative Colitis, Enteroclitis, Akne, Karies, Schlafstörungen und Epilepsie.
Der Großteil der Studien hat bis jetzt bestenfalls Hinweise aber keine Evidenz auf Wirksamkeit in der untersuchten Indikation erbracht.
....und wie wirksam haben sich Probiotika erwiesen?
Probiotika liegen im Zeitgeist, die rezeptfreien Produkte entsprechen dem Trend zur Selbstmedikation. Das Internet ist hier eine wesentliche Informationsquelle.
Wie steht es aber um die Qualität dieser Informationen?
Ein Team um Michel Goldman (Université Libre de Bruxelles) hat 2020 dazu eine erhellende Studie "Online-Informationen über Probiotika: Stimmen sie mit den wissenschaftlichen Erkenntnissen überein?" veröffentlicht (M. Neunez et al., Front. Med. 6:296. doi: 10.3389/fmed.2019.00296). Gestützt auf Cochrane Reviews hat das Team 150 der von Google zu "probiotics" retournierten Top-Webseiten auf Genauigkeit, Vollständigkeit und Qualität der Informationen ausgewertet.
| Abbildung 2 . Nur rund ein Viertel der geundheitsbezogenen Behauptungen für Probiotika sind durch stichhaltige Beweise gesichert. (Bild: modifiziert nach Neunez M., et al., 2020. Online Information on Probiotics: Does It Match Scientific Evidence? Front. Med. 6:296. doi: 10.3389/fmed.2019.00296. Lizenz: cc-by) |
Das Ergebnis: Am häufigsten kamen kommerzielle Webseiten und News vor. Die kommerziellen Webseiten haben im Durchschnitt die am wenigsten verlässlichen Informationen geliefert und viele der behaupteten Leistungen von Probiotika wurden nicht durch wissenschaftliche Evidenz gestützt. Insgesamt wurden nur 24 % der Behauptungen durch stichhaltige Belege untermauert; für 20 % gab es keine Belege. In den anderen Fällen reichten die gelieferten Belege nicht aus , um die Behauptungen zu rechtfertigen. Abbildung 2.
Nur insgesamt 25 % der Seiten (nur 8 % der kommerziellen Seiten) beinhalten auch Sicherheitsaspekte. Auch, wenn Probiotika ein gutes Sicherheitsprofil haben, kann es bei immunschwachen Personen zu einem Überfluten des Dünndarms mit den "guten" aber dort unverdaulichen Keimen kommen, insbesondere wenn die Magensäure durch Protonenpumpen-Inhibitoren neutralisiert wurde. Es kommen auch vereinzelte Fälle von bakterieller oder fungaler Sepsis vor.
Von besonderem Interesse ist es natürlich bei welchen Krankheiten stichhaltige Evidenz für die behauptete Wirksamkeit von Probiotika gefunden wurde. Abbildung 3. Dies war nur bei einigen Magen-Darmerkrankungen der Fall. Für den Großteil anderer Erkrankungen sind die Heilsversprechen als Wunschdenken anzusehen.
| Abbildung 2. Wissenschaftliche Evidenz für die behauptete Wirksamkeit von Probiotika auf online Webseiten bei diversen Erkrankungen; hergeleitet von Cochrane Reviews. Grün: stichhaltige Eviden, gelb: mäßige Evidenz, orange: geringe Evidenz, rot: keinerlei Evidenz. (Bild: unverändert von Neunez M., et al., 2020, Lizenz: cc-by) |
Zu einem ähnlichen Ergebnis kommt auch das Office of Dietary Supplements der NIH in Probiotics. Fact Seet for Health Professionals (https://ods.od.nih.gov/factsheets/Probiotics-HealthProfessional/). Metaanalysen verschiedener Krankheiten, in denen Probiotika angeblich wirkten, zeigten nur im Fall von Infektions- und Antibioka-verursachtem Durchfall bei Kindern Evidenz.
Fazit
Die Industrie hat in wenigen Jahren einen riesigen Markt für Probiotika aufgebaut. Diese sind zu lifestyle-Produkten geworden, auch wenn die gesundheitsbezogenen Behauptungen größtenteils noch leere Heilsversprechen ohne wissenschaftliche und klinische Evidenz sind.
Das Gebiet ist überaus komplex und steckt noch in den Kinderschuhen. Es fehlt an essentiellen wissenschaftlichen Grundlagen sowohl dazu, was ein "gesundes" balanciertes Mikrobiom ist, als auch wie dieses mit den Zellen der Darmwand interagiert und wie die enorme Fülle seiner Stoffwechselprodukte (Metabolom) unsere Organsysteme beeinflussen könnte. Sicherlich wird es noch Jahre dauern bis - aufbauend auf besseren Grundlagen - verstanden wird, wie "Gesundheit im Darm beginnt" und mit welchen Interventionen dies erreicht werden kann.
Soll man Probiotika nach dem Motto "hilft's nicht, schadets auch nicht" anwenden? In Anbetracht recht weniger, jedoch schwerer Nebenwirkungen ist Vorsicht angebracht.
Das Mikrobiom im ScienceBlog
Inge Schuster, 18.09.2020: Spermidin - ein Jungbrunnen, eine Panazee?
Inge Schuster, 03.01.2019: Wie Darmbakterien den Stoffwechsel von Arzneimitteln und anderen Fremdstoffen beeinflussen
Dario Valanzano, 28.06.2018: Mikroorganismen im Darm sind Schlüsselregulatoren für die Lebenserwartung ihres Wirts
Norbert Bischofberger, 24.05.2018: Auf dem Weg zu einer Medizin der Zukunft
Redaktion, 10.05.2018: Anton Weichselbaum und das menschliche Mikrobiom - ein Vortrag vor 125 Jahren
Francis S. Collins, 28.09.2017: Ein erweiterter Blick auf das Mikrobiom des Menschen
Redaktion, 22.12.2016: Kenne Dich selbst - aus wie vielen und welchen Körperzellen und Mikroben besteht unser Organismus?