Mi, 08.04.2020 — Redaktion

![]() Bereits vor drei Jahren haben wir über die Datenbank "BioNumbers" (- "database of key numbers in molecular and cell biology") berichtet [1,2], auf der quantitative biologische Daten in allen Details schnellstens aufgerufen werden können. Der Biologe Ron Milo (Professor am Weizmann-Institut, Rehovot), einer der Gründer der Datenbank, hat nun zusammen mit dem Biophysiker Rob Phillips (Professor am Caltech, Pasadena) und ihren Studenten Yinon Bar-On und Avi Flamholz die Datenbank erweitert: Sie haben Hunderte Studien - vor allem aus jüngster Zeit- zu Coronaviren kritisch durchgesehen und aus diesen die wichtigsten Informationen zur Biologie des Sars-CoV-2 Virus selbst und zur Infektion mit dem Virus zusammengefasst [3]. Von der Infektion über die Ausbreitung des Virus bis hin zu Ansätzen für Prävention und Therapie bieten die quantitativen Daten eine überaus wertvolle Basis für alle, die an der Eindämmung der Pandemie arbeiten.*
Bereits vor drei Jahren haben wir über die Datenbank "BioNumbers" (- "database of key numbers in molecular and cell biology") berichtet [1,2], auf der quantitative biologische Daten in allen Details schnellstens aufgerufen werden können. Der Biologe Ron Milo (Professor am Weizmann-Institut, Rehovot), einer der Gründer der Datenbank, hat nun zusammen mit dem Biophysiker Rob Phillips (Professor am Caltech, Pasadena) und ihren Studenten Yinon Bar-On und Avi Flamholz die Datenbank erweitert: Sie haben Hunderte Studien - vor allem aus jüngster Zeit- zu Coronaviren kritisch durchgesehen und aus diesen die wichtigsten Informationen zur Biologie des Sars-CoV-2 Virus selbst und zur Infektion mit dem Virus zusammengefasst [3]. Von der Infektion über die Ausbreitung des Virus bis hin zu Ansätzen für Prävention und Therapie bieten die quantitativen Daten eine überaus wertvolle Basis für alle, die an der Eindämmung der Pandemie arbeiten.*
Die derzeitige SARS-CoV-2-Pandemie ist eine schmerzliche Erinnerung an die Tatsache, dass die Virendynamik - ob bei einem einzelnen menschlichen Wirt oder auch bei einer, die Kontinente überziehenden Infektionswelle - meist eine Geschichte der Zahlen ist. In der folgenden Zusammenfassung wird eine kuratierte grafische Quelle über die wichtigsten Zahlen bereitgestellt, die helfen soll, das Virus zu verstehen, das zu unserer aktuellen globalen Krise führte. Die Zusammenfassung dreht sich um zwei große Themen:
- die Biologie des Virus selbst und
- die Merkmale der Infektion eines einzelnen menschlichen Wirts.
Die Zusammenfassung enthält die Schlüsselzahlen für SARS-CoV-2, die hauptsächlich auf von Experten geprüfter Literatur (peer-reviewed) basieren (und im Originaltext [3] mit den entsprechenden Referenzen belegt sind). Die Leser sollen sich jedoch daran erinnern, dass noch viel Unsicherheit besteht und sich das Wissen über diese Pandemie und das Virus, das diese antreibt, rasch weiterentwickelt.
In den folgenden Abschnitten sind vereinfachte Berechnungen vorgestellt, welche die Einblicke veranschaulichen, die sich aus der Kenntnis einiger Schlüsselzahlen und der Verwendung quantitativer Logik ergeben. Diese Berechnungen dienen dazu, unsere Einsicht zu verbessern, ersetzen jedoch keine detaillierte epidemiologische Analyse.
1.Wie lange dauert es bis eine infizierte Person eine Million Infizierter generiert?
Wenn sich alle so wie gewohnt verhalten, wie lange würde es dauern, bis sich von einer Person ausgehend die Pandemie auf eine Million infizierter Opfer ausbreitet?
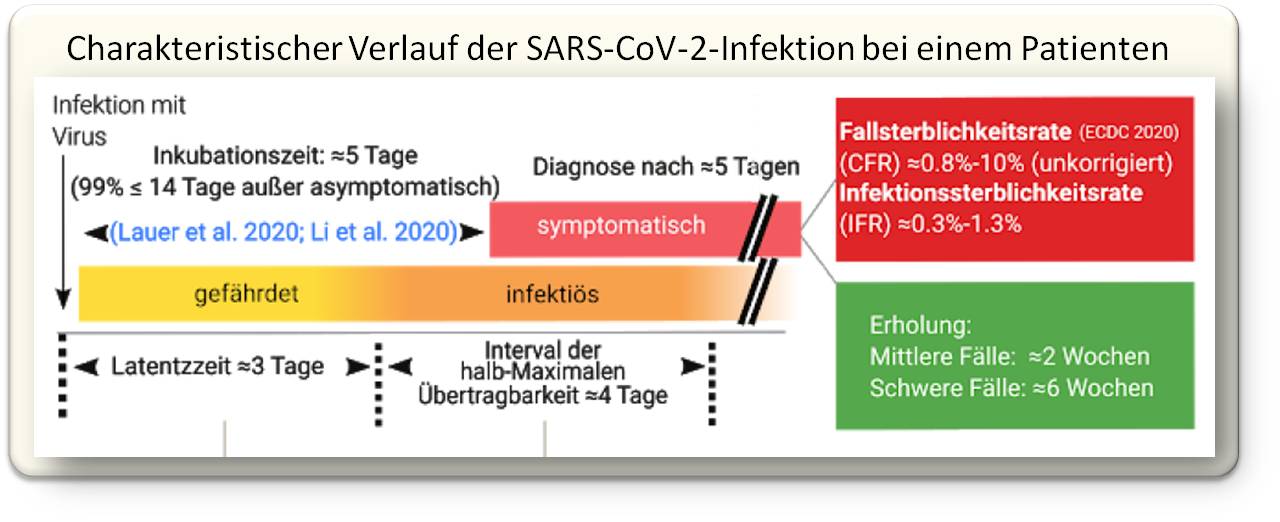
Die Basisreproduktionszahl R0 gibt an, dass jede Infektion auf direktem Weg 2 bis 4 weitere Infektionen erzeugt, sofern keine Gegenmaßnahmen wie beispielsweise soziale Distanzierung getroffen werden. Sobald man infiziert ist, dauert es eine gewisse Zeit, die sogenannte Latenzzeit, bevor man das Virus übertragen kann. Die derzeit beste Schätzung der mittleren Latenzzeit sind ungefähr 3 Tage, darauf folgen ca. 4 Tage der nahezu maximalen Infektiosität. Die genaue Dauer variiert bei den einzelnen Patienten und einige sind wesentlich länger ansteckend. Abbildung 1.
| Abbildung 1. Zeitlicher Verlauf der von einem Patienten ausgehenden Infektion. Die Schätzungen sind an Parametern des Bevölkerungsdurchschnitts in China angepasst und beschreiben nicht die interindividuelle Variabilität |
Bei Verwendung von R0 ≈ 4 vervierfacht sich die Anzahl der Fälle ca. alle 7 Tage oder verdoppelt sich alle ≈3Tage. Ein 1000-faches Wachstum (von einem Fall auf 103) erfordert 10 Verdopplungen (210 = 1024); 3 Tage × 10 Verdopplungen = 30 Tage oder ungefähr ein Monat. Wir erwarten also ein 1000-faches Wachstum in einem Monat, ein Wachstum auf eine Million (106) in zwei Monaten und auf eine Milliarde (109) in drei Monaten.
Auch wenn diese Berechnung stark vereinfacht ist und die Auswirkungen von „Superverbreitern“, Herden-Immunität und unvollständigen Testungen dabei ignoriert werden, zeigt sie doch ganz deutlich, dass sich Viren in einem enormen Tempo verbreiten können, wenn keine Gegenmaßnahmen ergriffen werden. Dies veranschaulicht, warum es entscheidend ist, die Verbreitung des Virus durch Maßnahmen einer sozialen Distanzierung einzudämmen.
Was ist die Berstgröße (burst size) und die Replikationszeit des Virus?
Zwei wichtige Merkmale des viralen Lebenszyklus sind die Zeit, die er benötigt, um neue infektiöse Nachkommen zu produzieren, und die Anzahl der Nachkommen, die jede infizierte Zelle produziert. Die Ausbeute an neuen Virionen, die pro infizierter Zelle freigesetzt werden - die Berstgröße -, ist in vivo sehr schwierig zu bestimmen. Daher greifen Forscher normalerweise auf das Messen dieser Werte in Gewebekulturen zurück. Abbildung2.
|
Abbildung 2.Innerhalb von 10 Stunden produziert eine infizierte Zelle rund 1000 neue Virionen. (* bedeutet größenordnungsmäßig; die Experimente wurden in Zellkulturen ausgeführt). |
2. Wie wirkt sich soziale Distanzierung aus?
Ein stark vereinfachtes, quantitatives Beispiel verdeutlicht die Notwendigkeit sozialer Distanzierung:
Angenommen, Sie sind infiziert und treffen im Laufe eines Arbeitstages, auf dem Weg zur Arbeit, im privaten Umgang oder bei Besorgungen auf 50 Personen. Um die Zahlen rund zu machen, nehmen wir weiter an, dass Sie bei jeder dieser Begegnungen eine 2%-ige Chance haben, das Virus zu übertragen, sodass Sie wahrscheinlich jeden Tag eine neue Person infizieren. Wenn Sie 4 Tage lang infektiös sind, infizieren Sie durchschnittlich 4 andere Personen, was ungefähr dem höheren R0-Werten für SARS-CoV-2 entspricht, wenn keine soziale Distanzierung besteht. Wenn Sie jedoch aufgrund von sozialer Distanzierung jeden Tag auf 5 Personen (noch besser auf weniger) treffen, infizieren Sie 0,1 Personen pro Tag oder 0,4 Personen, bevor Sie weniger infektiös werden. Der gewünschte Effekt der sozialen Distanzierung besteht darin, dass jede vorliegende Infektion weniger als 1 Neuinfektion hervorruft.
Eine effektive Reproduktionszahl (Re) kleiner als 1 wird sicherstellen, dass die Anzahl der Infektionen letztendlich zurückgeht. Es ist von entscheidender Bedeutung, schnell Re <1 zu erreichen und dies ist wesentlich besser erreichbar ist, als Re durch Maßnahmen im öffentlichen Gesundheitswesen auf nahe Null zu bringen
3. Warum beträgt die Quarantänezeit zwei Wochen?
Der Zeitraum von der Infektion bis zum Auftreten von Symptomen wird als Inkubationszeit bezeichnet. Die mittlere SARS-CoV-2-Inkubationszeit wird auf rund 5 Tage geschätzt. Dennoch gibt es von Mensch zu Mensch große Unterschiede. Etwa 99% derjenigen, die Symptome zeigen, zeigen diese vor dem 14. Tag, was die zweiwöchige Isolationszeit erklärt (Abbildung 1).
Was hier wichtig ist: bei dieser Analyse fallen infizierte Personen, die keinerlei Symptome zeigen, unter den Tisch. Da asymptomatische Menschen normalerweise nicht getestet werden, ist es immer noch nicht klar, wie viele solcher Fälle es gibt oder wie lange asymptomatische Menschen infektiös bleiben.
4. Wie blockieren N95-Masken SARS-CoV-2?
N95-Masken sind so konzipiert, dass sie mehr als 95% aller Partikel mit einem Durchmesser von mindestens 0,3 µm (Mikrometern = ⅟₁₀₀₀ mm) abhalten (NIOSH 42 CFR Part84). Tatsächlich zeigen Messungen zur Effizienz der Filtration von N95-Masken,dass sie in der Lage sind, 99,8% der Partikel mit einem Durchmesser von ~0,1 μm zu filtern. SARS-CoV-2 ist ein behülltes Virus mit einem Durchmesser von ~0,1 μm. Daher können N95-Masken die meisten freien Virionen filtern, aber sie leisten mehr als das.
Wie?
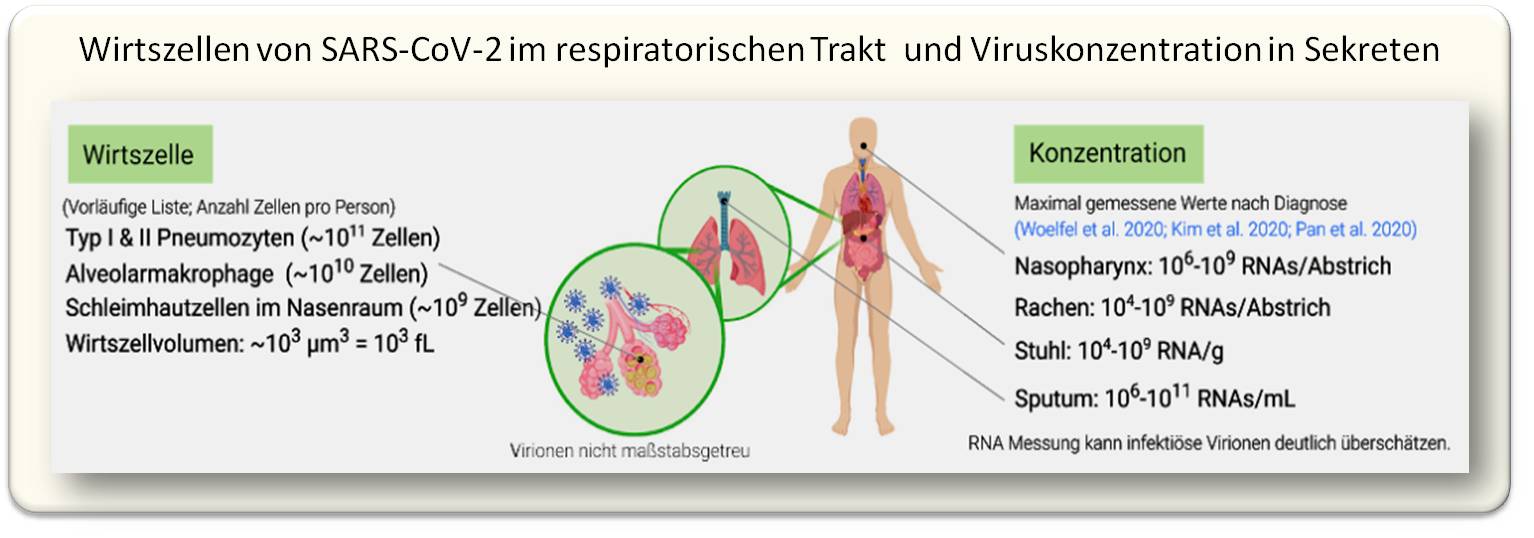
Viren werden häufig durch Atemtröpfchen übertragen, die durch Husten und Niesen entstehen. Wie viele Viren in den Sekreten enthalten sein können, ist in Abbildung 3 dargestellt.
| Abbildung 3. Targetzellen für SARS-CoV-2 im respiratorischen Trakt und maximale Virenkonzentration im Nasenrachenraum, Sputum und Stuhl von COVID-19 Patienten. (Anm. Redn.: Ein großes, durch Niesen erzeugtes Tröpfchen könnte demnach bis zu100 000 Viren enthalten; allerdings überschätzt die RNA-Bestimmung die Zahl aktiver Virionen - siehe Punkt 8.) |
Atemtröpfchen werden normalerweise in zwei Größen unterteilt, große Tröpfchen (>5 μm – Mikrometer=1/1000mm – im Durchmesser),die schnell auf den Boden fallen und daher nur über kurze Entfernungen übertragen werden, und kleine Tröpfchen (≤5 μm im Durchmesser). Kleine Tröpfchen können sich als Aerosole („Tröpfchenkerne“) verflüchtigen, bleiben über einen beträchtlichen Zeitraum in der Luft und können eingeatmet werden. Einige Viren wie Masern können durch Tröpfchenkerne übertragen werden. Derzeit gibt es keine direkten Hinweise auf eine SARS-CoV-2-Übertragung durch Tröpfchenkerne. Es wird vielmehr angenommen, dass größere Tröpfchen der Hauptvektor derSARS-CoV-2-Übertragung sind, üblicherweise durch Absetzen auf Oberflächen, die von Händen berührt und dann auf Schleimhäute wie Augen, Nase und Mundtransportiert werden (Details hier).
Der charakteristische Durchmesser großer Tröpfchen, die durch Niesen erzeugt werden, beträgt ~100 μm, während der Durchmesser von Tröpfchenkernen, die durch Hustenerzeugt werden, in der Größenordnung von ~1 μm liegen. Daher schützen N95-Masken wahrscheinlich vor verschiedenen Arten der Virusübertragung.
5. Wie ähnlich ist SARS-CoV-2 den Erkältungs- und Grippeviren?
SARS-CoV-2 ist ein Beta-Coronavirus, dessen Genom ein einzelner ≈30-kB (30 000 Basen)-RNA-Strang ist. Die Grippe (Influenza) wird durch eine völlig andere Familie von RNA-Viren verursacht, die Influenzaviren genannt werden. Grippeviren haben kleinere Genome (≈14 kB), die in 8 verschiedenen RNA-Strängen kodiert sind und sie infizieren menschliche Zellen auf andere Weise als Coronaviren.
Die „Erkältung“ wird durch eine Vielzahl von Viren verursacht, darunter einige Coronaviren und Rhinoviren. Erkältung verursachende Coronaviren (z. B. OC43- und 229E-Stämme) sind SARS-CoV-2 in der Länge des Genoms (innerhalb 10%) und der Zahl der Gene ziemlich ähnlich, sie unterscheiden sich jedoch in der Sequenz der Nukleotide von SARS-CoV-2 (≈50% Identität der Nukleotide) und im Schweregrad der Infektion.
Eine interessante Facette von Coronaviren ist, dass sie das größte Genom aller bekannten RNA-Viren haben (≈30 kb). Diese großen Genome haben Forscher veranlasst das Vorhandensein eines „Korrekturmechanismus“ zur Reduzierung der Mutationsrate und Stabilisierung des Genoms zu vermuten. Tatsächlich besitzen Coronaviren einen Korrekturmechanismus in Form einer Exonuklease namens ExoN, was ihre sehr niedrigen Mutationsraten (~10-6 pro Position und Replikationszyklus) im Vergleich zur Influenza (≈3×10-5 pro Position und Zyklus) erklärt.
Diese relativ niedrige Mutationsrate wird für künftige Studien von Interesse sein, die vorhersagen, mit welcher Geschwindigkeit Coronaviren unseren Bemühungen zur Immunisierung entgehen können
6. Wie viel ist über das Genom und Proteom von SARS-CoV-2 bekannt?
SARS-CoV-2 besitzt ein Genom, das aus einem Einzelstrang RNA (mit positiver Polarität) besteht, das für 10 Gene kodiert, die letztendlich 26 Proteine produzieren (entsprechend einer NCBI-Annotation NC_045512).
Wie kommt es, dass 10 Gene für mehr als 20 Proteine kodieren?
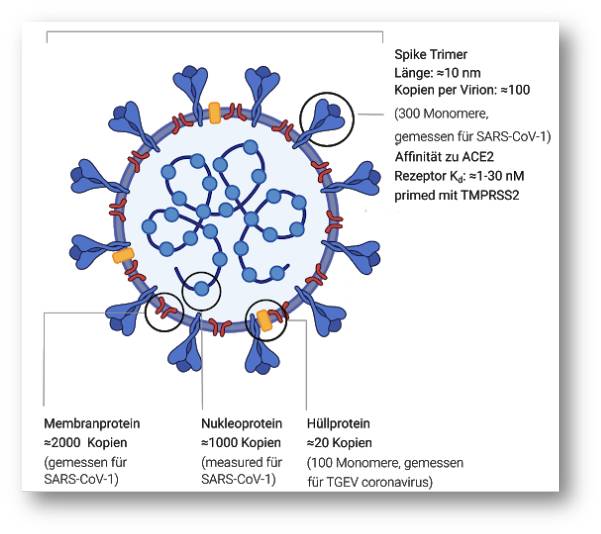
Ein langes Gen (orf1ab), kodiert für ein Polyprotein, das von Proteasen, die selbst Teil des Polyproteins sind, in 16 Proteine gespalten wird. Zusätzlich zu den Proteasen kodiert das Polyprotein eine RNA-Polymerase und zugehörige Faktoren, um das Genom, eine Korrektur-Exonuklease und mehrere andere Proteine, die keine Struktur-Proteine sind, zu kopieren. Die verbleibenden Gene kodieren überwiegend für Struktur-Proteine des Virus: (i) das Spike-Protein, das an den zugehörigen Rezeptor an einer menschlichen oder tierischen Zelle andockt; (ii) ein Nukleoprotein, welches das Genom verpackt; und (iii) zwei membrangebundene Proteine. Abbildung 4.
| Abbildung 4. Aufbau von SARS-CoV-2. Das Virus hat einen Durchmesser von ca. 100 nm (Nanometer – 0,1 µm) und ein Volumen von 106 nm3. |
Obwohl sich viele aktuelle Arbeiten auf das Verständnis der Rolle von „akzessorischen“ Proteinen im viralen Lebenszyklus konzentrieren, schätzen wir, dass es derzeit nur möglich ist, etwa der Hälfte der SARS-CoV-2-Genprodukte klare biochemische oder strukturelle Funktionen zuzuschreiben.
7. Was können wir aus der Mutationsrate des Virus lernen?
Bei der Untersuchung der Virusentwicklung verwenden die Forscher üblicherweise zwei Messgrößen, welche die Geschwindigkeit der genomischen Veränderung beschreiben.
Die erste ist die Evolutionsrate, die als die durchschnittliche Anzahl von Substitutionen definiert ist, die pro Jahr in Stämme des Virus fix eingebaut werden, angegeben in Einheiten von Mutationen pro Position und Jahr.
Die zweite ist die Mutationsrate, das heißt die Anzahl der Substitutionen pro Position pro Replikationszyklus. Wie können wir diese beiden Werte in Beziehung setzen?
Betrachten Sie eine einzelne Position am Ende eines Jahres. Die einzige Messung einer Mutationsrate in einem β-Coronavirus legt nahe, dass diese Position in jeder Replikationsrunde ungefähr 1 Millionstel (~10-6) Mutationen akkumuliert. Jede Runde des Replikationszyklus dauert ~10 Stunden. Es gibt also 1000 Zyklen pro Jahr. Multipliziert man die Mutationsrate mit der Anzahl der Replikationen und vernachlässigt man die möglichen Auswirkungen der evolutionären Selektion und des Drifts, so erhält man 1 Tausendstel (10-3) Mutationen pro Position und Jahr, was mit der, von den aussequenzierten Coronavirus-Genomen abgeleiteten Evolutionsrate übereinstimmt.
Da unsere Schätzung mit der gemessenen Rate übereinstimmt, schließen wir, dass sich das Virus in freier Wildbahn nahezu kontinuierlich repliziert und ständig neue Mutationen erzeugt, die sich im Laufe des Jahres ansammeln.
Mit unserem Wissen über die Mutationsrate können wir auch Rückschlüsse auf einzelne Infektionen ziehen. Da beispielsweise die Mutationsrate ~10-6 Mutationen / Stelle / Zyklus beträgt und ein ml Sputum möglicherweise mehr als 107 virale RNAs enthält, schließen wir, dass jede Stelle in solchen Proben mehr als einmal mutiert ist.
8. Wie stabil und wie ansteckend ist das Virion auf Oberflächen?
Abbildung 5.
| Abbildung 5.Wie stabil ist SARS-CoV in der Umwelt? Die Relevanz für den persönlichen Schutz ist (noch) unklar. Die Daten beruhen auf messbaren infektiösen Virionen. Virale RNA ist selbst nach Wochen auf Oberflächen noch nachweisbar. |
SARS-CoV-2-RNA wurde noch mehrere Wochen nach einem letzten Kontakt auf verschiedenen Oberflächen nachgewiesen. (Der Nachweis von viraler RNA in einer Probe impliziert nicht notwendigerweise das Vorhandensein von infektiösen Virionen; diese könnten defekt sein (z. B. durch Mutation) oder durch Umweltbedingungen inaktiviert worden sein.)
Die Wahrscheinlichkeit einer Infektion des Menschen durch eine solche Exposition ist noch nicht erhoben, da Experimente zur Durchführung dieser Bestimmung äußerst problematisch sind. Dessen ungeachtet müssen Vorsichts- und Schutzmaßnahmen getroffen werden.
Wir schätzen, dass während der Infektionsperiode eine nicht diagnostizierte infektiöse Person mehrere Dutzend Oberflächen berührt. Diese Oberflächen werden anschließend von Hunderten anderen Personen berührt. Aus der Basisreproduktionszahl R0 ≈2-4 können wir ableiten, dass nicht jeder, der diese Oberflächen berührt, infiziert wird.
Genaue Abgrenzungen des Infektionsrisikos bei Berühren von Oberflächen müssen dringend untersucht werden.
[1] Redaktion, 29.12.2020: Wie groß, wie viel, wie stark, wie schnell,… ? Auf dem Weg zu einer quantitativen Biologie.
[2] Redaktion, 22.12.2016: Kenne Dich selbst - aus wie vielen und welchen Körperzellen und Mikroben besteht unser Organismus?
[3] Yinon M. Bar-On, Avi Flamholz, Rob Phillips, and Ron Milo: SARS-CoV-2 (COVID-19) by the numbers . eLife, March 32, 2020.
https://elifesciences.org/articles/57309.
http://book.bionumbers.org/wp-content/uploads/2020/04/german_translation_0408.pdf
*Der Artikel von Yinon M. Bar-On et al.: SARS-CoV-2 (COVID-19) by the numbers (in der der deutschen Übersetzung von Lars Albert Eicholt, Vanessa Pahl: http://book.bionumbers.org/wp-content/uploads/2020/04/german_translation_0408.pdf) steht unter einer cc-by Lizenz. Er wurde im Wesentlichen unverändert übernommen (allerdings ohne den Großteil des Glossars). Die meisten Bilder aus der grafischen Zusammenfassung wurden in den Text eingefügt, die Legenden dazu stammen aus dem hier nicht angehängten Glossar. Wegen der besseren Lesbarkeit fehlen die zahlreichen, den Daten zugrundeliegen Literaturstellen in der ScienceBlog Version - diese können von der Originalversion [3] aus aufgerufen werden.
Zu SARS-CoV-2 im ScienceBlog erschienen:
Matthias Wolf, "Extrablatt" v. 06.04.2020: Ein kleines Corona – How-to.
IIASA, 02.04.2020: COVID-19 - Visualisierung regionaler Indikatoren für Europa.
Inge Schuster, 26.03.2020: Drug Repurposing - Hoffnung auf ein rasch verfügbares Arzneimittel zur Behandlung der Coronavirus-Infektion
Redaktion, 18.03.2020: Experimenteller Impfstoff gegen SARS-CoV-2 bereits in klinischer Phase 1-Testung
Francis S. Collins, 05.03.2020: Strukturbiologie weist den Weg zu einem Coronavirus-Impfstoff
- Printer-friendly version
- Log in to post comments