Do, 11.02.2021 — Francis S. Collins

![]() In den letzten Tagen überstürzen sich die Pressemeldungen über neue Varianten von SARS-CoV-2 - dem Verursacher von COVID-19 -, die in anderen Teilen der Welt aufgetreten sind und auch bei uns bereits entdeckt werden. Insbesondere bei der erstmals in Südafrika identifizierten, mit B.1.351 bezeichneten Variante, wächst die Besorgnis inwieweit deren Mutationen dem Virus helfen könnten den derzeit aktuellen Behandlungen mit Antikörpern und hochwirksamen Impfstoffen zu entkommen. Francis S. Collins, ehem. Leiter des Human Genome Projects, ist langjähriger Direktor der US-National Institutes of Health (NIH), die zusammen mit dem Unternehmen Moderna den eben zugelassenen COVID-19- Impfstoff mRNA-1723 designt und entwickelt haben. Er berichtet über NIH-geförderte Untersuchungen, welche aus der Kartierung der Mutationen am Strukturmodell des viralen Spike-Proteins Aussagen zu Wirksamkeit von Antikörpern und Vakzinen ermöglichen.*
In den letzten Tagen überstürzen sich die Pressemeldungen über neue Varianten von SARS-CoV-2 - dem Verursacher von COVID-19 -, die in anderen Teilen der Welt aufgetreten sind und auch bei uns bereits entdeckt werden. Insbesondere bei der erstmals in Südafrika identifizierten, mit B.1.351 bezeichneten Variante, wächst die Besorgnis inwieweit deren Mutationen dem Virus helfen könnten den derzeit aktuellen Behandlungen mit Antikörpern und hochwirksamen Impfstoffen zu entkommen. Francis S. Collins, ehem. Leiter des Human Genome Projects, ist langjähriger Direktor der US-National Institutes of Health (NIH), die zusammen mit dem Unternehmen Moderna den eben zugelassenen COVID-19- Impfstoff mRNA-1723 designt und entwickelt haben. Er berichtet über NIH-geförderte Untersuchungen, welche aus der Kartierung der Mutationen am Strukturmodell des viralen Spike-Proteins Aussagen zu Wirksamkeit von Antikörpern und Vakzinen ermöglichen.*
Im Laborversuch ist es bereits möglich vorherzusagen, welche Mutationen dem SARS-CoV-2 Virus dazu verhelfen werden, unseren Therapien und Impfstoffen zu entkommen, und sich sogar auf das Auftreten neuer Mutationen vorzubereiten, noch bevor diese auftreten. Eben dies hat eine NIH-finanzierte Studie gezeigt, die ursprünglich im November als bioRxiv-Vorabdruck erschien und kürzlich, von Experten begutachtet, in Science veröffentlicht wurde. In dieser Studie haben die Forscher alle möglichen Mutationen kartiert, die es SARS-CoV-2 ermöglichen würden, einer Behandlung mit drei verschiedenen monoklonalen Antikörpern zu widerstehen, welche für die Behandlung von COVID-19 entwickelt wurden [1].
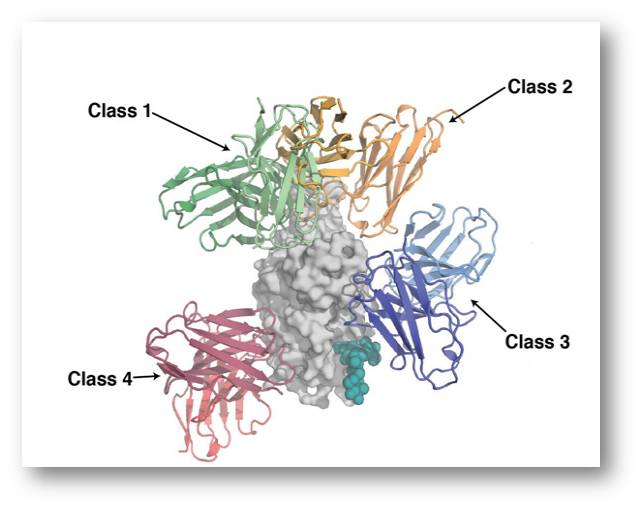
Unter der Leitung von Jesse Bloom, Allison Greaney und Tyler Starr vom Fred Hutchinson Cancer Center in Seattle hat sich die Untersuchung auf die Schlüsselregion des Spike-Proteins, die sogenannte Rezeptorbindungsdomäne (RBD), konzentriert. Mit dieser RBD dockt das an der Virusoberfläche sitzende Protein an den ACE2-Rezeptor menschlicher Zellen an, um dann in die Zellen einzudringen und sie zu infizieren. Das macht die RBD zu einem Hauptangriffspunkt für Antikörper, die unser Körper erzeugt, um sich gegen das Virus zu verteidigen. Abbildung 1.
| Abbildung 1. Die Rezeptorbindungsdomäne (grau) des Spike-Proteins mit Regionen, an die 4 unterschiedliche Antikörpertypen binden. Kryoelektronenmikroskopische Untersuchungen. Mit Sars-CoV-2 (oder anderen Erregern) infizierte Menschen generieren Tausende unterschiedliche Antikörper um den Eindringling abzuwehren. (Bild aus F.S.Collins https://directorsblog.nih.gov/2020/12/03/caught-on-camera-neutralizing-antibodies-interacting-with-sars-cov-2/credit:Christopher Barnes, California Institute of Technology, Pasadena) |
Mutationen in der RBD…
Um herauszufinden, welche Mutationen einen positiven oder negativen Einfluss auf die Bindung der RBD an den Rezeptor ACE2 haben und /oder Antikörper daran hindern auf ihr Ziel am Spike-Protein zu treffen, haben die Forscher eine Methode namens Deep Mutational Scanning angewandt. Diese funktioniert folgendermaßen: Statt auf das Auftreten neuer Mutationen zu warten, haben die Forscher eine Bibliothek von RBD-Fragmenten generiert, von denen jedes eine Änderung in einem einzelnen Nukleotid-„Buchstaben“ enthielt und zum Austausch einer Aminosäure durch eine andere führte. Es stellt sich heraus, dass mehr als 3.800 solcher Mutationen möglich sind, und das Team von Bloom hat es geschafft, alle bis auf eine Handvoll dieser Versionen des RBD-Fragments herzustellen.
…und Auswirkungen auf Antikörper-Erkennung und -Bindung
Das Team hat dann mittels einer Standardmethode systematisch analysiert, wie jeder dieser Einzelbuchstaben die Fähigkeit von RBD veränderte, an ACE2 zu binden und menschliche Zellen zu infizieren. Sie haben auch bestimmt, wie sich diese Veränderungen auf die Erkennung und Bindung von drei verschiedenen therapeutischen Antikörpern an die virale RBD auswirkten. Zu diesen Antikörpern gehören zwei von Regeneron entwickelte Antikörper (REGN10933 und REGN10987), die gemeinsam als Cocktail die Genehmigung der Notfallanwendung zur Behandlung von COVID-19 erhalten hatten. Es wurde auch ein von Eli Lilly entwickelter Antikörper (LY-CoV016) untersucht, der sich derzeit in klinischen Phase-3-Studien zur Behandlung von COVID-19 befindet.
"Flucht"karten
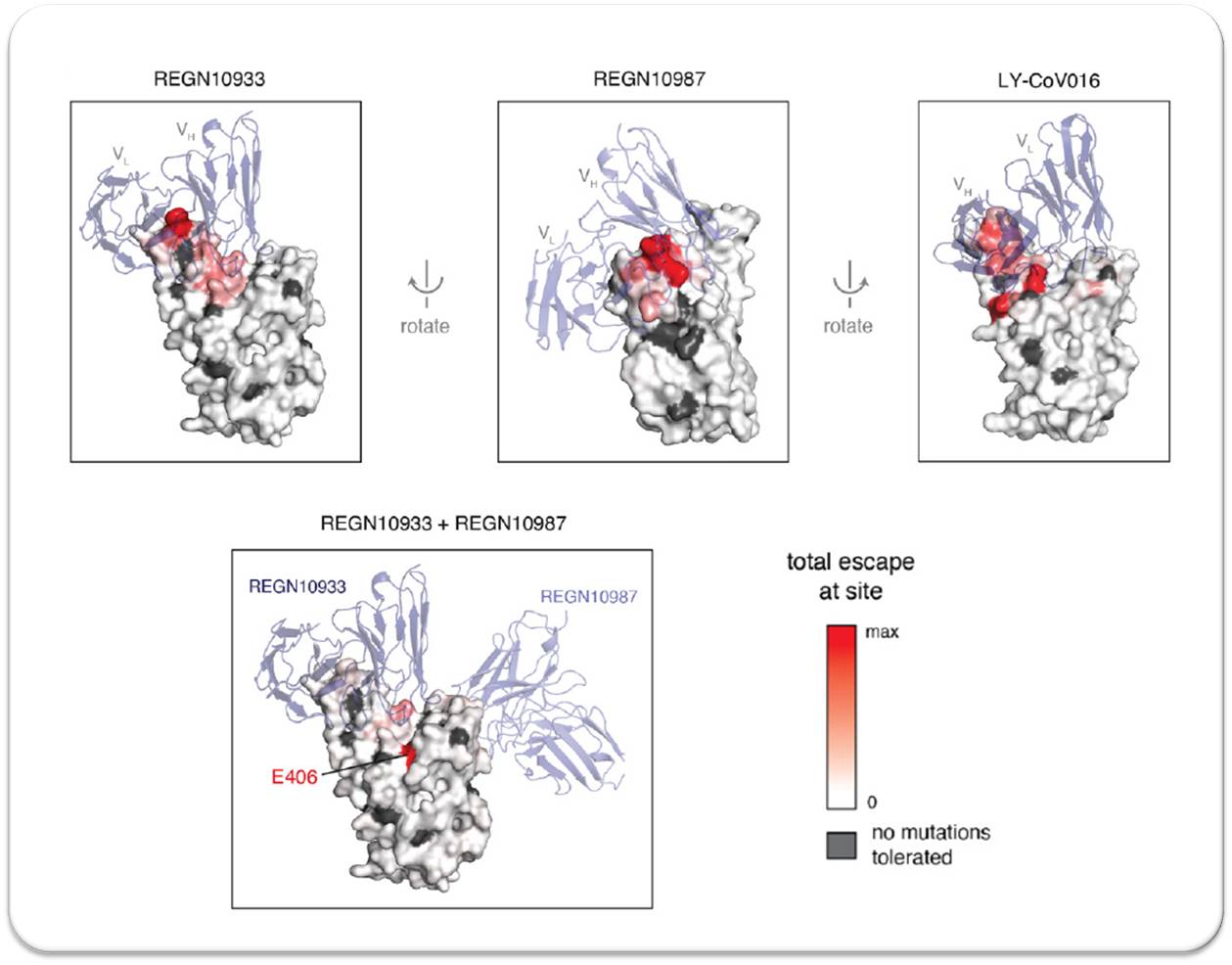
Basierend auf den Ergebnissen haben die Forscher vier Mutationskartierungen erstellt (Abbildung 2), die darstellen, wie SARS-CoV-2 jedem der drei therapeutischen Antikörper, sowie dem REGN-COV2-Cocktail entkommen kann. Die meisten Mutationen, die es SARS-CoV-2 ermöglichen würden, der Behandlung zu entkommen, waren bei beiden Regeneron-Antikörpern verschieden. Dies ist ermutigend, da es darauf hinweist, dass das Virus wahrscheinlich mehr als eine Mutation benötigt, um gegen den REGN-COV2-Cocktail resistent zu werden. Es scheint jedoch einen Punkt zu geben, an dem eine einzelne Mutation es dem Virus ermöglichen könnte, der REGN-COV2-Behandlung zu widerstehen.
| Abbildung. Die Rezeptorbindungsdomäne des Spike-Proteins, an die Antikörper (blass lila) gebunden sind. Diese „Fluchtkarte“ zeigt an, wo in der viralen RBD neue Mutationen die Antikörper am wahrscheinlichsten weniger wirksam machen (rot). Es zeigt auch Orte, an denen Mutationen die Antikörperbindung am wenigsten beeinflussen (weiß) und an denen Mutationen nicht fortbestehen können, weil sie die Funktionsfähigkeit der RBD beeinträchtigen würden (grau). (Bildnachweis: Nach TN Starr, Science, 2021 [1]). |
Die Fluchtkarte für LY-CoV016 zeigte ebenfalls eine Reihe von Mutationen, die es dem Virus ermöglichen könnten, zu entkommen. Während einige dieser Änderungen die Infektionsfähigkeit des Virus beeinträchtigen könnten, schienen die meisten von ihnen mit geringem bis keinem Nachteil für die Reproduktion des Virus verbunden zu sein.
Evolution des Virus bei langdauernder Antikörperbehandlung
In welcher Beziehung stehen diese Labordaten zur realen Welt? Um diese Frage zu untersuchen, haben sich die Forscher mit Jonathan Li (Brigham and Women's Hospital, Boston) zusammengetan. Sie haben den Fall eines immungeschwächten Patienten angesehen, der ungewöhnlich lange an COVID-19 litt und 145 Tage lang mit dem Regeneron-Cocktail behandelt wurde, was dem Virus Zeit gab sich zu replizieren und neue Mutationen zu erwerben.
Die viralen Genomdaten des infizierten Patienten zeigten, dass die Mutationskarten tatsächlich verwendet werden können, um wahrscheinliche Wege der viralen Evolution vorherzusagen. Im Verlauf der Antikörperbehandlung zeigte SARS-CoV-2 Veränderungen in der Häufigkeit von fünf Mutationen, welche die Konformation des Spike-Proteins und seiner RBD verändern dürften. Basierend auf den neu erstellten "Flucht"karten lässt sich erwarten, dass drei dieser fünf Mutationen die Wirksamkeit von REGN10933 verringern und eine der beiden anderen die Bindung an den anderen Antikörper, REGN10987.
Die Forscher haben auch Daten aller bekannten zirkulierenden SARS-CoV-2-Varianten (bis 11. Januar 2021) untersucht, um Hinweise auf "Flucht"mutationen zu erhalten. Sie fanden heraus, dass es insbesondere in Teilen Europas und Südafrikas bereits eine beträchtliche Anzahl von Mutationen gibt, die das Potential haben der Antikörperbehandlung zu entkommen.
Ausblick
Es ist allerdings zu beachten, dass die "Flucht"Karten nur drei wichtige Antikörperbehandlungen widerspiegeln. Nach Blooms Aussagen wird das Team weiterhin Mutationskarten für andere vielversprechende therapeutische Antikörper erstellen. Man wird auch weiterhin untersuchen, wo Veränderungen des Virus es ermöglichen könnten, den vielfältigeren Antikörpern zu entkommen, die unser Immunsystem nach einer COVID-19-Infektion oder -Impfung produziert.
Wenn es auch möglich ist, dass einige COVID-19-Impfstoffe weniger Schutz gegen einige dieser neuen Varianten bieten - und die jüngsten Ergebnisse deuten darauf hin, dass der AstraZeneca-Impfstoff möglicherweise nicht viel Schutz gegen die südafrikanische Variante bietet -, geben die meisten anderen aktuellen Impfstoffe immer noch genügend Schutz, um einen schweren Verlauf, eine Krankenhauseinweisung und den Tod zu verhindern. Um SARS-CoV-2 daran zu hindern, dass es neue Wege findet, auf denen es unseren laufenden Anstrengungen zur Beendigung dieser schrecklichen Pandemie entkommt, besteht der beste Weg darin , alles zu verdoppeln, was wir tun können, um zu verhindern, dass sich das Virus überhaupt vermehrt und verbreitet.
Jetzt sollte uns alle das Auftreten dieser neuen Varianten anspornen, Schritte zu unternehmen, um die Ausbreitung von SARS-CoV-2 zu verlangsamen. Das bedeutet: eine Maske tragen, auf die Distanz achten, die Hände häufig waschen. Es bedeutet auch, die Ärmel hochzukrempeln, um sich impfen zu lassen, sobald sich die Gelegenheit ergibt.
[1] Prospective mapping of viral mutations that escape antibodies used to treat COVID-19. Starr TN, Greaney AJ, Addetia A, Hannon WW, Choudhary MC, Dingens AS, Li JZ, Bloom JD. Science. 2021 Jan 25:eabf9302.
*Dieser Artikel von NIH Director Francis S. Collins, M.D., Ph.D. erschien zuerst (am 9. Feber 2021) im NIH Director’s Blog unter dem Titel: "Mapping Which Coronavirus Variants Will Resist Antibody Treatments " https://directorsblog.nih.gov/2021/02/09/mapping-which-coronavirus-variants-will-resist-antibody-treatments/. Der Artikel wurde von der Redaktion möglichst wortgetreu übersetzt und geringfügig (mit einigen Untertiteln) für den Blog adaptiert. Abbildung 1 stammt aus einem früheren Artikel von FS Collins (zitiert), Abbildung 2 aus [1]. Reprinted (and translated by ScienceBlog) with permission from the National Institutes of Health (NIH).
Artikel von Francis S. Collins zu COVID-19 im ScienceBlog
- 14.01.2021: Näher betrachtet: Auswirkungen von COVID-19 auf das Gehirn
- 22.10.2020: Schützende Antikörper bleiben nach überstandener SARS-CoV-2-Infektion monatelang bestehen
- 16.07.2020: Fortschritte auf dem Weg zu einem sicheren und wirksamen Coronaimpfstoff - Gepräch mit dem Leiter der NIH-COVID-19 Vakzine Entwicklung
- 07.05.2020: Die wichtigsten zellulären Ziele für das neuartige Coronavirus
- 16.04.2020: Können Smartphone-Apps helfen Pandemien zu besiegen?
- 05.03.2020: Strukturbiologie weist den Weg zu einem Coronavirus-Impfstoff
Bloom Lab ((Fred Hutchinson Cancer Center, Seattle) https://research.fredhutch.org/bloom/en.html
- Printer-friendly version
- Log in to post comments

Comments
Ein sehr interessanter…
Ein sehr interessanter Beitrag. Ich gehe davon aus, dass die diskutierten Arbeiten auch Ausgangspunkt für die Herstellung veränderter (an Mutanten angepasster) Impfstoffe sein wird.