Fr, 03.01.2014 - 06:36 — Peter Schuster
![]()
 Die Datenflut, die heute in den Naturwissenschaften erhoben wird, ist so gewaltig, daß sie mit dem menschlichen Auge nicht mehr erfasst, mit dem menschlichen Gehirn nicht mehr analysiert werden kann. Die Bioinformatik erstellt hier effiziente Computerprogramme, welche vor allem für den Fortschritt in den molekularen Lebenswissenschaften unabdingbar sind, jedoch kaum entsprechend gewürdigt werden.
Die Datenflut, die heute in den Naturwissenschaften erhoben wird, ist so gewaltig, daß sie mit dem menschlichen Auge nicht mehr erfasst, mit dem menschlichen Gehirn nicht mehr analysiert werden kann. Die Bioinformatik erstellt hier effiziente Computerprogramme, welche vor allem für den Fortschritt in den molekularen Lebenswissenschaften unabdingbar sind, jedoch kaum entsprechend gewürdigt werden.
(Bio)informatik ist aus den modernen Naturwissensschaften nicht mehr wegzudenken: Der Experimentator erhält Unmengen an Daten, die jedem Versuch einer direkten Betrachtung trotzen und nur mit Hilfe extensiver Computerprogramme bearbeitet werden können. Aber auch mathematische Beweise können häufig so komplex sein, daß sie zumindest teilweise durch den Computer ausgeführt werden. Diese Abhängigkeit führt zwangsläufig zur Frage:
Inwieweit können wir unseren Computern trauen?
Ist die umfangreiche Software, die auf unseren riesigen Maschinen läuft, fehlerfrei - frei von „Bugs“?
Vertreter unterschiedlicher Disziplinen der Naturwissenschaften und der, auch für diese essentiellen, Mathematik reagieren auf diese Fragen in unterschiedlicher Weise:
Mathematiker, die Puristen sind, akzeptieren nur sehr zögerlich Beweise, die mittels Computer erhoben wurden. Theoretische Physiker stehen dagegen derartigen Beweisen sehr offen gegenüber und vertrauen im Allgemeinen ihren gigantischen Maschinen. Den Chemikern sind Computermethoden in ihrem Fach geläufig, ihr Widerstand gegen theoretische Modelle schwindet in zunehmendem Maße. Biologen, schlussendlich, können in den modernen molekularen Fachrichtungen nichts ausrichten, ohne auf eine sehr eindrucksvolle Palette an Hilfsmitteln aus der Bioinformatik zuzugreifen.
Sisyphos und die Beweisbarkeit mathematischer Lehrsätze
Computerbeweise für bereits existierende Theoreme datieren in die 1950er Jahre zurück und gelten allgemein als brauchbare Verfahren in der reinen und angewandten Mathematik. Computerbeweise für noch offene Vermutungen spalten allerdings den Kreis der Mathematiker: die Verfechter dieses Verfahrens argumentieren damit, daß konventionelle Beweise für viele Theoreme derart komplex werden, daß sie mit dem menschlichen Gehirn - auf sich allein gestellt - nicht gefunden werden. Automatisierte Verfahren seien dagegen billig und hätten bei vielen, mit konventionellen Methoden bewiesenen Theoremen bereits Erfolg gezeigt.
Die Gegner argumentieren nicht weniger überzeugend: Um für den menschlichen Verstand begreifbar zu sein, muß die Beweisführung in eine Reihe von logischen Schlussfolgerungen unterteilt werden. Wird deren Zahl so hoch, daß der Beweis nicht in einigermaßen absehbarer Zeit verständlich gemacht werden kann, ist der Beweis zu verwerfen. Dies ist der Fall für viele der noch zu lösenden Aufgaben. Als Beispiel wird hier die „Keplersche Vermutung“ zur räumlich dichtesten Anordnung von gleich großen Kugeln angeführt (Abbildung 1).
Zu der für jeden Obsthändler trivialen Anordnung in einem regelmäßigen Gitter (kubisch-flächenzentrierte Packung und hexagonale Packung) hatte Johannes Kepler keinen mathematischen Beweis geliefert. Um einen derartigen Beweis zu führen und die enorme Zahl unterschiedlicher unregelmäßiger Anordnungen ausschließen zu können, bedarf es enormer Rechnerleistungen. Ein Beweis, den Thomas Hales und Sam Ferguson auf der Basis von drei Gigabyte an gespeicherten Computerprogrammen und Daten erhoben, wurde nach 5-jahrelanger Prüfung von den Begutachtern im Jahre 2003 als „zu 99 % korrekt“ eingestuft [1] .
Dies ist für einen mathematischen Beweis eindeutig zu wenig.
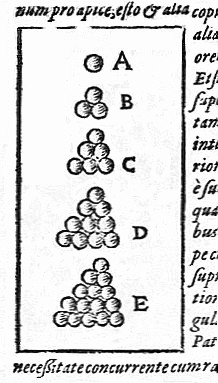 Abbildung 1. Vermutung zur dichtesten Packung von gleich großen Kugeln in einem regelmäßigen Gitter. Johannes Kepler (1611, Strena seu de nive sexangula – Über die sechseckige Schneeflocke)
Abbildung 1. Vermutung zur dichtesten Packung von gleich großen Kugeln in einem regelmäßigen Gitter. Johannes Kepler (1611, Strena seu de nive sexangula – Über die sechseckige Schneeflocke)
Abgesehen von den an Sisyphos erinnernden Anstrengungen der Beweisführung eines derart komplexen Problems, besteht hier Grund zur Skepsis und zwar auch hinsichtlich der Fehlerfreiheit von Hardware und Software. Thomas Hales sagt dazu: „Ich bin zu dem Schluß gekommen, daß kein Mikroprozessor jemals perfekt ist, daß diese sich der Perfektion aber immer mehr nähern“ und an einer anderen Stelle: „Bestehen Sie nicht darauf, daß jeder Fehler entfernt wird…Wenn der Programmierer einen kleineren Fehler entfernt, kann er dabei einen wesentlich schwerer wiegenden Fehler erzeugen“.
Um es kurz zu fassen: automatisierte Beweisführungen werden in Zukunft immer wichtiger, Computer-Wissenschafter müssen aber noch jede Menge „Hirnschmalz“ einsetzen, um ihre Maschinen und Programme verlässlicher zu machen. (Man sollte freilich auch nicht vergessen, daß nichts in unserer begrenzten Welt vollkommen fehlerfrei abläuft, wenn es nur ausreichend komplex ist.)
Computeranwendungen in Physik und Chemie
Physiker ebenso wie Chemiker wenden riesige Computerprogramme an, wenn sie u.a. in der Hochenergiephysik Daten sammeln und interpretieren, molekulare Strukturen mittels quantenmechanischer Modelle ermitteln oder sehr umfangreiche Simulierungen stochastischer (auf Wahrscheinlichkeitstheorie und Statistik basierender) Vorgänge beschreiben.
„Absolut fehlerfreie Programme“ sind hier - im Gegensatz zu den mathematischen Anwendungen – keine zentrale Forderung und der Grund dafür ist leicht verständlich: Das Ziel der Berechnungen in der Physik und Chemie ist es Daten zu produzieren, deren Fehlerbreite bloß erheblich geringer sein muß, als die der zugrunde liegenden, experimentell erhobenen Daten. Im Wesentlichen ist es daher nur erforderlich die Software auf Widersprüche zu testen und gröbere Fehler in den Programmen zu beseitigen.
Dazu kommt, daß numerische Mathematiker immer bessere Algorithmen generieren, Computer Spezialisten immer effizientere Programme. Die Geschwindigkeit der Rechner und ihre digitalen Speicherkapazitäten steigen ohne Unterbrechung seit den 1960-Jahren exponentiell – mit einer Verdopplungszeit von 18 Monaten – an. Dieses enorme Wachstum der Computerleistung wird aber von der Effizienzsteigerung der Algorithmen noch in den Schatten gestellt. Ein spektakuläres Beispiel dafür wurde in einem früheren Essay zitiert [2]: Wären zur Planung eines Produktionsablaufes im Jahr 1988 mit den damaligen Methoden 82 Jahre vonnöten gewesen, so war der Zeitbedarf im Jahr 2003 bereits auf 1 Minute abgesunken. Zu dieser insgesamt 43-millionenfachen Effizienzsteigerung trug die erhöhte Computerleistung mit einem Faktor 1000 bei, die verbesserten Algorithmen mit einem Faktor 43000.
Computeranwendungen in der Biologie
Die Biologie befindet sich in einer speziellen Situation, da weder traditionelle Fachrichtungen noch die frühe Molekularbiologie einer Unterstützung durch Computerwissenschaften bedurften. Die klassische Biologie hatte ja kaum irgendwelche mathematischen Grundlagen, Darwin’s Buch des Jahrhunderts „Ursprung der Arten“ enthält keine einzige Formel.
Sequenzierungen
Die zweite Hälfte des zwanzigsten Jahrhunderts brachte eine vollständige Änderung der Situation: neue Methoden der DNA-Sequenzierung erlaubten eine Aufklärung ganzer Genome in immer kürzerer Zeit.
Erschien die „händische“ Aufklärung der kleinen Genome von Viroiden und Viren zwar ziemlich mühsam, aber immerhin noch möglich, so war dies bei großen, aus Milliarden von Nukleotid-Bausteinen bestehenden, Genomen (wie z.B im Human Genome Project) ausgeschlossen. Zum Glück gab es ab 1970 bereits Algorithmen, die für Sequenzvergleiche erarbeitet worden waren. Diese dienten zwar zum Vergleich von Proteinsequenzen zur Erstellung von phylogenetischen Stammbäumen, konnten aber in Varianten für DNA-Sequenzen adaptiert werden. Die ununterbrochene Verbesserung der Algorithmen führte zu einer Reihe von Software-Paketen, ohne die ein modernes biologisches Labor nicht mehr auskommt, wie beispielsweise das weltweit angewandte, äußerst schnell arbeitende Programm BLAST (Basic Local Alignment Search Tool), das experimentell ermittelte DNA- oder Protein-Sequenzen mit den, in einer Datenbank bereits gespeicherten Sequenzen vergleicht und u.a. zur Auffindung homologer Gene/Proteine verwendet wird.
Analyse und Vorhersage von Struktur und Funktion
Enorm wichtige Programmentwicklungen befassen sich mit der Analyse der 3D-Strukturen von Biomolekülen, der Vorhersage dieser Strukturen auf Basis der Sequenzdaten und der Zuordnung von Funktionen zu einzelnen Strukturelementen: Die Entdeckung konservierter Strukturelemente in Teilstücken der DNA oder RNA deutet auf eine biologische Funktion dieser Regionen hin. Das aktuell von einem riesigen internationalen Team von mehr als 400 Forschern bearbeitete ENCODE („Encyclopedia of DNA Elements)-Projekt hat sich die systematische Erkundung aller funktionellen Elemente der DNA des humanen Genoms und anderer Aspekte seiner Organisation zum Ziel gesetzt [3]. Das bis jetzt erstaunlichste Ergebnis von ENCODE ist, daß rund 80 % der menschlichen DNA aktiv sind und nicht, wie bisher angenommen, nur die rund 2 % der Protein-codierenden Gene. Alle Daten aus ENCODE sind übrigens - zusammen mit den Software-Tools - frei abrufbar und anwendbar.
Weitere Anwendungen
Von der Vielzahl revolutionärer neuer Technologien, welche die Anwendung hocheffizienter Computerprogramme erfordern, sollen hier nur zwei aktuelle Verfahren angeführt werden: i) Das High-Throughput Screening (d.i. das Testen mit sehr hohem Durchsatz) und ii) das Modellieren ganzer großer Systeme.
High-Throughput Screening wird heute erfolgreich für biologische Fragestellungen und insbesondere in der medizinisch-, pharmazeutischen Forschung zur Auffindung neuer Wirkstoffe angewandt. Es erlaubt die gleichzeitige Testung von bis zu einer Million von Verbindungen hinsichtlich ihrer Wechselwirkungen mit Biomolekülen, Zellbestandteilen und auch intakten Zellen. Die gesamte Prozedur läuft vom Ansatz, über die Ausführung bis hin zur Auswertung der Ergebnisse vollkommen automatisch ab, gesteuert und analysiert von hocheffizienten Computerprogrammen.
Eine immens hohe Datenflut generiert das Modellieren ganzer biologischer Systeme – die Systembiologie. Bereits einzelne Zellen enthalten Tausende unterschiedliche, aktive Biomoleküle, die laufend miteinander reagieren, verstoffwechseln und verstoffwechselt werden. Die Modellierung derartig hochkomplexer Systeme - intakter Zellen, Organe bis hin zu lebenden Organismen – gehört zu den größten Herausforderungen in der näheren Zukunft.
Aus den enormen Datenmengen, die in Datenbanken zumeist frei verfügbar ruhen, kann – wie der berühmte Molekularbiologie Sidney Brenner meint - schlussendlich eine neue theoretische Biologie entstehen. Um hier relevantes Wissen aus Bergen von wenig informativem Material zu extrahieren, bedarf es einer soliden Grundlage in Mathematik und Computerwissenschaften.
Zur Wertschätzung der Computerwissenschafter
Im Titel dieses Essays hatte ich für die Vertreter dieser Fachrichtung, die Metapher „Marketender“ gebraucht. Dieser Begriff kommt aus dem mittelalterlichen Militärwesen und bezeichnet Personen, welche die Truppen begleiteten und für die militärische Logistik unabdingbar waren, da sie die Soldaten mit Gebrauchsgegenständen, Lebensmitteln und anderem Bedarf versorgten (Abbildung 2). Ihre Bedeutung ergab sich auch aus ihrer Unabhängigkeit, da sie nicht der Bürokratie des Heeres unterstellt waren und daher auf aktuelle Bedürfnisse sofort reagieren konnten.
Trotz ihrer Wichtigkeit für das Funktionieren eines effizienten Heeres, waren Marketender aber wenig geachtet.
 Abbildung 2. Marketender bietet seine Waren an (Holzstich aus 1516. Bild: Wikipedia)
Abbildung 2. Marketender bietet seine Waren an (Holzstich aus 1516. Bild: Wikipedia)
Die Analogie zu Computerwissenschaftern ist augenscheinlich, vor allem in der Biologie: Bioinformatiker sind für den Fortschritt biologischer Fachrichtungen essentiell, bei der Anerkennung der Erfolge werden sie aber gerne übersehen. Craig Venter, beispielsweise, wurde für seine Arbeiten zu Genomanalysen und Genomsynthesen weltberühmt – vollkommen zu Recht. Wer aber kennt die Namen der Computerwissenschafter, welche die, für diese Projekte essentiellen Programme zu Versuchsaufbau, Versuchsführung, Analyse und Interpretation erstellten?
In früheren Zeiten, als jeder Experimentator Versuche selbst aufbaute und Ergebnisse auch selbst interpretierte, war es nur selbstverständlich, daß er allein auch entsprechend gewürdigt wurde. Heute übernehmen Computerprogramme mehr und mehr an Verantwortung für Versuchsführung und Analyse. Es erscheint angebracht die Bedeutung der experimentellen in Relation zur rechnerischern Arbeit neu zu bewerten.
[1] ] Hales, T. C. A proof of the Kepler conjecture. Annals of Mathematics 2005, 162, 1065-1185.
[2] Peter Schuster: Wie Computermethoden die Forschung in den Naturwissenschaften verändern
[3] Das ENCODE Project; siehe auch den SB-Artikel Zentralismus und Komplexität.
Der vorliegende Essay ist eine stark verkürzte deutsche Fassung des Artikels „Are computer scientists the sutlers of modern biology?“, der eine komplette Liste von Literaturzitaten enthält und in Kürze von der Webseite des Autors abgerufen werden kann.
Weiterführende Links
Matthias Rarey: An der Schnittstelle: Informatik trifft Naturwissenschaften ( Zentrum für Bioinformatik Hamburg (ZBH); Universität Hamburg). Sehr leicht verständliches Video (als Werbung für ein Bioinformatik Studium gedacht) 1:09:21 h.
- Printer-friendly version
- Log in to post comments
