Fr, 11.08.2023 — Redaktion

![]() Das menschliche Genom kodiert für etwa 20 000 Protein. Viele dieser Proteine sind noch nicht charakterisiert und ihre Funktion ist unbekannt. Der Schwerpunkt der Forschung liegt auf bereits gut untersuchten Proteinen; dies gibt zur Besorgnis Anlass, dass in den vernachlässigten Teilen des Genoms/Proteoms wichtige biologische Prozesse kodiert sind. Um dem entgegenzuwirken, hat ein britisches Forscherteam eine "Unknome-Datenbank" entwickelt, in der die Proteine danach geordnet sind, wie wenig über sie bekannt ist. Die Datenbank soll die Auswahl bislang schlecht oder gar nicht charakterisierter Proteine von Menschen oder Modellorganismen erleichtern, damit sie gezielt untersucht werden können.
Das menschliche Genom kodiert für etwa 20 000 Protein. Viele dieser Proteine sind noch nicht charakterisiert und ihre Funktion ist unbekannt. Der Schwerpunkt der Forschung liegt auf bereits gut untersuchten Proteinen; dies gibt zur Besorgnis Anlass, dass in den vernachlässigten Teilen des Genoms/Proteoms wichtige biologische Prozesse kodiert sind. Um dem entgegenzuwirken, hat ein britisches Forscherteam eine "Unknome-Datenbank" entwickelt, in der die Proteine danach geordnet sind, wie wenig über sie bekannt ist. Die Datenbank soll die Auswahl bislang schlecht oder gar nicht charakterisierter Proteine von Menschen oder Modellorganismen erleichtern, damit sie gezielt untersucht werden können.
Das Human Genome Project
Mit der (nahezu) vollständigen Sequenzierung des menschlichen Genoms vor 20 Jahren ist zweifellos eine neue Ära der Biowissenschaften angebrochen. Tausende Wissenschafter, die rund um den Globus am Human Genome Project beteiligt waren, konnten einen lange angezweifelten Erfolg von "Big Science" feiern und eröffneten ein Eldorado an genetischen Informationen, das für jeden Interessierten frei zugänglich ist. Forscher wie Geldgeber (aus staatlichen Organisationen und Industrie) gingen davon aus, dass man nun schnell zu einem neuen Verständnis der Biologie des Menschen und seiner Krankheiten gelangen würde. Man erhoffte so in wenigen Jahren viele neue Zielstrukturen (Targets), die zumeist Proteine sind, für die Entwicklung erfolgversprechender Medikamente zu entdecken. Um den damaligen US-Präsidenten Bill Clinton anlässlich der Präsentation des ersten drafts des Human Genome Projects im Juni 2000 zu zitieren "werde das Projekt die Diagnose, Prävention und Therapie der meisten, wenn nicht aller Erkrankungen des Menschen revolutionieren “ und als eine wesentliche Aufgabe sah Clinton: "wir müssen die Fülle von Genomdaten durchforsten, um jedes menschliche Gen zu identifizieren. Wir müssen die Funktion dieser Gene und ihrer Proteinprodukte herausfinden, und dann müssen wir dieses Wissen schnell in Behandlungen umsetzen, die das Leben verlängern und bereichern können" [1].
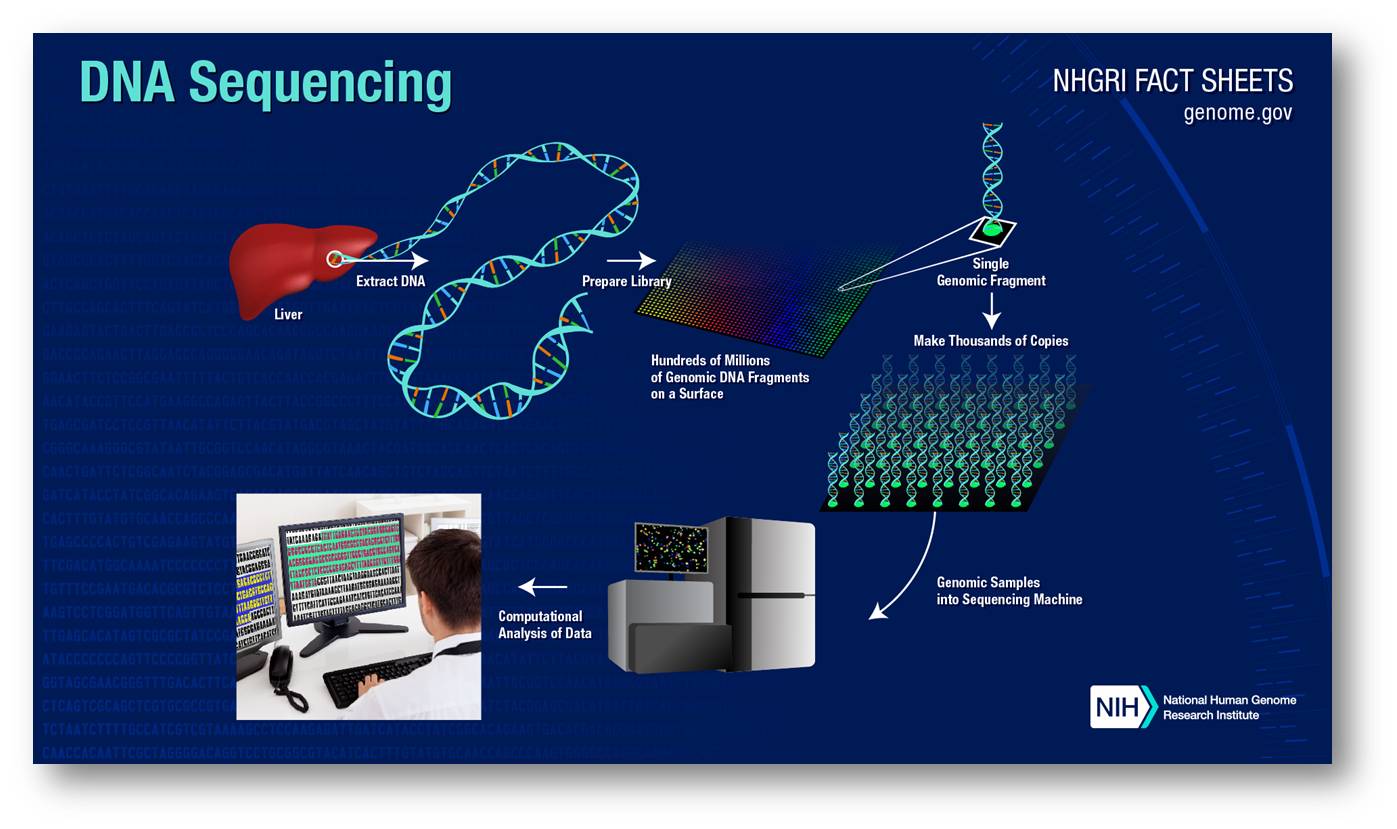
20 Jahre später sind auch die letzten Lücken im menschlichen Genom geschlossen. Mit neuen Technologien, die einen Preissturz der Sequenzierungen herbeiführten und sie beinahe schon für jeden erschwinglich machten, wurden bislang die Genome von mehr als einer Million Menschen sequenziert. Abbildung 1 zeigt ein vereinfachtes Schema der Genomsequenzierung.
| Abbildung 1 Schema der Gensequenzierung. Francis S. Collins, berühmter Pionier der Genforschung und damals Direktor des National Human Genome Research Institute (NHGRI) der NIH war de facto Leiter des aus Tausenden Forschern bestehenden International Human Genome Sequencing Consortium im Humangenomprojekt. Das NHGRI spielte darin eine wichtige Rolle Viele der neuen Technologien wurden im Rahmen und mit Unterstützung des Genomtechnologie-Programms des NHGRI entwickelt. (Bild: https://www.genome.gov/about-genomics/fact-sheets/DNA-Sequencing-Fact-Sheet; cc 0) |
Es wurden bislang zahlreiche Gene identifiziert , die mit Krankheiten assoziiert sind; dies kann frühe Diagnosen ermöglichen, bevor noch klinische Symptome auftreten. Zu den wichtigsten Erkenntnissen der Genomanalyse zählen zweifellos die Variationen in unseren Genen, die jeden von uns zu einem unterschiedlichen Individuum machen und sich auch auf unseren gesundheitlichen Status und auf das Krankheitsrisiko auswirken. In Folge ist eine Personalisierte Medizin entstanden, die diese Informationen in gezielte individuelle Behandlungen umsetzen möchte. (Basierend auf dem Genom eines Patienten können beispielsweise die für ihn geeignetsten - d.i. wirksamsten, nebenwirkungsärmsten - Medikamente ermittelt werden.)
Viele Gene und Proteine liegen noch im Dunkeln
Die Kenntnis aller Gensequenzen hat allerdings nicht zu einem wesentlichen Anstieg der Entdeckungsrate neuer Genfunktionen geführt, diese ist seit 2000 sogar noch zurück gegangen [2]. Es gibt einen hohen Anteil an Genen, deren Funktion unzureichend oder noch gar nicht charakterisiert ist. Dies gilt auch für die Charakterisierung der Proteine. Insgesamt enthält das Humangenom den Bauplan (d.h. es kodiert) für etwa 20.000 Proteine, Tausende davon waren zuvor in biochemischen und/oder genetischen Studien noch nicht identifiziert worden. Für viele dieser neuen Proteine ist auch 20 Jahre nach ihrer Entdeckung die Funktion noch unbekannt.
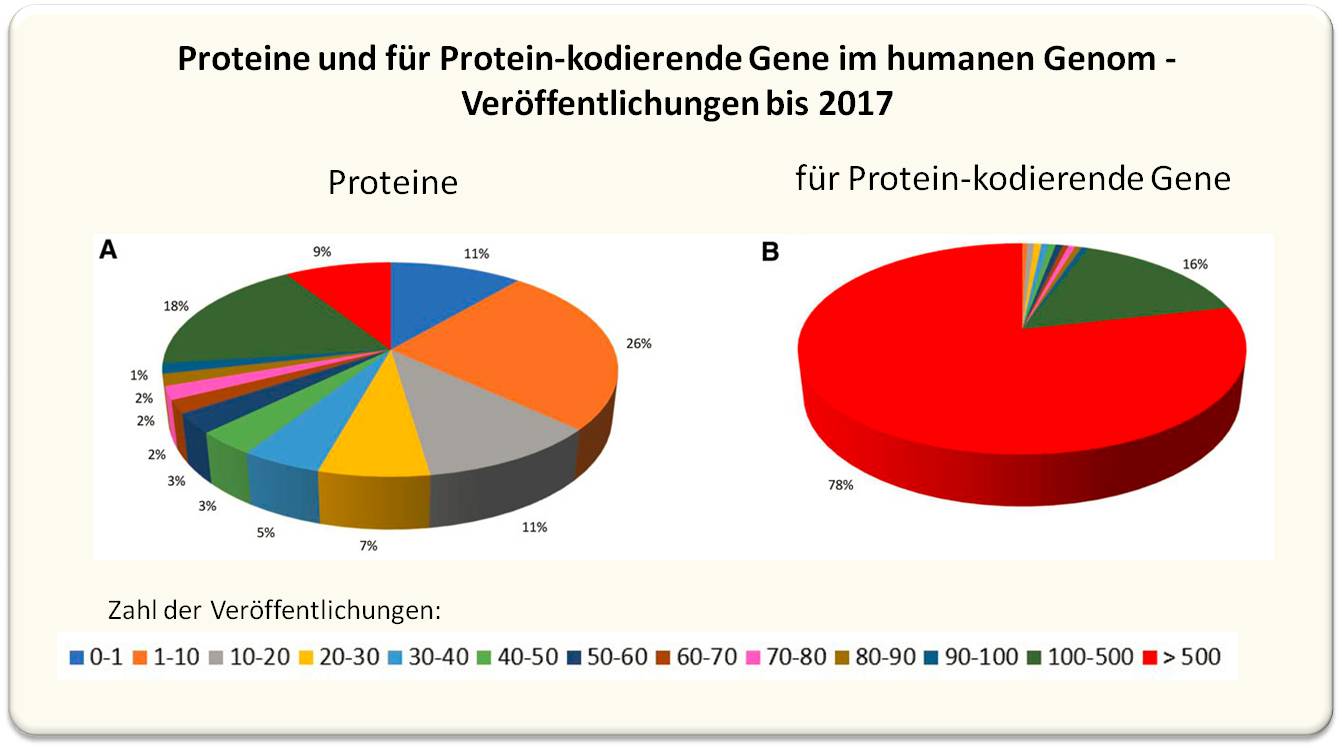
Ein rezenter Überblick über die biomedizinische Literatur bis 2017 zeigt, dass von rund 2000 Proteinen praktisch noch keine Informationen vorliegen und von rund 4600 Proteinen nur spärliche (d.i. 1 bis 10) Veröffentlichungen, dass also 37 % unserer Proteine nur unzureichend beschrieben sind [2]. Abbildung 2. Dagegen handelt es sich bei den am häufigsten - d.i. in mehr als 500 Veröffentlichungen - publizierten Genen und auch Proteinen um Biomoleküle, die ohnehin bereits gut charakterisiert sind.
Wie die Autoren des Übersichtartikels schlussfolgern "gibt es keine offensichtlichen wissenschaftlichen oder finanziellen Gründe für den Rückgang bei der Entdeckung biomolekularer Mechanismen; wahrscheinlich regen die derzeitigen Instrumente der Wissenschaftsförderung Forscher nicht dazu an oder entmutigen sie sogar sich mit den schwierigen Probleme der Entdeckung von Genfunktionen abzugeben."[2].
Natürlich ist auch eine gewisse Bequemlichkeit nicht von der Hand zu weisen. Der Ausflug ins Neuland erfordert ja proteinspezifische Reagenzien - Antikörper, Inhibitoren - und die Suche nach in vitro Systemen, die das neue Protein in ausreichendem Maße exprimieren.
| Abbildung 2. Status der bis 2017 vorhandenen Literatur zu Proteinen und für Proteine codierende Gene im humanen Genom. A) Anteil der Proteine, die in <1 bis >500 Veröffentlichungen beschrieben wurden. B) 94 % der gesamten Literatur über Protein-kodierende Gene bezieht sich auf solche, über die in mehr als 500, bzw. 100 bis 500 Arbeiten berichtet wurde. (Bild aus [2]: Sinha et al., Proteomics 18, 2018. https://doi.org/10.1002/pmic.201800093 PMID: 30265449. Lizenz: cc-by-nc.) |
Die Unknome Datenbank ...........
Die wissenschaftliche Forschung hat sich also bis jetzt auf gut untersuchte Proteine konzentriert und schlecht erforschte Gene mit möglicherweise wichtigen physiologischen Funktionen vernachlässigt. Dies hat zu der Besorgnis geführt, dass wichtige Grundlagen oder klinische Erkenntnisse sowie das Potenzial für therapeutische Interventionen unerkannt bleiben; mehrere Initiativen wurden daher gestartet, um das Problem anzugehen.
Eine dieser Initiativen ist die von Wissenschaftern in Großbritannien eingerichtete frei zugängliche, benutzergseteuerte Datenbank "Unknome" (zusammengesetzt aus "Unknown" und "Genome"), die anderen Wissenschaftern einen Anreiz geben soll , Licht in das Dunkel der unerforschten Gene/Genprodukte zu bringen (homepage: https://unknome.mrc-lmb.cam.ac.uk/about/) [3].
In der "Unknome"-Datenbank wird jedes Protein anhand eines "Bekanntheitsgrades" (Score) danach eingestuft, wie viel oder wie wenig die Wissenschaftler darüber - u.a. über Funktion, artenübergreifende Konservierung in Spezies, Lokalisierung in Zellen, etc. - wissen.
..............und ihre Brauchbarkeit zur Identifizierung von Funktionen bislang unbekannter Gene
Um die Brauchbarkeit von Unknome als Grundlage für experimentelle Arbeiten zu bewerten, haben die Forscher eine Reihe von 260 menschlichen Proteinen ausgewählt, die in orthologer Form (d.i. in hoch konservierter Basenabfolge) auch in dem Modellorganismus der Fliege Drosophila vorliegen, und deren Funktion in beiden Spezies noch unbekannt war (Score < 1) [3]. Um den Beitrag dieser orthologen Gene zu einem breiten Spektrum biologischer Prozesse zu testen, haben sie diese Gene nacheinander (mit Hilfe von RNA-Interferenz) (partiell) ausgeschaltet. Für 62 dieser Gene war ein kompletter Knockout mit dem Überleben der Fliege nicht vereinbar. Unter den restlichen nicht-essentiellen Genen wurden 59 entdeckt, die zu wichtigen biologischen Funktionen beitragen, darunter zu Fertilität, Entwicklung, Gewebewachstum, Qualitätskontrolle von Proteinen (Entfernung schadhafter Proteine), Widerstandsfähigkeit gegen Stress, Fortbewegung, Signalübertragung über den Notch-Signalweg [3].
Ob und welche Effekte die orthologen Gene beim Menschen haben, ist noch nicht untersucht. Jedenfalls kann aber gefolgert werden, dass in den bislang vernachlässigten Teilen des Genoms/Proteoms wichtige biologische Prozesse kodiert sind.
Fazit
Die Unknome-Datenbank ist eine Ressource für Forscher, welche die Chancen unerforschter Bereiche der Biologie nutzen wollen. In ihrer Studie zeigen die britischen Forscher auf, dass trotz jahrzehntelanger umfangreicher genetischer Untersuchungen es offensichtlich viele Fliegengene gibt, deren essentielle Funktionen noch unbekannt sind; dasselbe gilt auch für die orthologen Gene des Menschen. Die Forscher hoffen, dass sich diese Datenbank mit zunehmender Nutzung in den kommenden Jahren verkleinern und neue biologische und therapeutische Erkenntnisse liefern wird. Dabei ist nicht auszuschließen, dass man auf völlig neue Bereiche biologischer Funktionen stößt.
[1] June 2000 White House Event: https://www.genome.gov/10001356/june-2000-white-house-event
[2] S. Sinha et al., Darkness in the Human Gene and Protein Function Space: Widely Modest or Absent Illumination by the Life Science Literature and the Trend for Fewer Protein Function Discoveries Since 2000. Proteomics 2018, 18, 1800093. DOI: 10.1002/pmic.201800093
[3] Rocha JJ, et al. (2023) Functional unknomics: Systematic screening of conserved genes of unknown function. PLoS Biol 21(8): e3002222. https://doi.org/10.1371/journal.pbio.3002222
- Printer-friendly version
- Log in to post comments