Fr, 19.07.2013 -10:93 — Peter Schuster

 Themenschwerpunkt Synthetische Biologie
Themenschwerpunkt Synthetische Biologie
Können wir mit der Synthetischen Biologie etwas Besseres bewirken, als das, was Natur und Evolution im Laufe der Jahrmilliarden hervorgebracht haben? Der zweite Teil des Artikels handelt von der Schaffung neuartiger Strukturen, einerseits mit Methoden des Rationalen Design, andererseits mit Methoden, die nach den Prinzipien der biologischen Evolution – Variation und Selektion -arbeiten. Der Artikel basiert auf einem Vortrag des Autors anläßlich des Symposiums über Synthetische Biologie, das von der Österreichischen Akademie der Wissenschaften im Mai d.J. veranstaltet wurde und erscheint auf Grund seiner Länge in zwei aufeinander folgenden Teilen.
Zwei grundsätzlich unterschiedliche Strategien zur Erzeugung von Molekülen und Organismen mit vorbestimmten Eigenschaften stehen einander gegenüber:
- das rationale Design, welches auf unserem gesamten biologischen Wissen über Strukturen und Funktionen von Biomolekülen aufbaut, und
- das evolutionäre Design, das die Prinzipien der biologischen Evolution zur Selektion von Objekten mit gewünschten Eigenschaften anwendet.
Die Literatur zum Thema Design von Biomolekülen ist enorm umfangreich [1]. Wir müssen uns hier auf wenige Beispiele beschränken, welche die unterschiedliche Anwendbarkeit beider Strategien sowie ihre Stärken und Schwächen aufzeigen.
Rationales Design
Das rationale Design baut auf dem Paradigma der konventionellen theoretischen Strukturbiologie auf:  Bei bekannter Sequenz (= Primärstruktur, d.h. bekannter Abfolge von Aminosäuren in einem Protein, von Nukleotiden in einer Nukleinsäure) sollte die 3-dimensionale Struktur (= Tertiärstruktur) eines Moleküls vorhergesagt werden können, soferne die detaillierten Bedingungen bekannt sind, unter denen die Faltung erfolgt. Die aufgeklärte Struktur eines Moleküls erlaubt – so die Annahme der Strukturbiologie – Rückschlüsse auf die Funktion. Der Zusammenhang zwischen Primär-, Sekundär- und Tertiärstruktur eines Proteins ist in Abbildung 1 aufgezeigt.
Bei bekannter Sequenz (= Primärstruktur, d.h. bekannter Abfolge von Aminosäuren in einem Protein, von Nukleotiden in einer Nukleinsäure) sollte die 3-dimensionale Struktur (= Tertiärstruktur) eines Moleküls vorhergesagt werden können, soferne die detaillierten Bedingungen bekannt sind, unter denen die Faltung erfolgt. Die aufgeklärte Struktur eines Moleküls erlaubt – so die Annahme der Strukturbiologie – Rückschlüsse auf die Funktion. Der Zusammenhang zwischen Primär-, Sekundär- und Tertiärstruktur eines Proteins ist in Abbildung 1 aufgezeigt.
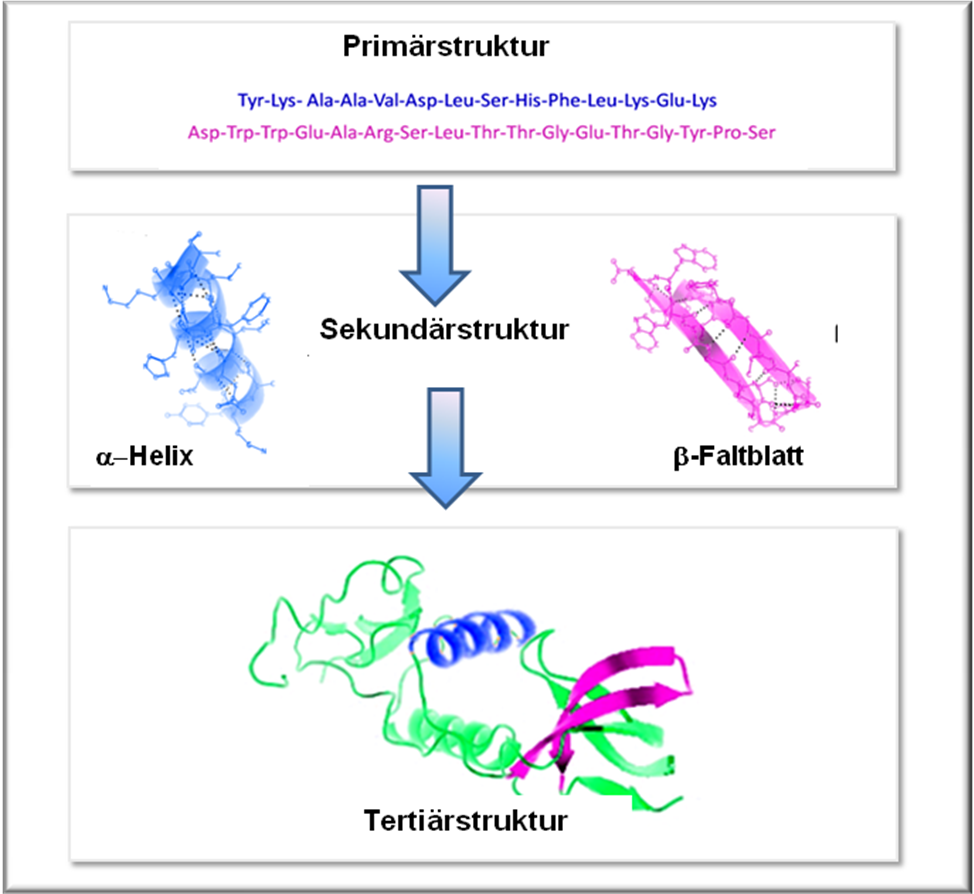 Abbildung 1. Hierarchischer Aufbau eines Proteins. Primärstruktur = Sequenz (Abfolge) der Aminosäuren in der Polypeptidkette, Sekundärstruktur: räumliche Anordnung von Abschnitten der Polypeptidkette (hier pink und blau); alpha-helix und beta-Faltblatt sind häufig auftretende Motive. Tertiärstruktur: übergeordnete 3D-Struktur aus den Sekundärstrukturelementen (modifiziert nach Wikimedia Commons).
Abbildung 1. Hierarchischer Aufbau eines Proteins. Primärstruktur = Sequenz (Abfolge) der Aminosäuren in der Polypeptidkette, Sekundärstruktur: räumliche Anordnung von Abschnitten der Polypeptidkette (hier pink und blau); alpha-helix und beta-Faltblatt sind häufig auftretende Motive. Tertiärstruktur: übergeordnete 3D-Struktur aus den Sekundärstrukturelementen (modifiziert nach Wikimedia Commons).
Rationales Design bietet den Vorteil einer direkten oder gezielten Suchstrategie und ist daher sowohl rasch als auch Material sparend. Sein Nachteil resultiert allerdings aus den zurzeit noch immer gegebenen Defiziten im Wissen um die Beziehung zwischen Strukturen und Funktionen von Biomolekülen: diese können nicht von „first principles“ aus berechnet werden, sondern benötigen möglichst viel empirischen Input, um einigermaßen verlässliche Vorhersagen zu ermöglichen.
In der Folge wird hier zuerst das Design von Enzymmolekülen erörtert, welches auch unter dem Namen „protein engineering“ bekannt ist, dann das Design von Ribonukleinsäuren (RNA-Molekülen).
Rationales Protein-Design
Voraussetzung für das rationale Protein-Design war die Entwicklung und Etablierung von Techniken, die es erlauben gezielt an jeder Position der Aminosäuresequenz (siehe Abbildung 1) jeden der zwanzig Aminosäurereste durch einen anderen zu ersetzen („site-directed“ Mutagenese). Anfänglich war das Protein-Design hauptsächlich mit der Analyse von Sequenz-Struktur Beziehungen befasst mit dem Ziel die Prinzipien der Proteinfaltung besser zu verstehen und stabile Strukturen möglichst verlässlich vorhersagen zu können. In den letzten Jahren des vorigen Jahrhunderts wurde eine neue, nun bereits gängige Vorgehensweise zu Vorhersage und Design von Proteinstrukturen eröffnet, der Computerrechnungen mit empirischen Daten verknüpft. Auf dieser Basis ist es gelungen, Enzyme durch gezielte Mutationen thermodynamisch stabiler zu machen, ohne deren enzymatische Aktivität zu verringern.
Ein prominentes Beispiel der technischen Verwertung von natürlichen und artifiziellen – designten – Enzymen findet sich in der Waschmittelindustrie. Der Gedanke Enzyme in Waschmitteln zu verwenden ist relativ alt. Bereits 1913 stellte der deutsche Pharmazeut, Chemiker und Unternehmer Otto Röhm einen Extrakt aus tierischen Bauspeicheldrüsen her, der Protein-spaltende Enzyme (Proteasen) enthielt, verwendete diesen für die Vorwäsche und erhielt auch ein Patent dafür. Wegen mangelnder Reinheit des Produktes und zu hohen Herstellungskosten war dem neuen Waschmittel allerdings kein Erfolg beschieden. Erst im Jahre 1959 wurde in der Schweiz das erste Waschmittel mit einer bakteriellen Protease eingeführt. Zehn Jahre später wurde die Verwendung von Enzymen allmählich populär und heute sind gentechnisch in Bakterien der Arten Bacillus licheniformis und Bacillus amyloliquefacies hergestellte Enzyme aus der Waschmittelindustrie nicht mehr wegzudenken. Etwa zwei Drittel der gesamten, für technische Verwendung produzierten Enzyme findet seinen Einsatz in Waschmitteln. Ein modernes Waschmittel für die Waschmaschine oder ein Geschirrspülmittel enthält Biomaterialien abbauende Enzyme aus vier Klassen: (i) Proteasen zur Spaltung von Proteinen, (ii) Amylasen für den Stärkeabbau, (iii) Lipasen für die Spaltung von Fettstoffen und (iv) Cellulasen für den oberflächlichen Abbau von Baumwollfasern, um die Gewebe weich zu erhalten. Protein-Design dient in erster Linie dazu, um die Enzyme stabiler zu machen und ihre Aktivität bei höheren (Wasch-)Temperaturen und alkalischen pH-Werten (Waschlauge) zu erhalten.
Rationales Design von Ribonukleinsäuren
Ribonukleinsäuren (RNA-Moleküle) gehören zu den versatilsten Molekülen der Biosphäre und kommen in einer Vielzahl unterschiedlicher Arten und Strukturen vor. Sie üben nicht nur Schlüsselfunktionen in der Regulation und Übertragung der in der DNA gespeicherten genetischen Information und der Synthese der Genprodukte – der Proteine – aus, sie können – in Form sogenannter Ribozyme – auch chemische Reaktionen katalysieren, sind dann also als Enzyme zu betrachten. Die Vielfalt an kürzlich entdeckten regulatorischen Funktionen läßt RNA-Moleküle als attraktive Zielmoleküle (Targets) für Therapeutika erscheinen, ebenso können sie aber auch selbst als hochspezifische Therapeutika und als Diagnostika Verwendung finden. Abbildung 2 zeigt Beispiele von Sequenz und Sekundärstruktur von RNA-Molekülen.
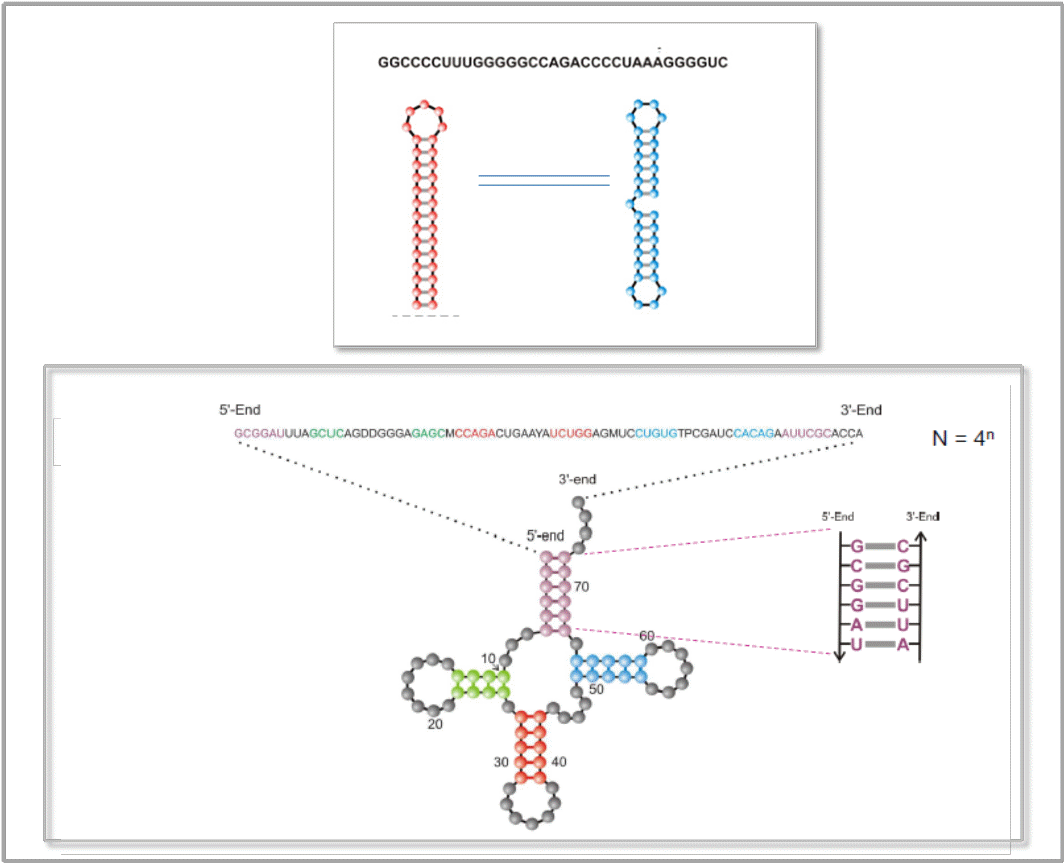 Abbildung 2. Sequenz und Sekundärstruktur von RNA-Molekülen. RNAs sind Polynukleotidketten, zusammengesetzt aus den Nukleotiden der Basen Guanin (G), Cytosin (C), Adenin (A) und Uracil (U). Durch Paarung der Basen C-G, G-U und A-U (durch Wasserstoffbrücken) entstehen „Stamm-Schleifen“-Strukturen („stem-loop“), d.i. doppelhelikale, gepaarte Teile („stem“) und Haarnadel-Schleifen (ungepaarte Nukleotide). Oben: ein aus 33 Nukleotiden bestehendes Molekül in seiner stabilen Sekundästruktur (rot) und eine seiner zahlreichen suboptimalen Strukturen (blau). Unten: Sequenz und stabilste Struktur eines Transfer-RNA-Moleküls (Phenylalanyl-tRNA)
Abbildung 2. Sequenz und Sekundärstruktur von RNA-Molekülen. RNAs sind Polynukleotidketten, zusammengesetzt aus den Nukleotiden der Basen Guanin (G), Cytosin (C), Adenin (A) und Uracil (U). Durch Paarung der Basen C-G, G-U und A-U (durch Wasserstoffbrücken) entstehen „Stamm-Schleifen“-Strukturen („stem-loop“), d.i. doppelhelikale, gepaarte Teile („stem“) und Haarnadel-Schleifen (ungepaarte Nukleotide). Oben: ein aus 33 Nukleotiden bestehendes Molekül in seiner stabilen Sekundästruktur (rot) und eine seiner zahlreichen suboptimalen Strukturen (blau). Unten: Sequenz und stabilste Struktur eines Transfer-RNA-Moleküls (Phenylalanyl-tRNA)
RNA-Moleküle eignen sich hervorragend für das rationale Design, da ihre Sekundärstrukturen einer rigorosen mathematischen Analyse zugänglich sind. In der Realität gibt es für RNA-Moleküle nicht nur eine einzige stabile Konformation - jeder Sequenz entspricht ein ganzes Spektrum von metastabilen suboptimalen Strukturen, welche sich hinsichtlich ihrer Lebensdauer unterscheiden. Die Zeit, die benötigt wird um von einer Konformation zur anderen zu gelangen, hängt von der Energiebarriere ab, die beim Übergang überwunden werden muß. Bei sehr hohen Energiebarrieren (Aufbrechen vieler Basenpaarungen) können aus einer Sequenz langlebige bistabile, multistabile Konformationen resultieren. Ein einfaches Beispiel zweier langlebiger RNA-Strukturen ist in Abbildung 2 (oben) gezeigt.
Für RNA-Moleküle ist es möglich Algorithmen zu entwickeln, die auch bistabile und multistabile RNA-Moleküle designen können. Alternative stabile Konformationen desselben RNA-Moleküls sind experimentell an vielen Beispielen nachgewiesen worden. Auch in der Natur sind RNA-Moleküle bekannt, die zwei Konformationen ausbilden, welche völlig unterschiedliche Funktionen besitzen können. Sie werden „Riboswitches“ genannt und regulieren (vor allem in Bakterien) u.a. die Synthese von Enzymen, die im Stoffwechsel eine Rolle spielen.
Evolutionäres Design
Das Darwinsche Prinzip der natürlichen Auslese baut auf drei Voraussetzungen auf: (i) Vermehrung durch Reproduktion, (ii) Variation und (iii) Selektion durch begrenzte Ressourcen. Keine dieser drei Voraussetzungen ist an zelluläres Leben gebunden und es ist daher zu erwarten, dass Darwinsche Evolution auch in zellfreien Systemen auftreten kann. Dies hat Sol Spiegelman schon in den Neunzehnhundertsechzigerjahren erkannt und die ersten erfolgreichen Versuche unternommen, Moleküle im Laborexperiment zu evolvieren.
Das Spiegelmansche Serial-Transfer Experiment
In diesem bahnbrechenden Experiment wurde RNA durch Replikation mit einer RNA-Polymerase kopiert und damit vermehrt. Variation kam durch fehlerhaftes Kopieren zustande, Selektion durch die Versuchsführung mittels „Serial-Transfer“: eine Lösung mit den Bausteinen (G, C, A, U) für die RNA-Synthese und der Polymerase im Reagenzglas A wurde mit einer kleinen Probe der zu kopierenden RNA versetzt, wobei sofort Replikation einsetzte. Nach einer bestimmten Zeitspanne (und dem Verbrauch der Bausteine in A) wurde eine kleine Probe in Reagenzglas B (ebenfalls in eine Lösung von Bausteinen und Polymerase) überimpft, wo erneut RNA-Replikation einsetzt, und dieser Vorgang etwa einhundert Mal wiederholt. Spiegelman beobachtete, dass im Laufe des Experiments die RNA-Synthesegeschwindigkeit zunahm und dass sich die RNA-Moleküle veränderten: durch Kopierfehler entstandene, rascher replizierende Moleküle verdrängten die ursprünglichen langsamer replizierenden Moleküle solange bis die Geschwindigkeit der Replikation einen maximalen Wert erreicht hatte. Derartige Experimente mit Molekülen unter Laborbedingungen stellen Evolution im Zeitraffer dar, da ein solches „Serial-Transfer“ Experiment in einem Tag ausgeführt werden kann.
Evolutionäre „Züchtung“ von Biomolekülen
Die Tatsache, dass Moleküle im Reagenzglasversuch im Darwinschen Sinne evolviert werden können, war der Anlass für die Entwicklung eines neuen Zweiges der Biotechnologie: das evolutionäre Design von Biomolekülen mit vorgegebenen Eigenschaften. Zum Unterschied vom rationalen Design ist es weder notwendig die molekularen Strukturen zu kennen noch muss man über die Beziehung zwischen Strukturen und Funktionen Bescheid wissen. Man benötigt lediglich ein Testsystem für die gewünschte Moleküleigenschaft und eine Selektionsmethode mit der man Moleküle, welche die Wunschvorstellungen am besten erfüllen, aus einer Mischung von Molekülen mit anderen Eigenschaften herausholen kann.
Am Anfang steht die Erzeugung einer Population von Molekülen mit hinreichend großer genetischer Vielfalt (beispielsweise mit Syntheseautomaten hergestellte Moleküle mit Zufallssequenzen). Mit Hilfe eines geeigneten Selektionsverfahrens wählt man die am besten geeigneten Moleküle aus und erzeugt durch Amplifikation und fehlerhafte Reproduktion eine neue Population, die nun wieder einer neuen Selektion unterworfen werden. Im Allgemeinen genügen zwanzig bis dreißig Selektionszyklen, um für den Verwendungszweck optimale Moleküle zu erhalten. Evolutionäre Methoden wurden für viele verschiedene Zwecke eingesetzt – wir erwähnen hier zwei davon: (i) die Züchtung von optimal bindenden RNA-Molekülen, sogenannten Aptameren und die gezielte Evolution von Proteinen.
Die SELEX-Methode
RNA-Moleküle sind für die Anwendung evolutionärer Methoden besonders gut geeignet, da sie unmittelbar repliziert und mutiert werden können. Ein typisches Beispiel ist die in Abbildung 3 dargestellte SELEX-Methode (Systematic Evolution of Ligands by EXponential Enrichment), welche heute routinemäßig eingesetzt wird, um Moleküle zu evolvieren, die an vorgegebene Zielstrukturen (targets) möglichst fest binden. Die Vorgangsweise lässt sich einfach beschreiben: Es wird zuerst ein „Pool“ an RNA-Sequenzen mit zufälliger Nukleotidabfolge angelegt und in eine geeignete Lösung transferiert und es wird eine Säule für die Affinitätschromatographie präpariert, welche die Zielmoleküle irreversibel an das Säulenmaterial gebunden enthält. Die Lösung wird über die Säule laufen gelassen und die am besten an die stationäre Phase bindenden Moleküle werden aus der Lösung entfernt. Mit einem anderen Lösungsmittel werden dann die an der Säule gebundenen Moleküle ausgewaschen und einem Selektionszyklus – Amplifikation, Diversifizierung und Selektion – unterworfen. Das Lösungsmittel wird von Zyklus zu Zyklus geändert, so dass es immer schwerer wird an die Zielmoleküle zu binden. Nach hinreichend vielen Zyklen erhält man optimal und äußerst fest an die Targets bindende Moleküle.
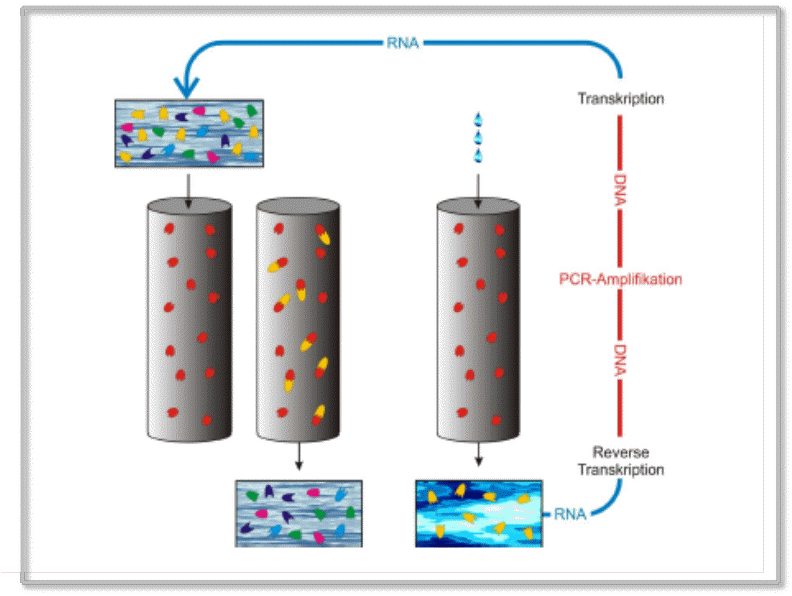 Abbildung 3. Die SELEX-Methode zur Erzeugung optimal an Zielstrukturen bindender RNA-Moleküle (Aptamere). Beispielsweise soll zur Blockierung eines therapeutischen Zielmoleküls (Target) ein möglichst spezifisches, festbindendes RNA-Molekül gezüchtet werden. Dazu wird eine Lösung mit einer Vielfalt an RNA-Molekülen (A) auf eine Trennsäule aufgebracht, welche das an das Trennmaterial fixierte Target-Molekül (rot) enthält (B). RNAs, die nicht an das Target-Molekül binden, werden nicht in der Säule festgehalten (C) und ausgewaschen. Festbindende RNAs (gelb) werden mit „schärferen“ Lösungsmitteln eluiert (E), mit klassischen Methoden in DNA umgeschrieben, diese mittels Polymerase-Kettenreaktion (PCR) amplifiziert und wieder in RNA umgeschrieben (F). Die so entstandene Lösung enthält ein konzentriertes Gemisch aus festbindenden RNAs, die nun wieder Selektionsprozessen unter immer verschärfteren Elutionsbedingungen unterworfen werden
Abbildung 3. Die SELEX-Methode zur Erzeugung optimal an Zielstrukturen bindender RNA-Moleküle (Aptamere). Beispielsweise soll zur Blockierung eines therapeutischen Zielmoleküls (Target) ein möglichst spezifisches, festbindendes RNA-Molekül gezüchtet werden. Dazu wird eine Lösung mit einer Vielfalt an RNA-Molekülen (A) auf eine Trennsäule aufgebracht, welche das an das Trennmaterial fixierte Target-Molekül (rot) enthält (B). RNAs, die nicht an das Target-Molekül binden, werden nicht in der Säule festgehalten (C) und ausgewaschen. Festbindende RNAs (gelb) werden mit „schärferen“ Lösungsmitteln eluiert (E), mit klassischen Methoden in DNA umgeschrieben, diese mittels Polymerase-Kettenreaktion (PCR) amplifiziert und wieder in RNA umgeschrieben (F). Die so entstandene Lösung enthält ein konzentriertes Gemisch aus festbindenden RNAs, die nun wieder Selektionsprozessen unter immer verschärfteren Elutionsbedingungen unterworfen werden
Gezielte Evolution von Proteinen
Diese verfolgt von Beginn an zwei verschiedene Ziele: i. ein besseres Verstehen der Stabilitäten und der Funktionen von Proteinen, welche in der natürlichen Evolution von vielen anderen und oft komplexen Bedingungen überlagert sind, und
ii. die Erzeugung von nichtnatürlichen Proteinen, welche ein Licht auf die physikalisch-chemischen Eigenschaften der Moleküle werfen, welche nicht von den Evolutionsbedingungen überschattet sind.
Insbesondere ist es bei in vitro Evolution möglich alle Zwischenstufen eines evolutionären Prozesses zu isolieren und zu studieren und damit einen sonst nicht erzielbaren Einblick zu gewinnen, auf welchen Wegen Populationen von Molekülen evolutionär optimiert werden.
Synthetische Biologie „quo vadis“?
Der Name „synthetische Biologie“ taucht erstmals im Jahre 1913 in einem Letter to Nature mit dem Titel: „Synthetic biology and the mechanism of life“ auf und bezieht sich auf ein „La biologie synthétique“ übertiteltes Buch von Stéphane Leduc. Der Autor, ein französischer Biologe, versuchte darin Lebensvorgänge auf die physikalische Chemie der Diffusion in flüssigen Lösungen zurückzuführen.
Meilensteine in der Entstehung der Synthetischen Biologie
Im vergangenen Jahrhundert hat die synthetische Biologie Gestalt angenommen – keine einheitliche aber eine, die auf dem Boden der molekularen Biologie und insbesondere der molekularen Genetik gewachsen ist. Als Meilensteine kann man nennen:
i. das Watson-Crick’sche Strukturmodell der DNA (Nature 1953, Nobelpreis für Physiologie 1962),
ii. die Entdeckung der Restriktionsnukleasen durch Werner Arber, Daniel Nathans und Hamilton Othanel Smith (Nobelpreis für Physiologie, 1978) und
iii. ihre Anwendung in der Molekulargenetik in Form der rekombinanten Klonierungstechnik durch Paul Berg (Nobelpreis für Chemie 1980),
iv. die Entwicklung neuer DNA-Sequenzierungmethoden, welche erstmals die Sequenzierung ganzer Genome ermöglichten, durch Walter Gilbert and Frederick Sanger (Nobelpreis für Chemie 1980),
v. die Herstellung eines künstlichen Oszillators, Repressilator genannt, in vivo durch Einschleusen drei Repressor-Genen in Escherichia coli oder der Einbau eines reversiblen genetischen Schalters in dasselbe Bakterium,
vi. die chemische Totalsynthese und der Einbau eines Genoms in eine Bakterienzelle.
Neue Anwendungen
In den letzten Jahren hat sich ein neuer obgleich naheliegender Anwendungsbereich für synthetische DNA aufgetan: die Verwendung als Speicher von digitaler Information. George Church und Mitarbeiter haben eine DNA synthetisiert, die ein ganzes Buch mit 53 426 Worten, 11 jpeg-Bildern und einem Java-Script auf einer kodierenden Länge von 5.27 MegaBit enthält. Damit hält diese Speicherung zurzeit den Rekord hinsichtlich der Informationsdichte von fast 1016 Bits pro mm3, und übertrifft damit alle physikalischen Speicher einschließlich der Quantenholographie.
Der Schlüssel zu einer neuen DNA-basierten Technologie ist die Synthese von DNA in ausreichend großen Mengen zu hinreichend niedrigen Kosten. In der Tat scheinen neue als „next-generation technology“ apostrophierten Methoden diese Möglichkeiten zu eröffnen. Die Wissenschaftler und Techniker der Firma Gen9 in Cambridge (MA) erklären, dass sie in einzigem Labor ebenso viel DNA synthetisieren können wie der Rest der Welt.
Die „American Chemical Society (ACS)“ hat vor drei Monaten ein Exposé mit dem Titel „Engineering for the 21st Century: Synthetic Biology“ mit den Worten geschlossen:
„For years, scientists have hoped that biology would find its engineering counterpart – a series of principles that could be used as reliably as chemical engineering is for chemistry. Thanks to major advances in synthetic biology, those hopes may soon be realized”.
Als eine solche Core-Technologie wird die Herstellung von DNA-Konstrukten angesehen und ihre Verwendung für mannigfache Anwendungen von DNA-Nanotechnologie, über gezielte ribosomale Proteinsynthese mit natürlichen und künstlichen Aminosäuren bis hin zur genetischen Veränderung von ganzen Organismen. Ebenso wie die chemische Technologie eine ungeheure Fülle von verschiedensten Prozessen um die Kernbereiche herum gruppiert und integriert, könnte die neue Biotechnologie die große Vielfalt der heute als Sammelsurium empfundenen Teilbereiche der synthetischen Biologie miteinander vereinigen.
[1] Auf Grund der sehr umfangreichen Literatur zu Teil 1 und 2 des Essays wird an dieser Stelle auf eine Zitierung verzichtet. Diese sind in einem ausführlichen Übersichtsartikel des Autors zu zahlreichen im Essay besprochenen Aspekten zu finden („Modeling in biological chemistry. From biochemical kinetics to systems biology” PDF; Monatsh Chem 139, 427–446 (2008)).
Auf Anfrage können zitierte Artikel vom Autor erhalten werden (http://www.tbi.univie.ac.at/~pks). Ein leicht verständlicher Artikel des Autors, der ebenfalls mehrere Aspekte des vorliegenden Essays anschneidet, findet sich unter: Ursprung des Lebens aus der Sicht der Chemie (PDF).
Die komplette Sammlung aller Artikel zum Themenschwerpunkt Synthetische Biologie finden Sie hier.
- Printer-friendly version
- Log in to post comments
