Epigenetik - Wie die Umsetzung unserer Erbinformation gesteuert wird
Epigenetik - Wie die Umsetzung unserer Erbinformation gesteuert wirdDo, 15.09.2022 — Christina Beck 
Als im Jahr 2001 die Entschlüsselung des menschlichen Genoms fertiggestellt wurde, war die Euphorie groß: Mehr als drei Milliarden Buchstaben umfasst unsere DNA, und die waren endlich bekannt. Wenn es jetzt noch gelänge, in diesem Buchstabensalat die entsprechenden Gene ausfindig zu machen, würde man die Details unseres genetischen Bauplans und damit auch die Ursachen etlicher Krankheiten kennen, so die Meinung vieler Forschender. »Im Rückblick waren unsere damaligen Annahmen über die Funktionsweise des Genoms dermaßen naiv, dass es fast schon peinlich ist«, zitierte die ZEIT im Jahr 2008 den Biochemiker Craig Venter, der mit seiner Firma Celera an der Genomentschlüsselung beteiligt war. Die Epigenetik liefert nun ein neues Verständnis, wie Ablesung und Umsetzung der Gene durch Markierungen an den Genen - wie sie durch Umwelteinflüsse und Lebensstil zustande kommen und auch weiter vererbt werden können - gesteuert werden. Die Zellbiologin Christina Beck, Leiterin der Kommunikation der Max-Planck-Gesellschaft, berichtet darüber.*
Jahrzehntelang galt das Genom als unveränderlicher Bauplan, der bereits bei der Geburt festgelegt ist. Nicht umsonst wurde das Genom als „Buch des Lebens“ bezeichnet, geschrieben mit einem Alphabet aus vier Buchstaben – den Basen Adenin, Guanin, Cytosin und Thymin. Gene waren Schicksal: Sie sollten Aussehen, Persönlichkeit und Krankheitsrisiken bestimmen. Heute wissen wir, dass die in der DNA gespeicherten Informationen keine 1:1 Blaupause für den Organismus sind. Vielmehr stehen unsere Gene in Wechselwirkung mit verschiedenen Faktoren. Dazu gehören auch sogenannte epigenetische Markierungen. Sie dienen dazu, bestimmte Abschnitte der DNA – quasi wie mit einem „bookmark“ – als „lesenswert“ zu markieren, durch Verweise auf weiter entfernt liegende Abschnitte in neue Zusammenhänge zu bringen oder dem Zugriff der Übersetzungsmaschinerie durch eine Art „Passwort-Schutz“ zu entziehen.
Ein Code jenseits der DNA
Das Ganze darf man wohl als Informationsmanagement bezeichnen. Angesichts der ungeheuren Komplexität des Genoms, insbesondere von Säugetieren, eine absolute Notwendigkeit – aber auch Grundlage für Regulation: Schließlich müssen viele verschiedene Zelltypen koordiniert und aufrechterhalten werden. Und anders als die Buchstaben der DNA unterscheiden sich die epigenetischen Markierungen zwischen unterschiedlichen Arten von Zellen. Zusammengenommen ergeben sie das Epigenom – eine Art Code, der kontextabhängig ausgelesen wird und die Umsetzung der Erbinformation steuert: „Die Epigenetik ist eine zusätzliche Informationsebene, die festlegt, welche Gene potenziell aktivierbar sind“, sagt der Molekularbiologe Alexander Meissner, Direktor am Max-Planck-Institut für molekulare Genetik in Berlin. Gemeinsam mit seinem Team arbeitet er daran, die biochemischen Grundlagen dieser epigenetischen Steuerung zu entschlüsseln. Die Forschenden wollen verstehen, wie Gene an- oder ausgeschaltet werden, was ihre Aktivität verstärkt oder herunterdimmt.
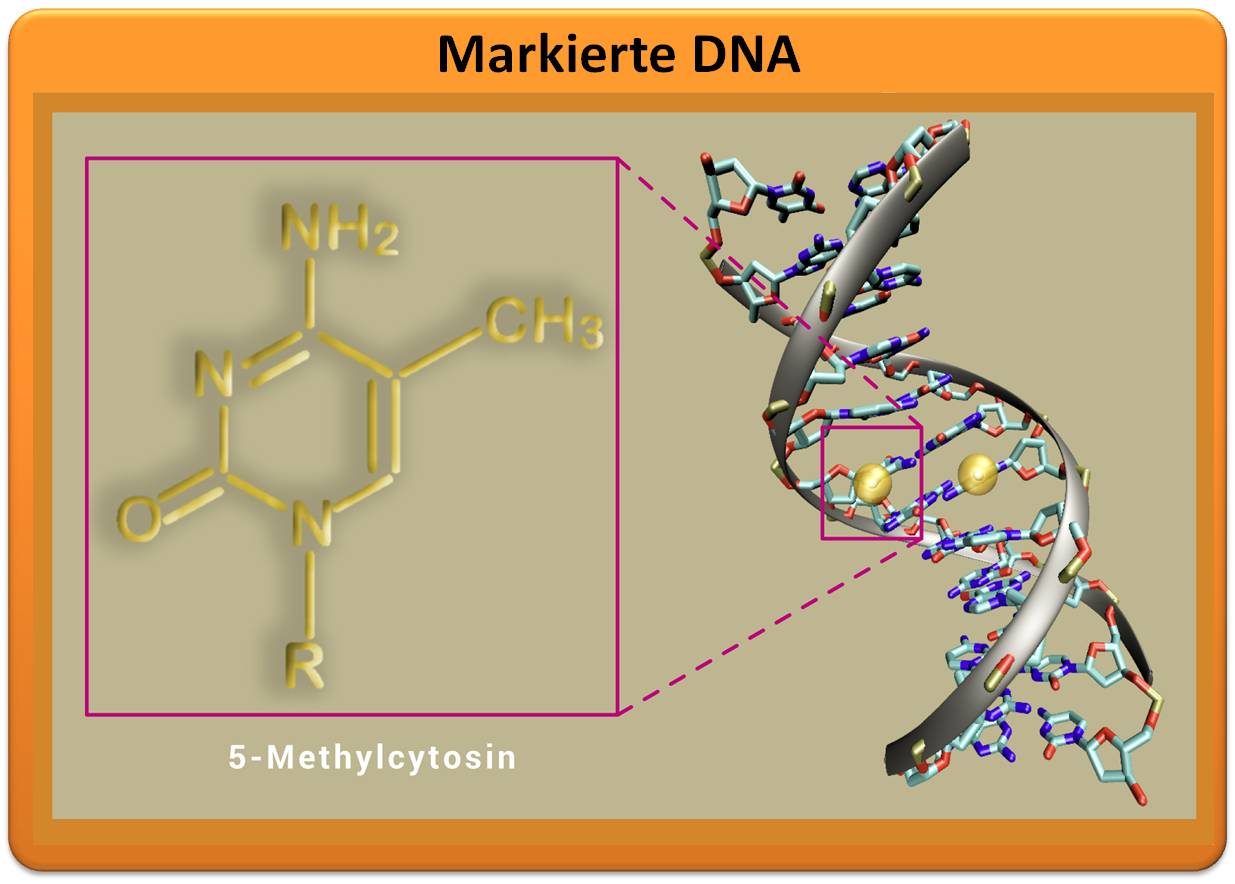
| Abbildung 1: Ausschnitt aus einem DNA-Molekül, bei dem an beiden Strängen ein Cytosin methyliert ist (gelbe Kugeln). Die DNA-Methylierung spielt eine wichtige Rolle bei der epigenetischen Genregulation und dient dazu, DNA-Abschnitte „stumm“ zu schalten. Neben einzelnen Basen können auch Histone methyliert sein.© links: HN; rechts: Christoph Bock/beide CC BY-NC-SA 4.0 |
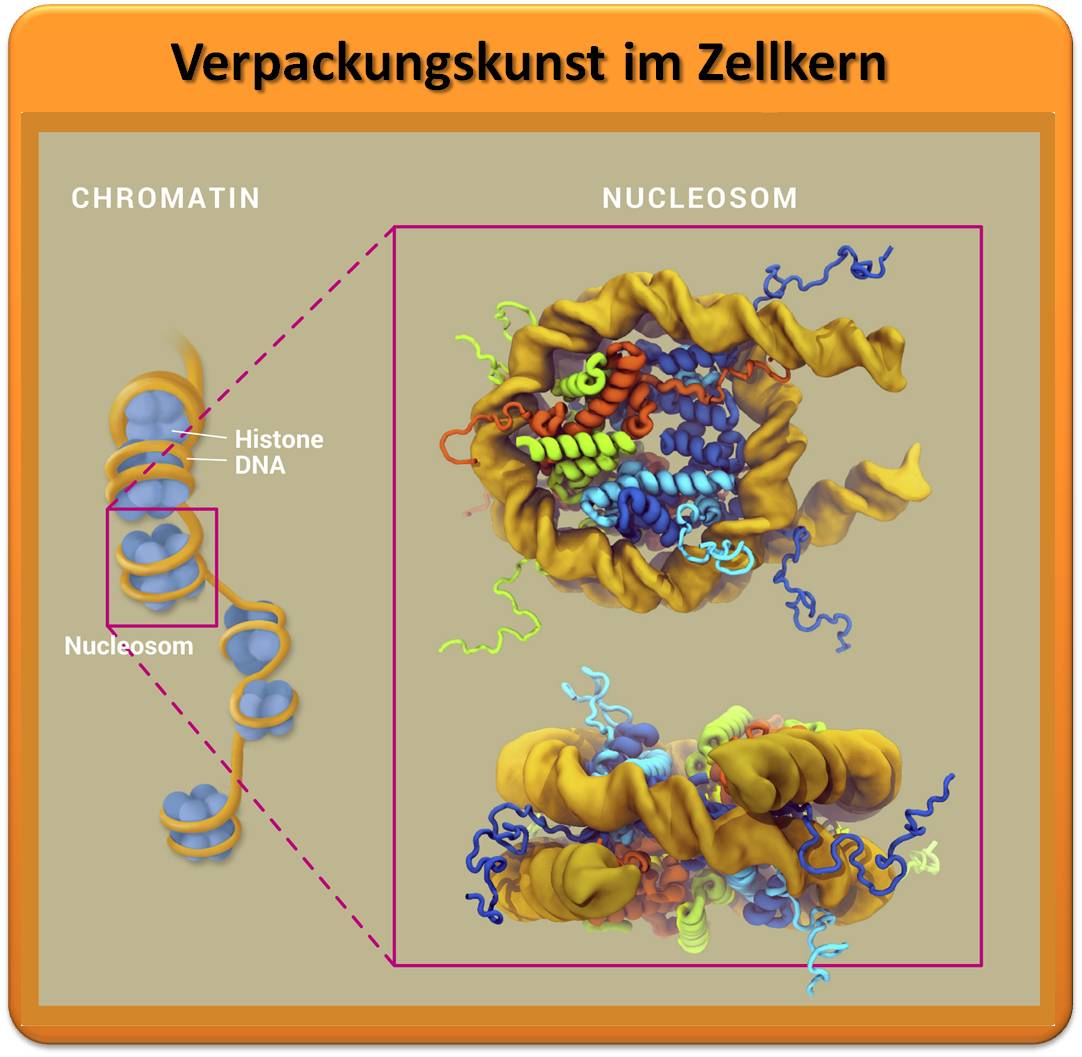
Einer der Regelungsmechanismen setzt direkt an der DNA an: Chemische Markierungen – die Methylgruppen – werden von speziellen Enzymen an die Cytosinbasen der Erbsubstanz angehängt (Abbildung 1). Die so modifizierten DNA-Abschnitte werden daraufhin nicht mehr ausgelesen und in Proteine übersetzt – das Gen ist „stummgeschaltet“. Eine andere Möglichkeit besteht darin, das „Verpackungsmaterial“ der DNA mit chemischen Markierungen zu versehen. Die rund 3,3 Milliarden Nucleotidbasen des menschlichen Genoms ergeben aneinandergereiht einen Faden, der zwei Meter lang ist! Nur durch geschickte Verpackung ist es möglich, den DNA-Faden im Zellkern unterzubringen. Als „Spulen“ dienen dabei die Histone – spezielle Proteine, um die sich die DNA wickelt (Abbildung 2). Aus den so entstehenden Histon-DNA-Partikeln (Nucleosomen) ragen die Proteinschwänze der Histone heraus. An diesen Histonschwänzen sitzen bestimmte epigenetische Markierungen, die zahlreiche Eigenschaften des Chromatins kontrollieren. Dazu zählt etwa, ob das Chromatin locker oder dicht gepackt vorliegt. Die unterschiedlich dichte Verpackung reguliert, welche Gene exprimiert werden und damit, welche Proteine eine Zelle produziert.
Zugang für Ablese-Enzyme
Damit Enzyme die Erbinformationen lesen und abschreiben können, muss die betreffende DNA-Region für sie zugänglich sein. Zugang finden sie jedoch nur, wenn das Chromatin – etwa durch Acetylierung der Histonschwänze – in lockerer Form als sogenanntes Euchromatin vorliegt. Die zusätzlich angehängten Acetylgruppen heben die positiven Ladungen der Histonschwänze auf. So können die negativ geladenen DNA-Moleküle nicht mehr hin reichend neutralisiert werden, die Chromatinstruktur wird instabil und zugänglicher. Auch eine Phosphorylierung der Histonschwänze verändert durch zusätzliche negative Ladungen den Packungszustand des Chromatins und erleichtert das Ablesen bestimmter DNA- Regionen. Durch Reduktion der Acetylgruppen oder auch durch eine Methylierung der Histone nimmt dagegen die Packungsdichte des Chromatins zu. Damit sinkt die Wahrscheinlichkeit, dass eine DNA-Sequenz abgelesen wird und so ihre Funktion ausüben kann. Forschende bezeichnen solche Bereiche als Heterochromatin.
Ein Schritt bei der Öffnung des Chromatins ist die Bewegung der DNA, während sie in Nucleosomen eingewickelt ist. Wie alle molekularen Strukturen in unseren Zellen sind auch die Nucleosomen recht dynamisch. Sie bewegen sich, drehen sich um die eigene Achse, wickeln sich aus und dann wieder ein. Forschende am Max-Planck-Institut für molekulare Biomedizin in Münster haben mithilfe von Computersimulationen gezeigt, dass zwei Histonschwänze dafür verantwortlich sind, das Nucleosom geschlossen zu halten. Nur wenn diese sich von bestimmten Regionen der DNA wegbewegen, kann sich das Nucleosom öffnen, sodass die DNA für Enzyme zugänglich wird und abgelesen werden kann. Die typischen Bewegungen, mit der sich Nucleosomen öffnen und wieder schließen, bezeichnen Forschende als Nucleosomenatmung.
| Abbildung 2: Forschende haben Nucleosomen in Bewegung untersucht. Die oben dargestellten statischen Ansichten sind um 90 Grad gedreht und zeigen die DNA (gelb) zusammen mit den unterschiedlichen Histonen H3 (dunkelblau), H4 (hellblau), H2A (rot) und H2B (hellgrün). Die Histonschwänze ragen aus dem Nucleosom heraus. Sie sind flexibel und spielen eine wichtige Rolle bei der Genexpression. © V. Cojocaru; MPI für molekulare Biomedizin Münster, Utrecht Universität, Babeș-Bolyai-Universität Cluj-Napoca / CC BY-NC-SA 4.0 |
Wie die Vielfalt der Zellen entsteht
Menschen besitzen mehr als 250 verschiedene Zelltypen. Dabei enthalten Hautzellen, Knochenzellen oder Nervenzellen alle dieselbe DNA-Sequenz, obwohl sie ganz unterschiedliche Funktionen erfüllen. Für die Spezialisierung sorgen epigenetische Mechanismen, die den genetischen „Basiscode“ erweitern und die Vielfalt an Expressionsmustern erhöhen: Sie schalten nicht benötigte Gene in bestimmten Zellen stumm, während sie in anderen Zellen aktiv sein können. Alexander Meissner und sein Team vom Max-Planck-Institut für molekulare Genetik in Berlin haben untersucht, wie epigenetische Mechanismen zur Bildung unterschiedlicher Gewebe und Organe im wachsenden Embryo beitragen. Dafür haben sie befruchtete Mäuse-Eizellen mit der Genschere Cas9 (siehe 1) so modifiziert, dass die Enzyme für das Anbringen epigenetischer Markierungen nicht gebildet wurden. Nach einer Entwicklungszeit von sechs bis neun Tagen untersuchten die Forschenden die sich entwickelnden Embryonen. Dabei zeigte sich, dass der Anteil der verschiedenen Zelltypen zum Teil stark verändert war. Von bestimmten Zellen wurden viel zu viele produziert, während andere komplett fehlten. Die Embryonen waren dadurch nicht überlebensfähig. „Wir verstehen jetzt besser, wie epigenetische Regulatoren dafür sorgen, dass wir so viele verschiedene Arten von Zellen im Körper haben“, fasst Meissner die Ergebnisse zusammen. Je nachdem, welche Funktion eine Zelle ausüben soll, versehen Enzyme die DNA mit unterschiedlichen epigenetischen Modifikationen. Sie sorgen dafür, dass sich Stammzellen etwa in Nerven-, Haut-, Herz- oder Muskelzellen weiterentwickeln, sodass sich im wachsenden Embryo unterschiedliche Gewebe und Organe ausbilden.
Folgenreiche Fehlprägung
Epigenetische Mechanismen stecken auch hinter einem Phänomen, das als genomische Prägung bekannt ist und bei verschiedenen Krankheiten eine Rolle spielt: Es tritt bei etwa einem Prozent unserer Gene auf und bewirkt, dass jeweils eine der beiden von den Eltern vererbten Genkopien stummgeschaltet wird. Schädliche Mutationen, die von der Mutter oder dem Vater erworben wurden, können somit nicht durch eine zweite Genkopie ausgeglichen werden. Dies kann zu Krankheiten wie dem Angelman-Syndrom oder dem Prader-Willi-Syndrom, aber auch zu Krebs und Herz-Kreislauf-Erkrankungen führen. Ließe sich das stummgeschaltete gesunde Gen wieder aktivieren, könnte man damit die Störungen des aktiven fehlerhaften Gens möglicherweise ausgleichen. Die Max-Planck-Forschenden wollen daher verstehen, welche Mechanismen für die Inaktivierung der Gene verantwortlich sind. „Erst in den vergangenen Jahren hat sich herauskristallisiert, dass die Prägung auf molekularer Ebene über verschiedene Wege erreicht wird“, sagt Alexander Meissner. Demnach kann auch bei der genomischen Prägung entweder die „Verpackung“ des Erbgutes oder die DNA selbst chemisch modifiziert sein. Um zwischen den verschiedenen Möglichkeiten zu unterscheiden, nutzten die Forschenden wiederum die Genschere Cas9. In befruchteten Mäuse-Eizellen verhinderten sie so die Bildung eines bestimmten Enzyms, das für das Anbringen epigenetischer Markierungen zuständig ist. Anschließend beobachteten sie, ob das fehlgeprägte, stumm geschaltete Gen im weiteren Entwicklungsverlauf wieder aktiv wurde. Auf diese Weise gelang es ihnen nicht nur, die Stummschaltung aufzuheben. Sie konnten auch unterscheiden, welche epigenetischen Mechanismen dahintersteckten. „In den meisten Fällen war das die DNA-Methylierung“, sagt Alexander Meissner.
Schutz vor Eindringlingen
Neben der Genregulation haben epigenetische Mechanismen auch noch eine andere lebenswichtige Funktion: Indem die Zelle bestimmte DNA-Abschnitte stilllegt, kann sie sich vor „parasitären“ Anteilen des Genoms, den Transposons, schützen. „In den meisten Genomen – auch denen von Menschen und Mäusen – verbergen sich tausende virusähnliche Sequenzen, die sich über die Jahrmillionen im Erbgut ihrer Wirte verewigt haben“, so Meissner. Diese auch als „springende Gene“ bekannten Transposons sind in der Lage, sich spontan zu vervielfältigen und an einer beliebigen Stelle der DNA einzubauen. Transposons haben sich im Genom ausgebreitet und machen etwa 40 Prozent des Erbmaterials in Mäusen und Menschen aus. „Methylierung hält diese potenziell schädlichen Erbgutabschnitte in Schach“, sagt Chuck Haggerty, Wissenschaftler in Meissners Team: „Wenn ein Transposon oder Virus mitten in ein Gen springt, könnte das dessen Funktionen beeinträchtigen.“ Die Zelle muss also unbedingt verhindern, dass sich solche Sequenzen unkontrolliert im Genom ausbreiten. Spezielle Enzyme fahnden daher gezielt danach und heften Methylgruppen als chemische Warnhinweise an. Die so markierten DNA-Abschnitte werden daraufhin von der Zelle ignoriert. Ein solches Enzym aus der Gruppe der Methyltransferasen haben die Berliner Forschenden charakterisiert: Es ist dafür zuständig, den epigenetischen Ist-Zustand aufrecht zu erhalten und kann virusartige Erbgutabschnitte auch gezielt stumm schalten.
Verschiedene Studien haben untersucht was passiert, wenn die für die Methylierung zuständigen Enzyme fehlen. In diesem Fall werden viele invasive Elemente wieder aktiviert, und die Mutationsrate der Zellen schnellt nach oben. Experimente wie diese werfen die Frage auf, ob epigenetische Änderungen möglicherweise das genetische Chaos befördern, das mit Krebserkrankungen einhergeht. Tumorzellen tragen nämlich insgesamt oft zu wenige Methylgruppen am Genom, an bestimmten Stellen jedoch wiederum zu viele. Gene für wichtige Reparaturenzyme oder Schutzmechanismen werden dadurch epigenetisch ausgeschaltet. Möglicherweise ergeben sich daraus aber auch Chancen für die Krebstherapie. Denn während Zellen ihre DNA mit hohem Aufwand vor Mutationen schützen, werden epigenetische Markierungen ständig neu gesetzt oder entfernt. Im Prinzip ließen sich daher Medikamente entwickeln, die ganze Gruppen von Genen über epigenetische Effekte wieder an- oder abschalten.
Markierung nach Maß
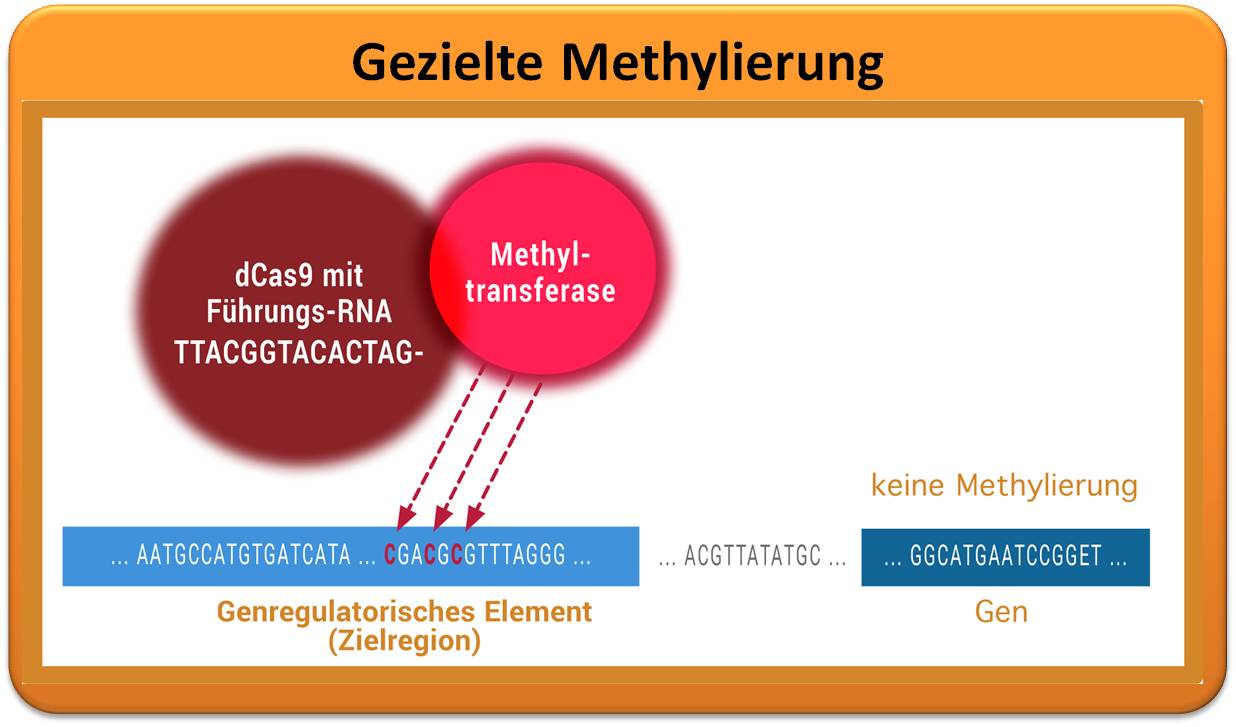
Alexander Meissner und sein Team wollen daher bestimmte Stellen im Genom gezielt methylieren, um herauszufinden, welchen Effekt die künstlich angebrachten Markierungen haben. Auch dabei kommt ihnen eine umgebaute Version von Cas9 zu Hilfe: Das zentrale Element der Genschere ist Cas9 – ein Enzym, das sich mithilfe von synthetisch hergestellten Führungs-RNAs (Single Guide RNA) gezielt an ausgewählte Regionen im Genom lenken lässt und dort die DNA schneiden kann. Dank der rasanten Entwicklung von Cas9 und ähnlichen Enzymen verfügen Molekularbiologinnen und -biologen heute auch über Cas9-Varianten ohne Schneidefunktion. Genauso wie die schneidende Variante sind auch sie in der Lage, bestimmte Regionen im Genom anzusteuern. Da sie sich darüber hinaus flexibel mit Methyltransferasen koppeln lassen, hoffen die Forschenden, damit künftig gezielt einzelne Basen im Genom methylieren zu können (Abbildung 3). Dafür müssen die Forschenden aber zuerst noch an den Details tüfteln: „Bisher ist die Methylierung leider noch zu unspezifisch“, sagt Alexander Meissner. Denn während der Enzymkomplex nach seinem Ziel sucht, werden hier und da bereits Methylgruppen angehängt. Der Wissenschaftler ist jedoch zuversichtlich, dass sich die Methylierung noch zielgenauer machen lässt. Die vielen neuen Möglichkeiten, die sich daraus ergeben würden, sind der größte Ansporn für ihn und sein Team: „Es hätte enormes Potenzial, wenn wir das Epigenom auf diese Weise gezielt verändern könnten.“
| Abbildung 3. Gezielte Methylierung im Genom. Um Cytosinbasen im Genom gezielt zu methylieren, wird eine DNA-Methyltransferase mit einer speziellen Version von Cas9 ohne Schneidefunktion (dCas9) gekoppelt. Da die Führungs-RNA komplementär zur Zielregion ist, lässt sich die Methyltransferase so gezielt in bestimmte Regionen im Genom lenken. Dort versieht sie die Cytosinbasen mit Methylgruppen. Im Idealfall werden dabei nur bestimmte Cytosine methyliert, während weiter entfernt liegende unverändert bleiben. Mit der Methode wird erforscht, welche Methylierungen wann und wo für die Regulation bestimmter Gene verantwortlich sind. © A. Meissner, MPI für molekulare Genetik / CC BY-NC-SA 4.0 |
Gedächtnis für Umwelteinflüsse
Epigenetische Markierungen können angefügt, aber auch wieder entfernt werden. Dadurch ist das Epigenom im Gegensatz zur DNA-Sequenz relativ flexibel und kann auf Umwelteinflüsse reagieren. Andererseits lassen sich epigenetische Signaturen aber auch konservieren: Während der DNA-Replikation – d.h. bei der Verdopplung der Chromosomen in der teilungsbereiten Zelle – können Methylierungsmuster originalgetreu kopiert werden. Das ermöglicht es, die enthaltenen Informationen von einer Zellgeneration an die nächste weiterzugeben und damit dauerhaft zu speichern. Organismen bauen auf diese Weise ein zelluläres Gedächtnis für Umwelteinflüsse auf. Ob Stress, Ernährung, Sport oder Drogenkonsum – letztlich hinterlässt unsere gesamte Lebensweise Spuren im Epigenom. Zwillingsstudien zeigen dies besonders eindrucksvoll: Eineiige Zwillinge haben exakt die gleichen Gene, trotzdem unterscheiden sie sich in den Mustern ihrer Genaktivität und damit auch in ihren Eigenschaften. Die Unterschiede sind epigenetisch bedingt und nehmen mit dem Lebensalter zu: Während bei dreijährigen Zwillingen die Gene noch nahezu gleich „ticken“, sind die Unterschiede bei 50-jährigen fast viermal so häufig. Besonders ungleich sind jene Pärchen, die schon früh getrennte Wege gehen.
Wie der Großvater, so die Enkelin?
Ein besonders spannendes Gebiet der Molekularbiologie ist derzeit die transgenerationelle Epigenetik, die sich mit der Vererbung epigenetischer Informationen befasst: Immer mehr Studien an so unterschiedlichen Organismen wie Fruchtfliegen, Mäusen und Menschen deuten darauf hin, dass epigenetische Muster nicht nur zwischen verschiedenen Zellgenerationen innerhalb des Körpers weitergegeben werden, sondern zumindest in gewissen Situationen sogar über Generationen hinweg. Demnach können etwa die Ernährungslage während der Schwangerschaft, Traumata, Umweltgifte oder Nikotinkonsum nicht nur das Leben der Kinder bis ins hohe Alter beeinflussen, sondern sogar noch bei den Enkeln und darüber hinaus fortwirken. Inwieweit wir nicht nur unsere Gene, sondern auch epigenetische Veränderungen vererben, muss noch genauer erforscht werden. Es scheint, als könne das Leben unserer Großeltern – das Essen, das sie gegessen haben oder die Erfahrungen, die sie gemacht haben – uns womöglich noch Jahrzehnte später beeinflussen. Und das, obwohl wir selbst diese Dinge nie erfahren haben!
---------------------------------------
[1] Christina Beck,23.04.2020: Genom Editierung mit CRISPR-Cas9 - was ist jetzt möglich?
* Der Artikel unter dem Titel "Epigenetik - das Gedächtnis unserer Gene" stammt aus dem BIOMAX 23-Heft der Max-Planck-Gesellschaft https://www.max-wissen.de/max-hefte/biomax-23-epigenetik/, das im Sommer 2022 unter Mitwirkung von Dr. Elke Maier (Redaktion Max-Planck-Forschung) aktualisiert wurde. Mit Ausnahme des Titels wurde der unter einer cc-by-n-sa Lizenz stehende Artikel unverändert in den Blog übernommen.
Weiterführende Links
Max Planck-Gesellschaft: Epigenetik - Änderungen jenseits des genetischen Codes. Video 5:04 min. https://www.youtube.com/watch?v=xshPL5hU0Kg&t=291s
Max Planck-Gesellschaft Epigenetik - Verpackungskunst in der Zelle. Video 8:09 min. https://www.youtube.com/watch?v=0VQ62pD5eqQ
Peter Spork, Newsletter Epigenetik: https://www.peter-spork.de/86-0-Newsletter-Epigenetik.html
Epigenetik im ScienceBlog
- Gottfried Schatz, 22.08.2014: Jenseits der Gene — Wie uns der Informationsreichtum der Erbsubstanz Freiheit schenkt
- Norbert Bischofberger, 24.05.2018: Auf dem Weg zu einer Medizin der Zukunft
- Peter Schuster, 12.03.2020: Molekularbiologie im 21. Jahrhundert