Fr. 17.03.2023 — Redaktion
![]()
Die weltweite Ausbreitung der Antibiotikaresistenz in Umwelt und Bevölkerung könnte nicht - wie bisher angenommen - nur auf den übermäßigen Einsatz von Antibiotika in Landwirtschaft und Medizin zurückzuführen sein. Eine neue, enorm umfangreiche Analyse über die Verbreitung von Resistenzgenen gegen die seit den 1940er Jahren angewandte Antibiotika-Klasse der Aminoglykoside bringt wichtige neue Erkenntnisse über eine wesentliche Rolle von Ökologie (Biome) und menschlich generiertem Austausch (Importe durch Einwanderung und Wareneinfuhr). Die Rolle der Ökologie erscheint nicht verwunderlich, beruhen doch die meisten Antibiotika auf von Bakterien und Pilzen produzierten Naturstoffen, gegen die andere Mikroorgannismen - auch ganz ohne menschliche Einflussnahme - Resistenzen entwickelt haben und weiter entwickeln.*
Nach wie vor stellt Antibiotikaresistenz - die Fähigkeit von Bakterien, selbst die härtesten klinischen Behandlungen zu überleben - in der ganzen Welt ein massives Problem für die Gesundheit der Bevölkerung dar. Häufig wird der übermäßige Einsatz von Antibiotika in Medizin und Landwirtschaft als treibende Kraft für die Entstehung und Ausbreitung von Bakterien angesehen, die gegen Antibiotika resistent sind. In Gebieten, die stark von den Tätigkeiten des Menschen geprägt sind, kann exzessive Antibiotika Anwendung sicherlich die Selektion von Bakterien mit resistenten Genen gegen Antibiotika erklären. Dass aber Resistenzgene gegen klinisch relevante Antibiotika in Gegenden weitab von Krankenhäusern und landwirtschaftlichen Betrieben weit verbreitet sind, ist so nicht erklärbar. Tatsächlich wurden solche Gene sogar in so entlegenen Umwelten wie dem arktischen Permafrost und der Antarktis gefunden.
Nun berichten Léa Pradier und Stéphanie Bedhomme von der Universität Montpellier im Fachjournal eLife über die Ergebnisse einer Studie, die Licht in diese Fragestellung bringt [1]. Die Forscherinnen haben eine der bisher umfangreichsten Studien über Resistenzgene gegen Antibiotika durchgeführt, wobei der Schwerpunkt auf der Resistenz gegen Aminoglykoside lag.
Aminoglykoside
sind eine altbekannte, häufig angewandte Familie von Antibiotika, welche die Proteinsynthese von Bakterien blockieren. Der erste Vertreter dieser Klasse - das aus dem Bodenbakterium Streptomyces griseus isolierte Streptomycin - wurde bereits 1944 erfolgreich gegen Tuberkulose eingesetzt und erwies sich als erstes wirksames Mittel gegen gram-negative Bakterien. Später landeten noch weitere Isolate wie Kanamycin, Neomycin, Apramycin und chemisch modifizierte weiere Aminoglykoside in der Klinik. Bald tauchten aber erste Befunde zu resistenten Bakterienstämmen auf; zusammen mit einer Injektion erfordernden Applikationsart und ernsten Nebenwirkungen auf Niere und Gehör kam es zu einer stark reduzierten Anwendung von Aminoglykosiden in der Humanmedizin, die nun im wesentlichen als Mittel der zweiten Wahl oder als letzte Möglichkeit in der Behandlung von Infektionen mit gram-negativen Bakterien eingesetzt werden. Nach wie vor haben diese Antibiotika hohe Bedeutung in der Veterinärmedizin.
Die häufigste Ursache für die Entstehung von Aminoglykosidresistenz in klinischen Isolaten sind inaktivierende Enzyme, die die Übertragung chemischer Gruppen an Aminoglykosidmolekülen katalysieren: ein so modifiziertes Medikament kann dann nur mehr schlecht an seine Zielstruktur binden - es wurde unwirksam. Solche Aminoglykosid-modifizierende Enzyme (AMEs) sind biochemisch gut charakterisiert, ihre Bezeichnung basiert auf der Gruppe, die sie übertragen Die klassische Nomenklatur der AMEs basiert auf der Gruppe, die sie übertragen (d. h. Acetyltransferasen, AACs; Nukleotidyltransferasen, ANTs; und Phosphotransferasen, APHs).
Die Studie
Mit dem Ziel die zeitlichen, räumlichen und ökologischen Verteilungsmuster der Aminoglykosid-Resistenz zu beschreiben, haben Pradier und Bedhomme mehr als 160.000 öffentlich zugängliche Genome von Bakterien auf 27 Cluster von homologen Genen (CHGs - Gene, deren Sequenz einen gemeinsamen Vorläufer wahrscheinlich macht) untersucht, die für Aminoglykosid-modifizierende Enzyme kodieren (AME-Gene). Die Sequenzierungsdaten stammten von Bakterien aus allen Kontinenten (außer der Antarktis) und terrestrischen Ökoregionen (Biomen) und betreffen mit insgesamt 54 Bakterienstämmen (Phyla) die weitaus überwiegende Mehrheit aller Bakterien-Phyla. Die zur Sequenzierung herangezogenen Bakterien waren im Zeitraum zwischen 1885 und 2019 gesammelt worden, die meisten davon erst nach 1990.
Zusätzlich zu Ort und Datum der Probenahme wurden in der Studie auch die Anzahl der in den einzelnen Ländern konsumierten Antibiotika, die Handelsrouten und die menschliche Migration berücksichtigt.
Die Ergebnisse
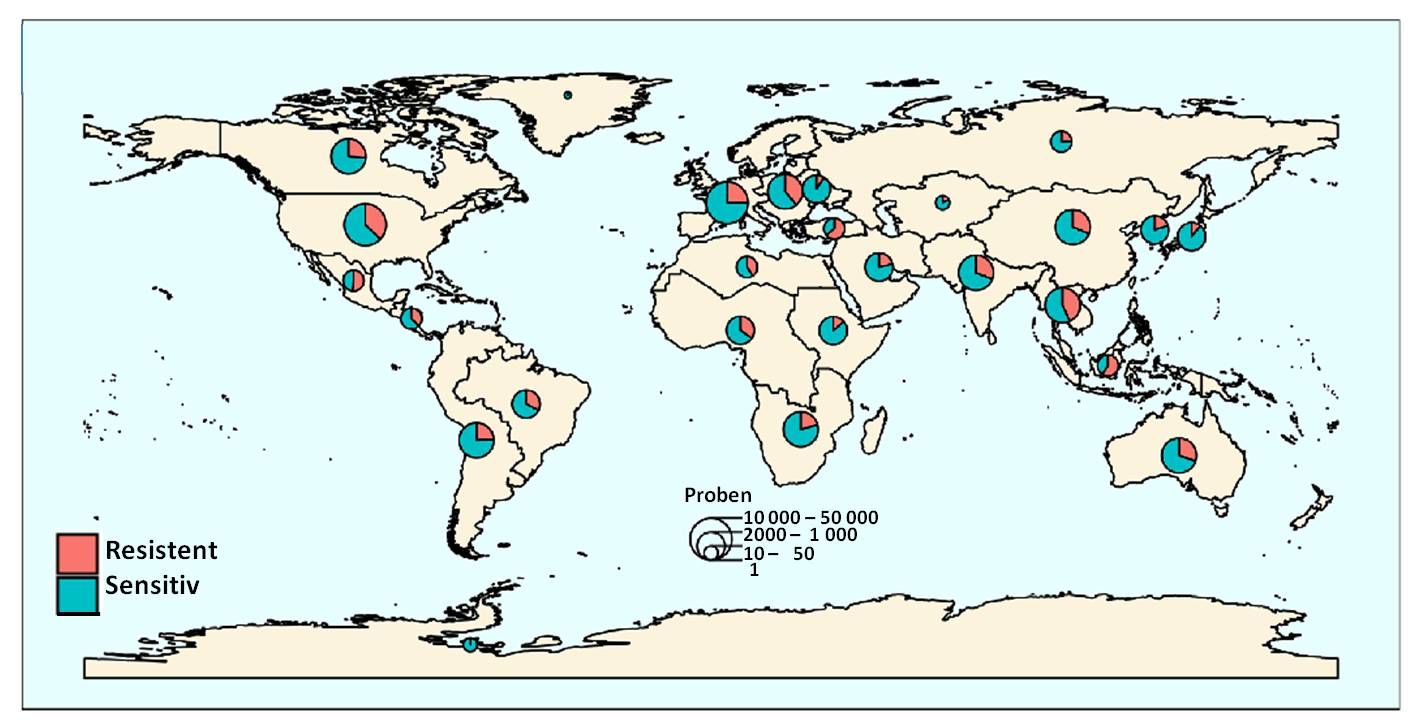
| Abbildung 1 Globale Verteilung von Aminoglykosid-resistenten Bakterien. Die Größe der Kreise ist ein Maß für die Zahl der untersuchten Genome. (Bild von Redn. eingefügt und leicht modifiziert nach L. Pradier and S. Bedhomme (2023), [1]; Lizenz cc-by.) |
Resistenzgene gegen Aminoglykoside
zeigen eine weltweite geographische Verbreitung, sie kommen in allen Ökoregionen (Biomen) vor und wurden in 23 der 54 sequenzierten Bakterienstämme entdeckt. In den insgesamt rund 160 000 bakteriellen Genomen wurden 46 053 AME-Gene - d.i. in rund 25 % aller untersuchten Bakterien-Genomen - gefunden. In den meisten Regionen liegt die Häufigkeit der AME-Gen-tragenden Bakterien zwischen 20 % und 40 %; sie reicht von 10 % in Japan, Osteuropa und Ostafrika bis zu 50% in Indonesien, Mexiko und der Türkei. Abbildung 1.
Die Verteilung der für AME-kodierenden Gencluster
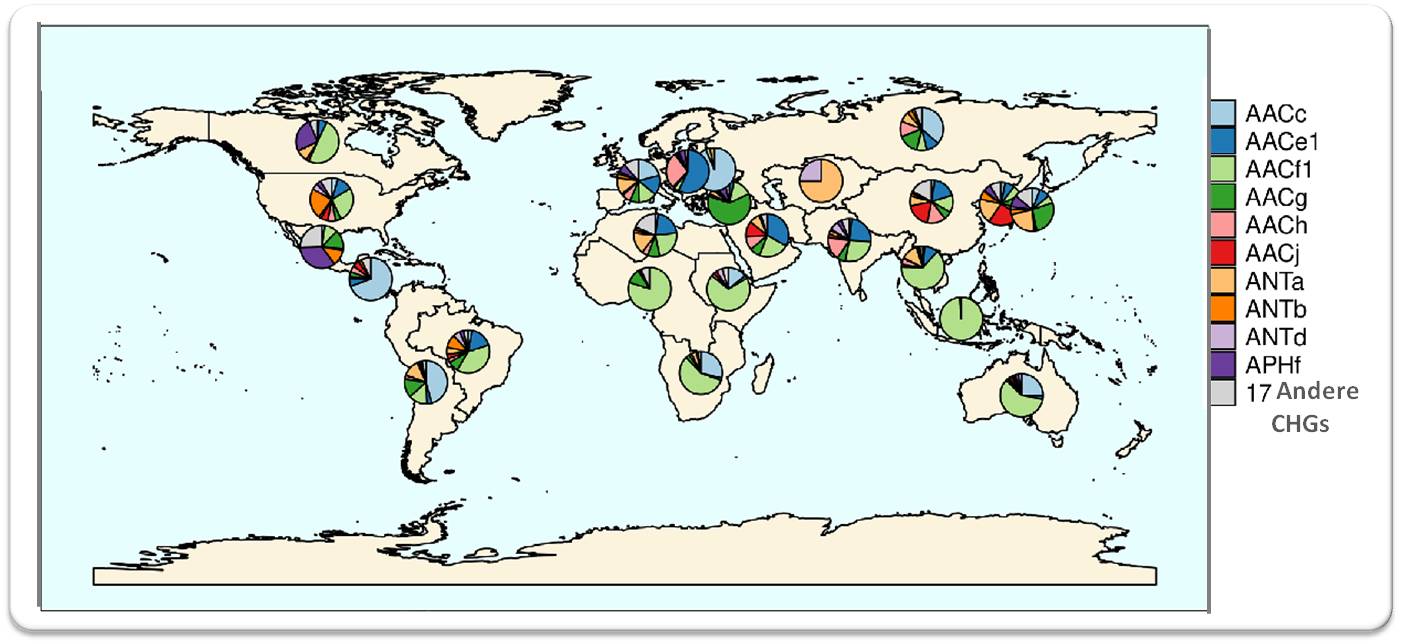
weist geographisch eine hohe Heterogenität auf (Abbildung 2). Bestimmte (für Acetyltransferasen AACf1 kodierende) Resistenzgene dominierten in der südlichen Hemisphäre von Afrika, Asien und Australien , während in den US und Westeuropa eine ausgewogene Mischung von Resistenzgenen detektiert wurde.
| Abbildung 2 Wie sich die 27 für die Aminoglykosid-modifizierenden Enzyme (AME) kodierenden Gencluster global verteilen. Die Bezeichnung der Gencluster basiert auf der Gruppe, die die entsprechenden Enzyme übertragen: AACs = Acetyltransferasen; ANTs = Nukleotidyltransferasen, und APHs = Phosphotransferasen). (Bild von Redn. eingefügt und leicht modifiziert nach L. Pradier and S. Bedhomme (2023), [1]; Lizenz cc-by.) |
Die Forscherinnen fanden heraus, dass die Häufigkeit von Aminoglykosid-Resistenzgenen zwischen den 1940er und 1980er Jahren zugenommen hat, was wahrscheinlich auf den verstärkten Einsatz von Aminoglykosid-Antibiotika nach der Entdeckung von Streptomycin im Jahr 1943 zurückzuführen ist, dann aber - trotz eines allgemeinen Rückgangs des Verbrauchs - bei einer Prävalenz von etwa 30 % blieb. Entscheidend ist, dass sie auch entdeckten, dass etwa 40 % der Resistenzgene potenziell mobil sind, d. h. leicht zwischen Bakterien ausgetauscht werden können.
Vorkommen in den Biomen
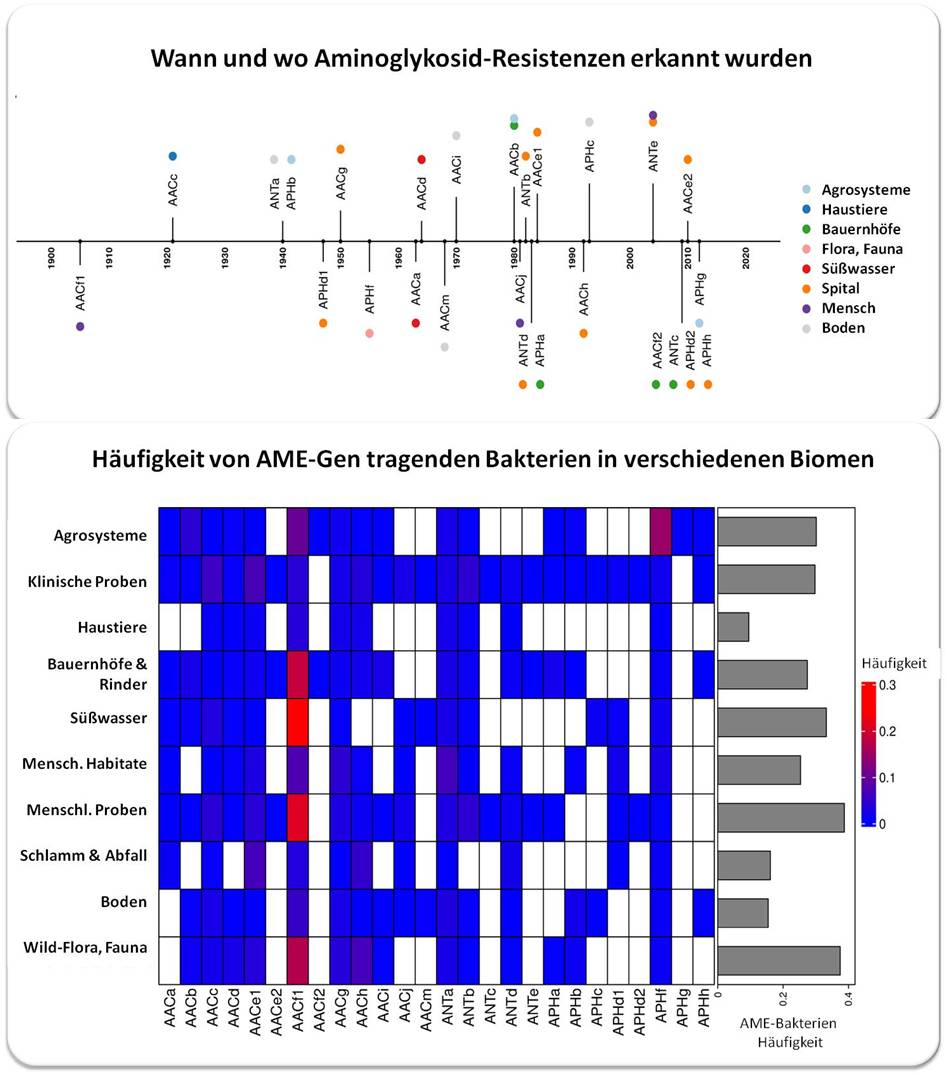
Pradier und Bedhomme stellten außerdem fest, dass antibiotikaresistente Bakterien in den meisten Biomen vorkommen, nicht nur in Krankenhäusern und landwirtschaftlichen Betrieben. (Abbildung 3).
| Abbildung 3.Resistenzgene gegen Aminoglykoside wurden bereits 4 Jahrzehnte vor der erstmaligen klinischen Anwendung entdeckt (oben) und sie sind nun in allen untersuchten Biomen in einer Häufigkeit von knapp über 0 bis 30 % präsent (unten: blau bis rot; leere Kästchen bedeuten 0 Resistenzgene). Die Bezeichnung der Gencluster basiert auf der Gruppe, welche die entsprechenden Enzyme übertragen: AACs = Acetyltransferasen; ANTs = Nukleotidyltransferasen, und APHs = Phosphotransferasen. (Bild von Redn. eingefügt und leicht modifiziert nach L. Pradier and S. Bedhomme (2023), [1]; Lizenz cc-by) |
Darüber hinaus fanden sie, dass die Prävalenz von Aminoglykosid-Resistenzgenen von Biom zu Biom stärker variierte, als dies mit der geografischen Lage von Menschen oder der Menge der verwendeten Antibiotika der Fall war. Für das Biom Mensch bedeutet dies, dass die in einem Land festgestellte Antibiotikaresistenz viel wahrscheinlicher mit der Antibiotikaresistenz in einem entfernteren Land korreliert ist, als mit der in der näheren Umgebung im Biom Boden oder im Biom Tiere.
Außerdem entdeckten sie, dass Biome wie Boden und Abwasser wahrscheinlich eine Schlüsselrolle bei der Verbreitung der Gene für die Antibiotikaresistenz über verschiedene Biome hinweg spielen. Eine Analyse der in Europa zwischen 1997 und 2018 gesammelten Proben zeigt deutlich wovon die Häufigkeit des Vorkommens von 16 AME-Gencluster abhängt: in 80 % der Fälle ist die Ökologie treibende Kraft in der Verbreitung der Resistenzgene, in 13 % menschlich generierter Austausch (Importe durch Einwanderung und Wareneinfuhr) und nur in 7 % der Fälle (übermäßige ) Anwendung.
Fragen
Diese Ergebnisse werfen wichtige Fragen zu den Mechanismen auf, die der Ausbreitung der Antibiotikaresistenz zugrunde liegen.
- Welche Faktoren begünstigen die Ausbreitung der Antibiotikaresistenz in Umgebungen, die nicht durch menschliche Aktivitäten beeinflusst werden?
- Können wir die von den Aminoglykosiden erhaltenen Ergebnisse auf alle anderen Antibiotikaklassen übertragen?
- Ist es möglich, dass die Antibiotikaresistenz eher auf Interaktionen mit lokalen mikrobiellen Gemeinschaften zurückzuführen ist, als auf die Exposition gegenüber kommerziellen Antibiotika?
- Verbreiten sich die Gene für die Antibiotikaresistenz in den pathogenen Bakterien, die für Infektionen bei Mensch und Tier verantwortlich sind, auf die gleiche Weise wie in den nicht-pathogenen Bakterien?
- Was begrenzt angesichts des Ausmaßes des Selektionsdrucks durch die menschliche Verschmutzung die Ausbreitung von Antibiotikaresistenzgenen zwischen Biomen, insbesondere angesichts des großen Anteils von Genen, die auf mobilen Elementen sitzen?
Es kann gut sein, dass der Verbrauch immer noch eine überragende Rolle spielt, wenn es um die Resistenz gegen Antibiotika geht, die zur Behandlung von Infektionen eingesetzt werden, insbesondere beim Menschen und in klinischen Biomen. Dennoch ist klar, dass wir der Rolle der Umwelt bei der Formulierung von Plänen zur Bekämpfung der Antibiotikaresistenz auf globaler Ebene mehr Aufmerksamkeit schenken müssen.
[1] Pradier L, Bedhomme S. 2023. Ecology, more than antibiotics consumption, is the major predictor for the global distribution of aminoglycoside-modifying enzymes. eLife 12:e77015. doi: 10.7554/eLife.77015
*Eine Zusammenfassung des Artikels von Léa Pradier und Stéphanie Bedhomme, 2023, [1] verfasst von Carolina Oliveira de Santana et al., ist am 8.3.2023 unter dem Titel "Antibiotic Resistance: A mobile Target " im eLife Magazin erschienen: : https://elifesciences.org/articles/86697 . Der Text wurde von der Redaktion ins Deutsche übersetzt und mit einigen Textststellen und 4 Abbildungen aus dem Originaltext [1] plus Legenden ergänzt. eLife ist ein open access Journal, alle Inhalte stehen unter einer cc-by Lizenz.
Antibiotika-Resistenz im ScienceBlog
Redaktion, 11.12.2022: Die globale Krise der Antibiotikaresistenz und diesbezügliches Wissen und Verhaltensweisen der EU-Bürger (Spezial Eurobarometer 522)
08.07.2021, Redaktion: Phagen und Vakzinen im Kampf gegen Antibiotika-resistente Bakterien
Karin Moelling, 29.08.2019: Ein Comeback der Phagentherapie?
Karin Moelling, 4.07.2019: Viren gegen multiresistente Bakterien. Teil 1: Was sind Phagen?
Inge Schuster, 23.09.2016: Gehen wir auf eine Post-Antibiotika Ära zu?
Gottfried Schatz, 30.05.2013: Planet der Mikroben — Warum wir Infektionskrankheiten nie endgültig besiegen werden
- Printer-friendly version
- Log in to post comments