Wie Gene aktiv werden
Wie Gene aktiv werdenFr, 26.08.2016 - 10:22 — Patrick Cramer

![]() Um die Erbinformation in lebenden Zellen zu nutzen, müssen Gene aktiviert werden. Die Gen-Aktivierung beginnt mit einem Kopiervorgang, der Transkription, bei dem eine Genkopie in Form von RNA erstellt wird. Der Biochemiker Patrick Cramer (Direktor am Max-Planck Institut für biophysikalische Chemie, Göttingen) erforscht mit seinem Team, wie diese Kopiermaschinen ("RNA-Polymerasen") im Detail aufgebaut sind, wie sie arbeiten und gesteuert werden. Es sind bahnbrechenden Untersuchungen mittels strukturbiologischer Methoden, die nun erstmals eine Beschreibung des Kopiervorgangs und der Kopiermaschinen - der RNA-Polymerasen - in atomarem Detail ermöglichen.*
Um die Erbinformation in lebenden Zellen zu nutzen, müssen Gene aktiviert werden. Die Gen-Aktivierung beginnt mit einem Kopiervorgang, der Transkription, bei dem eine Genkopie in Form von RNA erstellt wird. Der Biochemiker Patrick Cramer (Direktor am Max-Planck Institut für biophysikalische Chemie, Göttingen) erforscht mit seinem Team, wie diese Kopiermaschinen ("RNA-Polymerasen") im Detail aufgebaut sind, wie sie arbeiten und gesteuert werden. Es sind bahnbrechenden Untersuchungen mittels strukturbiologischer Methoden, die nun erstmals eine Beschreibung des Kopiervorgangs und der Kopiermaschinen - der RNA-Polymerasen - in atomarem Detail ermöglichen.*
Gene zum Sprechen bringen
Zu den Kennzeichen des Lebens gehört, dass Organismen sich entwickeln und am Ende die Art erhalten. Dazu bedarf es der Erbinformation, die in lebenden Zellen in Form der DNA vorliegt. Die DNA ist ein fadenförmiges Molekül mit tausenden von funktionalen Abschnitten, wozu die Gene gehören. Gene sind für sich genommen stumm. Doch es kann ihnen Sprache verliehen werden. Dies geschieht während der Gen-Ausprägung, auch Gen-Expression genannt, bei der die genetische Information zur Synthese von Proteinen genutzt wird. Der erste Schritt in diesem Prozess ist die Transkription, ein Kopiervorgang, bei dem RNA-Kopien von Genen erstellt werden. Die biologischen Kopiermaschinen heißen RNA-Polymerasen und sind in der Lage, DNA in RNA zu übersetzen. Zu verstehen, wie Gene aktiviert werden und wie Genaktivität reguliert wird, ist von großem biomedizinischem Interesse.
Die Gen-Kopiermaschinen
In eukaryontischen Zellen gibt es mehrere RNA-Polymerasen, die verschiedene Gene kopieren. Dreidimensionale Strukturen von Polymerasen wurden in den letzten Jahren mit Hilfe der Röntgenkristallographie bestimmt. Die Struktur der RNA-Polymerase I umfasst 14 Untereinheiten und zeigt, wie diese Maschine in einem inaktiven Zustand auf ihren Einsatz zur Kopie ihres Zielgens wartet (Abbildung. 1; [1]). 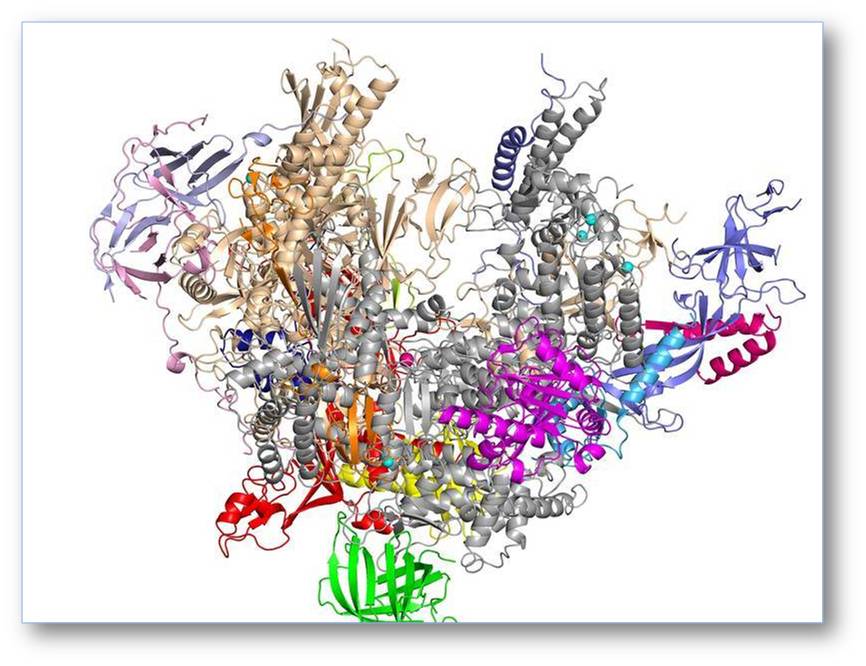
Abbildung 1: Atomare Struktur der RNA-Polymerase I. Die Struktur dieses sehr großen (Molekulargewicht 590 000), aus 14 Untereinheiten bestehenden Enzyms wurde im Labor des Autors mithilfe der Röntgenkristallographie ermittelt [1]. © Max-Planck-Institut für biophysikalische Chemie/Cramer.
Die Struktur der RNA-Polymerase II ist bereits aus mehreren aktiven und inaktiven Zuständen bekannt [2]. Aus der Vielzahl der strukturellen Schnappschüsse konnte so ein Film der Gen-Transkription erstellt werden (Abbildung 2; [3]). Eine spezialisierte, viel kleinere RNA-Polymerase findet sich in den Kraftwerken der Zelle, den Mitochondrien [4].
Alle Polymerasen haben ein aktives Zentrum, das RNA-Moleküle anhand einer DNA-Vorlage synthetisieren kann. Die Kopiermaschinen unterscheiden sich aber auf ihrer Oberfläche. Dies ermöglicht es, verschiedene Polymerasen zu ihren spezifischen Zielgenen zu bringen und individuell zu regulieren.
 Abbildung 2: Atomare Struktur der RNA-Polymerase II. Auch die Struktur dieses großen, komplexen Proteins (Molekulargewicht 550 000) wurde im Labor des Autors mithilfe der Röntgenkristallographie ermittelt [2]. Die Doppelhelix der DNA (blau-türkis), im aktiven Zentrum des Enzyms angedockt, ist hier bereits geöffnet, ein Strang dient als Vorlage (Template) für die Synthese eines neuen RNA-Strangs (roter Strang). © Max-Planck-Institut für biophysikalische Chemie/Cramer
Abbildung 2: Atomare Struktur der RNA-Polymerase II. Auch die Struktur dieses großen, komplexen Proteins (Molekulargewicht 550 000) wurde im Labor des Autors mithilfe der Röntgenkristallographie ermittelt [2]. Die Doppelhelix der DNA (blau-türkis), im aktiven Zentrum des Enzyms angedockt, ist hier bereits geöffnet, ein Strang dient als Vorlage (Template) für die Synthese eines neuen RNA-Strangs (roter Strang). © Max-Planck-Institut für biophysikalische Chemie/Cramer
Wie die Gen-Abschrift beginnt
Wie die Transkription startet, ist am besten für die RNA-Polymerase II verstanden. Dieses Enzym arbeitet dazu mit mehreren spezifischen Faktoren zusammen. Die Faktoren helfen zunächst, den Beginn eines Gens auf der DNA zu finden. Dann wird die DNA, die als Doppelhelix vorliegt, entwunden, was den Matrizenstrang freilegt. Nun kann die RNA-Polymerase den Matrizenstrang binden und den Kopiervorgang einleiten. Wie einige der Schritte dieser sogenannten Initiation ablaufen, konnte kürzlich sichtbar gemacht werden [5, 6]. Dabei weisen sowohl die RNA-Polymerase als auch die zusätzlichen Faktoren eine große Flexibilität auf. Die Dynamik der Transkription und die Beteiligung dutzender von Faktoren, die nur vorrübergehend präsent sind, erschweren allerdings die Strukturanalyse und eine vollständige Aufklärung des Prozesses.
Wie der Kopiervorgang reguliert wird
In menschlichen Zellen sind über Tausend Faktoren bekannt, die die Transkription regulieren. Sie sorgen dafür, dass die Transkription nur an denjenigen Genen startet, die zu einer bestimmten Zeit und an einem bestimmten Ort im Organismus aktiviert werden müssen. Die regulatorischen Faktoren können den Prozess der Initiation nur indirekt steuern, indem Sie auf sogenannte Koaktivatoren Einfluss nehmen. Der prominenteste Koaktivator ist der sogenannte Mediator-Komplex, der fast dreimal so groß wie die RNA-Polymerase II selbst ist und aus 25 bis 35 Untereinheiten besteht. Es ist jetzt gelungen, Einsichten in den Aufbau des Mediators zu erlangen [7] und seine Position auf der RNA-Polymerase II zu bestimmen (Abbildung 3; [8]).
 Abbildung. 3: Regulierung des Transkriptionsvorgangs. Oben: Schematische Darstellung der RNA-Polymerase II, die zusammen mit vielen Faktoren - darunter dem riesige Mediatorkomplex - eine hochkomplexe, dynamische Genkopiermaschine bildet (Bild von der Homepage des Autors eingefügt; Red.). Unten: Derzeitiges Modell der RNA-Polymerase II (silber) mit gebundenem Mediatorkomplex (blau) und weiteren Faktoren. Diese Struktur wurde mithilfe der Kryo-Elektronenmikroskopie bestimmt. © Max-Planck-Institut für biophysikalische Chemie/Cramer
Abbildung. 3: Regulierung des Transkriptionsvorgangs. Oben: Schematische Darstellung der RNA-Polymerase II, die zusammen mit vielen Faktoren - darunter dem riesige Mediatorkomplex - eine hochkomplexe, dynamische Genkopiermaschine bildet (Bild von der Homepage des Autors eingefügt; Red.). Unten: Derzeitiges Modell der RNA-Polymerase II (silber) mit gebundenem Mediatorkomplex (blau) und weiteren Faktoren. Diese Struktur wurde mithilfe der Kryo-Elektronenmikroskopie bestimmt. © Max-Planck-Institut für biophysikalische Chemie/Cramer
Für diese Arbeiten wurde die Kryo-Elektronenmikroskopie angewandt, die es nun aufgrund von technischen Entwicklungen ermöglicht, auch sehr große und flexible Komplexe in molekularem Detail sichtbar zu machen. So wurde beispielsweise erkannt, wie der Mediator den Prozess der Initiation der Transkription erleichtern und so Gene aktivieren kann.
Vom Molekül zum System
Zukünftig muss die Genaktivität möglichst als Ganzes im lebenden System der Zelle untersucht werden, um so Zusammenhänge zwischen der Ausprägung einzelner Gene besser zuordnen zu können. Dazu werden immer mehr Methoden entwickelt, die es erlauben, die Aktivität aller Gene gleichzeitig zu studieren. In jüngeren Studien wurde gezeigt, dass alle aktiven Gene eine besondere, modifizierte Form der RNA-Polymerase II tragen [9]. Auch gibt es in Zellen einen Mechanismus, der dafür sorgt, dass fehlgeleitete Transkription generell gestoppt wird [10]. So werden Polymerasen, die fälschlicherweise Regionen im Erbgut kopieren, die außerhalb der Gene liegen, von der DNA losgelöst. Das unbrauchbare RNA-Produkt wird nachfolgend abgebaut.
Nun gilt es, die molekularen und systemischen Ansätze derart zu kombinieren, dass ein umfassendes Verständnis der Genaktivität erreicht wird. Ein tiefes Verständnis dieser fundamentalen Prozesse könnte es ermöglichen, eine Fehlregulation der Genaktivität, wie sie bei vielen Krankheitsprozessen und insbesondere beim Krebs auftritt, zu korrigieren.
Literaturhinweise
[1] Engel, C.; Sainsbury, S.; Cheung, A. C.; Kostrewa, D.; Cramer, P. RNA polymerase I structure and transcription regulation. Nature 502, 650-655 (2013)
[2] Cheung, A.C.; Cramer, P. Structural basis of RNA polymerase II backtracking, arrest and reactivation. Nature 471, 249-253 (2011)
[3] Cheung, A.C.; Cramer, P. A movie of RNA polymerase II transcription. Cell 149, 1431-1437 (2012)
[4] Ringel, R.; Sologub, M.; Morozov, Y.I.; Litonin, D.; Cramer, P.; Temiakov, D. Structure of human mitochondrial RNA polymerase. Nature 478, 269-273 (2011)
[5] Kostrewa, D.; Zeller, M.E.; Armache, K.J.; Seizl, M.; Leike, K.; Thomm, M.; Cramer, P. RNA polymerase II-TFIIB structure and mechanism of transcription initiation. Nature 462, 323-330 (2009)
[6] Sainsbury, S.; Niesser, J.; Cramer, P. Structure and function of the initially transcribing RNA polymerase II-TFIIB complex. Nature 493, 437-440 (2013)
[7] Laivière, L.; Plaschka, C.; Seizl, M.; Wenzeck, L.; Kurth, F.; Cramer, P. Structure of the Mediator head module. Nature 492, 448-451 (2012)
[8] Plaschka, C.; Larivière, L.; Wenzeck, L.; Seizl, M.; Hemann, M.; Tegunov, D.; Petrotchenko, E.V.; Borchers, C.H.; Baumeister, W.; Herzog, F.; Villa, E.; Cramer, P. Architecture of the RNA polymerase II-Mediator core initiation complex. Nature 518, 376-380 (2015)
[9] Mayer, A.; Heidemann, M.; Lidschreiber, M.; Schreieck, A.; Sun, M.; Hintermair, C.; Kremmer, E.; Eick, D.; Cramer, P. CTD tyrosine phosphorylation impairs termination factor recruitment to RNA polymerase II. Science 336, 1723-1725 (2012)
[10] Schulz, D.; Schwalb, B.; Kiesel, A.; Baejen, C.; Torkler, P.; Gagneur, J.; Soeding, J.; Cramer, P.Transcriptome surveillance by selective termination of noncoding RNA synthesis. Cell 155, 1075-1087 (2013)
* Der gleichnamige im Jahrbuch der Max-Planck Gesellschaft 2016 erschienene Artikel ( https://www.mpg.de/9956356/MPIbpc_JB_20161?c=10583665&force_lang=de ) wurde mit freundlicher Zustimmung des Autors und der MPG-Pressestelle ScienceBlog.at zur Verfügung gestellt. Er erscheint hier geringfügig für den Blog adaptiert (u.a. wurde eine Grafik von der homepage des Autors in Abbildung 3 eingefügt). Die nicht frei zugänglichen Literaturstellen können auf Anfrage zugesandt werden.
Weiterführende Links
Max-Planck-Institut für biophysikalische Chemie, Göttingen http://www.mpibpc.mpg.de/de
A movie of RNA Polymerase II transcription, (Cramer Group). Der erste Film, der den Prozess der Transkription in atomarer Auflösung zeigt. Video 6:05 min, (Standard-YouTube-Lizenz). (Unter dieser Adresse finden sich weitere 5 Videos aus der Cramer Gruppe zu Mechanismen der Transkription)
Animation: The Central Dogma, Nature Video, 10:47 min (englisch; Standard-YouTube-Lizenz)