Wie Computermethoden die Forschung in den Naturwissenschaften verändern
Wie Computermethoden die Forschung in den Naturwissenschaften verändernFr, 28.03.2013 - 04:20 — Peter Schuster

![]() Immanuel Kant (1786) Metaphysische Anfangsgründe der Naturwissenschaft: „…Ich behaupte nur, daß in jeder besonderen Naturlehre nur so viel eigentliche Wissenschaft angetroffen werden könnte, als darin Mathematik anzutreffen ist. …“
Immanuel Kant (1786) Metaphysische Anfangsgründe der Naturwissenschaft: „…Ich behaupte nur, daß in jeder besonderen Naturlehre nur so viel eigentliche Wissenschaft angetroffen werden könnte, als darin Mathematik anzutreffen ist. …“
Bahnbrechende Entwicklungen im 20. Jahrhundert
Basierend auf außerordentlichen technischen Innovationen im zwanzigsten Jahrhundert haben sich die Methoden in den Naturwissenschaften grundlegend geändert. Neue Techniken haben, aufeinander aufbauend, zu einer Flut an weiteren Entwicklungen und diversesten Anwendungen geführt. Einige der bedeutsamsten neuen Methoden sollen hier in chronologischer Reihenfolge aufgezählt werden:
- die Entwicklung hochauflösender spektroskopischer Methoden (beispielsweise Röntgen-, Fluroreszenz-, Kernresonanz-, Elektronenresonanzpektrometrie u.a.) die gleichermaßen ihre Anwendung in der Physik, Chemie und Medizin finden,
- die Entwicklung von Mikromethoden zur Bearbeitung und Analyse von (kleinsten) Proben in Physik, Chemie, Biologie und Medizin,
- die Revolution in der numerischen Mathematik durch elektronische Rechner (Computer),
- die molekulare Revolution in der Biologie, welche vor allem durch neue Techniken zur Sequenzierung von Nukleinsäuren ausgelöst wurde und u.a. zur Sequenzierung des humanen Genoms geführt hat, ebenso wie durch die Kristallisierung von Proteinen, die deren Strukturanalyse und Rückschlüsse auf deren Funktion erlaubte und schließlich durch „lab-on-chip“ Methoden, welche alle Funktionen eines makroskopischen Test-und Analysesystems auf einem nur plastikkartengroßen Chip vereinigen,
- die Entwicklung der Computer-unterstützten Quantenchemie (dafür wurde 1998 der Nobelpreis an – den ursprünglich aus Wien stammenden - Walter Kohn und an John A. Pople vergeben),
- die Revolution in der Bioinformatik, die mehr und mehr die Beschreibung hochkomplexer Systeme möglich macht und zu einer „Systembiologie“ - d.h. einer holistischen Darstellung der Chemie in der Biologie – führt
All diesen Techniken und ihren unterschiedlichen Weiterentwicklungen ist gemeinsam, daß sie ohne „wissenschaftliches Rechnen“, das heißt ohne den intensiven Einsatz von Computermethoden, nicht denkbar wären. Auf diese primär in Punkt 3 und 6 genannten Methoden nimmt der gegenwärtige Artikel – entsprechend seinem Titel – Bezug.
Steigerung von Rechenleistung und Speichervermögen moderner Computer
Die beobachtete Steigerung von Leistungsfähigkeit und Speichervermögen moderner Computer folgt einer empirischen Regel, die Gordon Moore im Jahr 1965 für integrierte Schaltkreise feststellte und fand, daß sich deren Komplexität bei minimalen Kosten der Komponenten innerhalb von 12 – 24 Monaten verdoppelte. Abbildung 1 zeigt, daß diese Regel auch für die Entwicklung von Computern Gültigkeit hat. Der Zugang zu billiger Rechenleistung ist sehr einfach geworden. Im Jahr 2000 lag die Leistung bereits bei 100 Millionen Rechenoperationen pro Sekunde zu einem Preis von $ 1,0 (Abbildung 1). Dazu kommt, daß auch die technischen Probleme der langfristigen Datenspeicherung gelöst sind. Damit werden nun auch hochkomplexe Rechenprobleme immer mehr zur Routine.
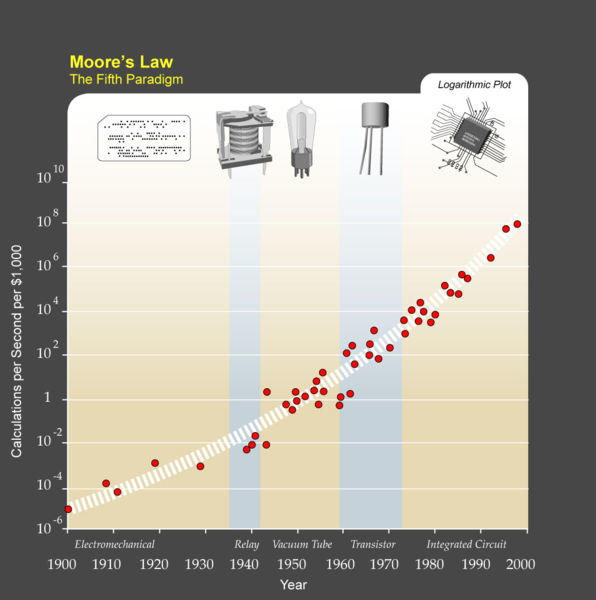 Abbildung 1 Wie sich die Leistungsfähigkeit von Rechnern entwickelte. Adaptation der Moore’schen Regel auf die unterschiedlichen Typen von Rechnern. Zu beachten ist die logarithmische Einteilung der senkrechten Achse: jeweils 100-fache Werte von Teilstrich zu Teilstrich! (Quelle: Ray Kurzweil http://en.wikipedia.org/wiki/Image:PPTMooresLawai.jpg)
Abbildung 1 Wie sich die Leistungsfähigkeit von Rechnern entwickelte. Adaptation der Moore’schen Regel auf die unterschiedlichen Typen von Rechnern. Zu beachten ist die logarithmische Einteilung der senkrechten Achse: jeweils 100-fache Werte von Teilstrich zu Teilstrich! (Quelle: Ray Kurzweil http://en.wikipedia.org/wiki/Image:PPTMooresLawai.jpg)
Was aber noch nicht so sehr Eingang in das öffentliche Bewußtsein gefunden hat, ist die enorme Leistungsfähigkeit von rechnerischen Lösungsansätzen, von Algorithmen, welche die Leistungsfähigkeit der Hardware bereits überflügelt haben. (Unter einem Algorithmus versteht man eine aus endlich vielen Schritten bestehende, eindeutige und ausführbare Handlungsvorschrift zur Lösung eines Problems.) Numerische Mathematiker und Algorithmen-Entwickler schaffen hier ein enorm breites Repertoire an Methoden, welche Lösungen für diverseste Probleme bieten, die auf Grund ihrer Komplexität noch vor kurzem als nicht durchführbar angesehen wurden. Hier sollte insbesondere auf die Arbeiten von Bruno Buchberger hingewiesen werden, dem Gründer und Leiter des Research Institute for Symbolic Computation (RISC) an der Johannes Kepler Universität Linz.
Martin Grötschel, ein deutscher Mathematiker und Experte in Optimierungsfragen an der TU Berlin, hat die Effizienzsteigerung von Computerleistung und Leistungsfähigkeit von Algorithmen am Beispiel eines Modells zur Planung eines Produktionsablaufes aufgezeigt: Wären im Jahr 1988 mit den damaligen Methoden zur Lösung des Problems 82 Jahre vonnöten gewesen, so war der Zeitbedarf 15 Jahre später, im Jahr 2003, bereits auf 1 Minute abgesunken. Zu dieser insgesamt 43-millionenfachen Effizienzsteigerung trug die erhöhte Computerleistung mit einem Faktor 1000 bei, die verbesserten Algorithmen mit einem Faktor 43000.
Von der qualitativen Beschreibung biologischer Vorgänge…
Bis in die siebziger Jahre des vergangenen Jahrhunderts war die quantitative, mathematische Beschreibung von chemisch/biochemischen Prozessen – beispielsweise wie einzelne Enzyme funktionieren oder wie ein Biopolymer seine dreidimensionale Struktur ändert - ein zentrales Element in der biowissenschaftlichen Forschung. Um 1950 wurde dann von dem englischen Strukturbiologen W.T. Astbury der Begriff „Molekularbiologie“ geprägt: diese sollte, über die dreidimensionalen Strukturen der Biomoleküle hinaus, die Fragen nach deren Entstehung, Funktionen und Regulation auf immer höheren Organisationebenen, d.h. immer komplexere Lebensvorgänge, einschließen [1].
Die außerordentlichen Erfolge der Molekularbiologie über mehrere Dekaden hinweg sind unbestritten – eine unvollständige chronologische Aufzählung wichtiger Meilensteine ist in Abbildung 2 dargestellt. Allerdings schloß die Komplexität der Prozesse vorerst eine mathematische Betrachtungsweise und damit eine quantitative Verfolgung über die Zeit hin weitgehend aus.
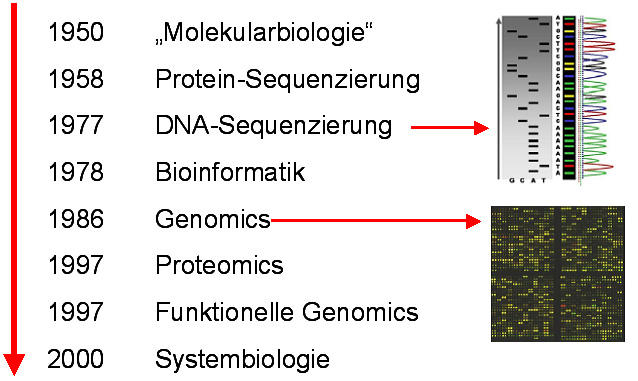 Abbildung 2 . Meilensteine in der molekularen Beschreibung biologischer Vorgänge. Genomics: Sequenzierung und Analyse von Struktur und Funktion des gesamten Genoms in einem System. Proteomics: Sequenzierung und Analyse von Struktur und Funktion aller in einem System exprimierten Proteine (incl. aller Modifikationen).
Abbildung 2 . Meilensteine in der molekularen Beschreibung biologischer Vorgänge. Genomics: Sequenzierung und Analyse von Struktur und Funktion des gesamten Genoms in einem System. Proteomics: Sequenzierung und Analyse von Struktur und Funktion aller in einem System exprimierten Proteine (incl. aller Modifikationen).
Ganz im Gegensatz zu den quantitativen Betrachtungsweisen, die in der Chemie und Physik üblich sind, bot die Molekularbiologie damit ein statisches, qualitatives Bild der Natur, basierend auf „Ja / Nein“-Antworten (beispielsweise: zu einem bestimmten Zeitpunkt ist ein Gen exprimiert oder nicht exprimiert). Dementsprechend sind wir es von der Molekularbiologie gewohnt, Strukturen von Molekülen in grob vereinfachten Bildern dargestellt zu sehen, und Funktionen von Molekülen werden mit Hilfe von Cartoons illustriert anstatt durch mathematische Gleichungen beschrieben zu werden.
Erschwerend kommt dazu, daß eine generell akzeptierte Bezeichnungsweise (Nomenklatur) für die -zigtausend Gene und Genprodukte bislang fehlt: nach wie vor benennen Molekularbiologen neu entdeckte Gene mit willkürlich gewählten Namen, beispielsweise nach den Seiten des Laborjournals oder dem Code des Experiments. Das so entstandene Tohuwabohu an Bezeichnungen macht es selbst für Fachkollegen häufig schwierig den Ausführungen eines Vortragenden zu folgen. Die Molekularbiologie befindet sich hinsichtlich der Nomenklatur noch in einer prä-Linneischen Phase.
…zur holistischen Beschreibung biologischer Systeme
Ziel der biologischen Forschung muß es sein, Arten und Organismen als robuste Einheiten – Systeme – zu verstehen, deren Eigenschaften durch die Dynamik der in ihnen auf unterschiedlichen Zeitskalen und in ständigem Austausch von Energie und Material mit der Umgebung ablaufenden Vorgänge geprägt sind. Das Wissen um diese Vorgänge ist gleichzeitig die Basis dafür, daß Voraussagen über das Verhalten eines Systems gemacht werden können, die auch pathologische Fehlfunktionen erklären und zu deren Behebung beitragen können.
Die Systembiologie will also ein Gesamtbild von den dynamischen Lebensvorgängen und Regulationsvorgängen schaffen: in einer Zellorganelle, einer gesamten Zelle, einem Zellverband, einem gesamten Organismus unter Einbeziehung sämtlicher Ebenen – vom Genom über das Proteom bis hin zu den Regulationsvorgängen kompletter Zellen oder gar einem vollständigen Organismus.
Worum handelt es sich also, wenn man in quantitativer Weise etwa eine typische Säugetierzelle als biologisches System beschreiben will?
Eine Darstellung in einem rezenten Nature-Artikel [2] zeigt am Beispiel einer Herzmuskelzelle, welche Komponenten in Betracht gezogen werden müssen:
- Etwa 20 000 Gene (Genom) werden in mehr als 1 Million unterschiedliche m-RNAs (Transkriptom) und diese in ebenso viele Proteine umgeschrieben.
- Die Proteine werden dann in verschiedener Weise zu mehr als 10 Millionen Proteinen mit veränderten Eigenschaften und Funktionen modifiziert (Proteom),
- die nun den Stoffwechsel der Zelle unter Bildung von mehreren tausend unterschiedlichen kleinen Molekülen (Metabolom) regulieren.
- Zu dem riesigen Datensatz, der die Konzentrationen aller Komponenten zeitabhängig beschreibt (Kinetik der Komponenten), kommen noch die Informationen, welche die Wechselwirkungen zwischen den Komponenten quantitativ zu erfassen versuchen und insgesamt ein enorm komplexes Netzwerk an Regulationsvorgängen ergeben.
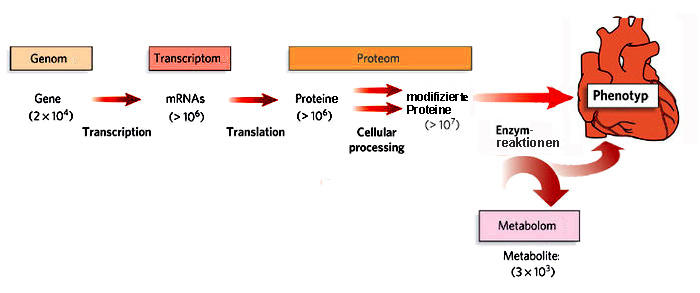 Abbildung 3 . Die Komplexität der Information in einer Säugetierzelle: Vom Genotyp zum Phänotyp. Beschreibung siehe Text. (Quelle: Nature Education [2])
Abbildung 3 . Die Komplexität der Information in einer Säugetierzelle: Vom Genotyp zum Phänotyp. Beschreibung siehe Text. (Quelle: Nature Education [2])
Primär bedürfen systembiologische Untersuchungen daher einer großangelegten, interdisziplinären Zusammenarbeit, vor allem von Chemikern, Biologen, Medizinern, Mathematikern, Computerwissenschaftern, Systemwissenschaftern und Ingenieuren.
Wie wird „systembiologisch“ vorgegangen?
Es müssen vorerst die molekularbiologischen Daten aller Komponenten auf allen Organisationsebenen erhoben und die dynamischen Wechselwirkungen der Komponenten untereinander analysiert werden und dann die enorme Datenmenge durch präzise mathematische Modelle quantitativ beschrieben werden. Das zentrale Element systembiologischer Arbeiten ist dabei ein iterativer Prozeß zwischen Laborexperiment und Computer-Modellierung, der in Abbildung 4 in grober Vereinfachung dargestellt ist: experimentell erhobene Daten werden anhand entsprechender mathematischer Modelle analysiert, mit Hilfe der neu gewonnenen biologischen Information werden bestehende Hypothesen erweitert/verändert, diese durch geeignete experimentelle Ansätze geprüft, anhand von Modellen analysiert, die bestehenden Hypothesen abgeändert usw usf. 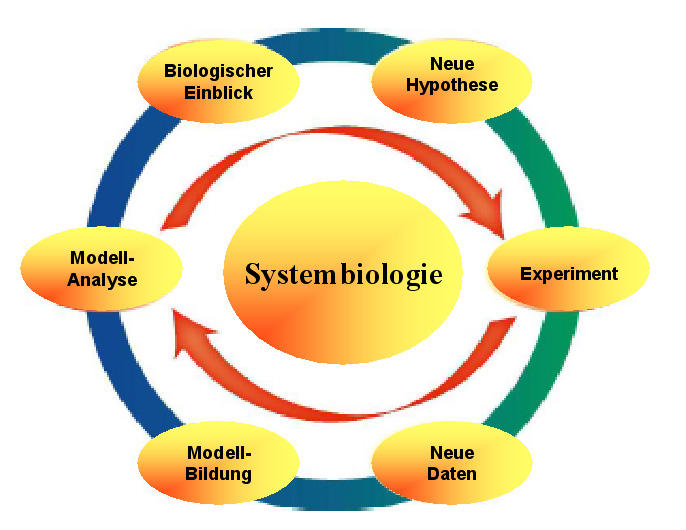 Abbildung 4. Der iterative Prozeß in der Systembiologie (Beschreibung: siehe Text)
Abbildung 4. Der iterative Prozeß in der Systembiologie (Beschreibung: siehe Text)
Ist eine systembiologische Beschreibung komplexer Systeme noch reine Utopie?
Zweifellos ist dies nicht mehr der Fall. In der kurzen Zeit seit ihren Anfängen hat sich die Systembiologie weltweit zu einer der dynamischsten Forschungsdisziplinen entwickelt und nimmt unter diesen bereits eine Spitzenposition ein. Reichlich gefördert, auch von öffentlicher Hand, begeben sich mehr und mehr Forschergruppen in dieses zukunftsträchtige Gebiet und untersuchen unterschiedlichste Systeme – von primitiven Einzellern bis hin zu komplexen Säugerzellen und ganzen Organismen. Um hier nur eine (kleine) Auswahl der Institutionen aufzuzählen:
In den USA wird Systembiologie u.a. durch das National Institute of Health (NIH) gefördert: die “National Centers for Systems Biology”, sind 15 an den prominentesten Universitäten angesiedelte Institutionen, die sowohl Grundlagen erarbeiten („The quantity and quality of data required for the approaches often challenge current technologies, and the development of new technologies and cross-disciplinary collaborations may be required.“) als auch die unterschiedlichsten Systeme von Hefezellen und Pflanzenzellen bis hin zu einer virtuellen Laborratte systembiologisch untersuchen.
In Japan wurde bereits im Jahr 2000 das „Systems Biology Institute“ (SBI) von Hiroaki Kitano gegründet – einem der Väter der Systembiologie; dieses untersucht Systeme in Hinblick auf das Design von Pharmazeutika, auf toxikologische Probleme, auf Infektions-ausgelöste Antworten des Wirts u.v.a.m.
In Europa haben sich sehr viele nationale und internationale systembiologisch arbeitende Gruppen angesiedelt. So gibt es in der Schweiz die „Swiss Research Initiative in Systems Biology“, in der zur Zeit mehr als 1000 Wissenschafter in rund 300 Forschungsgruppen mehr als 1000 Projekte bearbeiten.
In Barcelona ist eine Gruppe um Luis Serrano mit der systembiologischen Beschreibung eines der einfachsten Organismen, dem Bakterium Mycoplasma pneumoniae beschäftigt: dieses Bakterium ist eine der kleinsten Lebensformen, es wird von einer Minimalausrüstung von nur rund 700 Genen gesteuert (beim Menschen sind es 30 mal so viele), weist aber enorm komplexe Regulationsvorgänge auf.
Zahlreiche Projekte werden in Deutschland u.a. vom Deutschen Bundesministerium für Bildung und Forschung gefördert – ein halbjährlich erscheinendes Magazin gibt in leichtverständlicher Sprache Auskunft über Initiativen und Fortschritte [3]. Beispielsweise wurde in der Initiative „Hepatosys“ eine virtuelle Leberzelle modelliert, welche breite Anwendungsmöglichkeiten für die Medizin, die Pharmaforschung oder den Bereich Ernährung verspricht [4].
Transnationale Initiativen in Europa werden u.a. durch die “European Research Area for Systems Biology” – ERASysBio gefördert (beispielsweise untersuchen hier Forschergruppen aus mehreren Staaten gemeinsam "Systems Biology of Microorganisms").
Ausblick
Vor 11 Jahren hat der Biologe (und Nobelpreisträger) Sidney Brenner für die Vorgangsweise in der modernen biologischen Forschung die Metapher von Jägern und Sammlern gebraucht [5]: „Im prä-genomischen Zeitalter (Anmerkung: d.i. vor der Sequenzierung menschlichen Genoms im Jahr 2001) hatte man mir beigebracht Jäger zu sein. Ich hatte gelernt, wilde Tiere zu identifizieren, aufzustöbern und zu erlegen. Nun aber sind wir gezwungen Sammler zu sein, alles Herumliegende zusammenzutragen und in Lagerhallen zu speichern. Eines Tages wird hoffentlich jemand kommen, der sämtlichen Mist beseitigt, aber die wesentlichen Befunde behält (die Schwierigkeit liegt darin diese zu erkennen).“
Das Sammeln hat seit dieser Zeit zugenommen.
Die enorme Steigerung an Rechnerleistung und an der Leistungsfähigkeit rechnerischer Modelle ermöglicht die Speicherung, Analyse und Bearbeitung ungeheurer Datenmengen. Die quantitative Beschreibung komplexer Lebensformen erscheint nicht mehr als Utopie; es ist aber erst ansatzmäßig möglich die Funktion einfachster zellulärer Netzwerke darzustellen: vergleicht man ein derartiges Netzwerk mit einem Orchester, so sind wohl die enorm zahlreichen Instrumente - Gene, Gen-Transkripte, Proteine und Metaboliten – durch Hochdurchsatz- (high-throughput)- Techniken bestimmt worden, es fehlen aber weitestgehend noch essentielle Angaben zu deren räumlicher und zeitlicher Abstimmung – also die Partitur. Derartige „spatiotemporal maps of molecular behavior” werden von einer Reihe Institutionen, u.a. an den “National Centers for Systems Biology” (siehe oben) erarbeitet.
[1] WT Astbury (1961) Molecular Biology or Ultrastructural Biology? Nature 17:1124
[2] G Potters (2010) Systems Biology of the Cell. Nature Education 3(9):33
[3] systembiologie.de: MAGAZIN FÜR SYSTEMBIOLOGISCHE FORSCHUNG IN DEUTSCHLAND
[4] Systems Biology - Results, Progress and Innovations from BMBF Funding
[5] S Brenner (2002) Hunters and gatherers. The Scientist 16(3):14.
Weiterführende links
Science Education: Computers in Biology. Website des NIH mit sehr umfangreichen, leicht verständlichen Darstellungen (englisch)
Mycoplasma: Ein Modellorganismus für die Systembiologie.
Die virtuelle Leber (Virtual Liver Networks: “The Virtual Liver will be a dynamic model that represents, rather than fully replicates, human liver physiology morphology and function, integrating quantitive data from all levels of organisation”). Englisch.
Vorträge des Autors auch zu diesem Thema können von seiner Webseite heruntergeladen werden.