Superauflösende Mikroskopie zeigt Aufbau und Dynamik der Bausteine in lebenden Zellen
Superauflösende Mikroskopie zeigt Aufbau und Dynamik der Bausteine in lebenden ZellenFr, 04.09.2015 - 16:33 — Redaktion
Vor einer Woche ist im Fachjournal Science eine Studie erschienen*, die weltweites Aufsehen erregt. Mittels neuer fluoreszenzmikroskopischer Methoden gelang es Forschern um Eric Betzig (Howard Hughes Medical Institute, Janelia Farm) den inneren Aufbau lebender Zellen und darin ablaufende Prozesse in hoher räumlicher Auflösung – bis hin zur Sichtbarmachung einzelner Proteine - und zeitlicher Auflösung (Millisekundenbereich) zu filmen. An Hand von Beispielen - u.a. dem Aufbau/Umbau des Zytoskeletts oder der Entwicklung von Mitochondrien – lässt sich das ungeheure Potential dieser superauflösenden Mikroskopie erahnen, die in unterschiedlichsten Labors leicht implementierbar sein dürfte und damit eine Revolution der biologischen, biomedizinischen (Grundlagen)Forschung verspricht.
Jeder biologisch arbeitende Forscher wünscht sich wohl biochemische Prozesse nicht nur unter mehr oder weniger artifiziellen Bedingungen im Reagenzglas - in vitro - untersuchen zu können. Vielmehr möchte er direkt verfolgen, wie die Vorgänge ablaufen, wie Biomoleküle in ihrer komplexen natürlichen Umgebung, der lebenden Zelle, reagieren, wie dort Strukturen auf- und umgebaut werden, wie Organellen entstehen und funktionieren.
Die Grenzen des Lichtmikroskops
Der zellbiologisch arbeitende Forscher verbringt meistens sehr viel von seiner Zeit über das Mikroskop gebeugt, in die Beobachtung seiner wachsenden, sich teilenden, verändernden, sterbenden Zellen vertieft. Für Untersuchungen auf molekularer Ebene erweisen sich herkömmliche Lichtmikroskope allerdings kaum geeignet. Auch wenn sie mit den denkbar besten Optiken ausgestattet sind, können Lichtmikroskope Objekte nur dann getrennt, scharf wahrnehmen, wenn diese mindestens 200 – 300 Nanometer (nm - Milliardstel Meter) voneinander entfernt vorliegen. Diese Grenze der Auflösung wird physikalisch durch die Beugung der Lichtwellen bestimmt und hängt im Wesentlichen von der Wellenlänge des verwendeten Lichts (der Wellenlängenbereich des für Menschen sichtbaren Lichtspektrums erstreckt sich von etwa 380 nm bis 780 nm) und dem Öffnungswinkel des Objektivs ab. Diese – nicht nur auf Lichtwellen beschränkte - Erkenntnis hat der deutsche Physiker Ernst Carl Abbe um 1870 in dem berühmten, nach ihm benannten Gesetz formuliert (Abbildung 1).
Dass man mit dem Lichtmikroskop Strukturen unterhalb einer Größe von 200 nm nicht sehen kann, wurde zu einem über 100 Jahre streng geltenden Dogma. Damit erschien eine direkte Betrachtung essentieller Zellbausteine in ihrem natürlichen Umfeld ausgeschlossen. Als Beispiele sind Proteinmoleküle zu nennen, deren durchschnittliche Abmessungen im niedrigen Nanometerbereich liegen. Auch viele gerade noch erkennbare Zellorganellen (beispielsweise Mitochondrien, Endosomen) sind zu klein für detailliertere Untersuchungen zu Aufbau und Funktion.
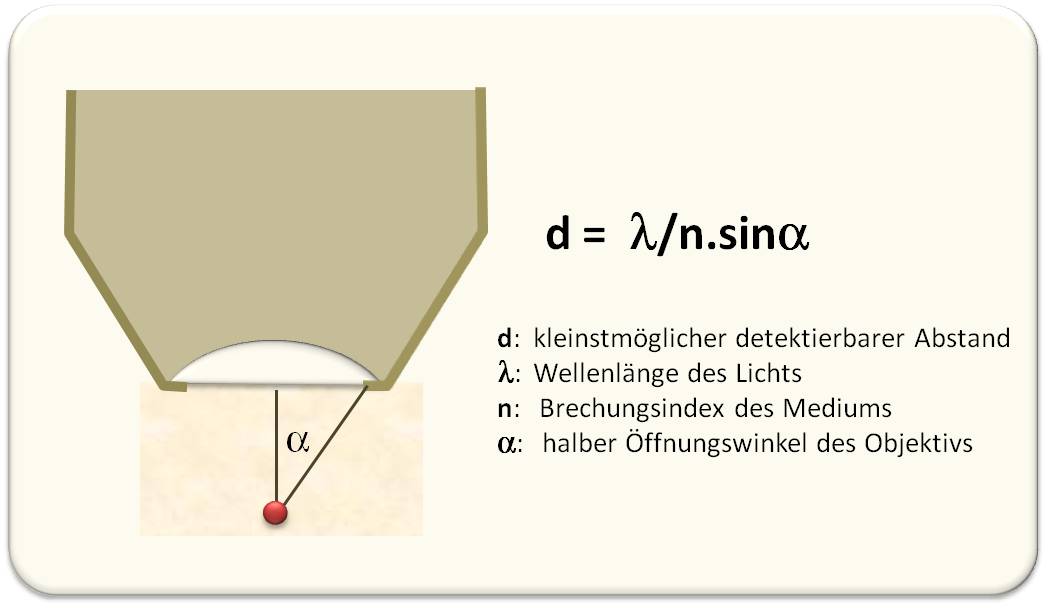 Abbildung 1. Die Auflösung in der Mikroskopie – die Abbe’sche Formel. Der Wellenlängenbereich des für Menschen sichtbaren Lichtspektrums erstreckt sich von etwa 380 nm bis 780 nm. Daraus berechnet sich ein Grenzwert der Auflösung von 200 – 300 nm. (Objektiv: olivgrün, Objekt: rot)
Abbildung 1. Die Auflösung in der Mikroskopie – die Abbe’sche Formel. Der Wellenlängenbereich des für Menschen sichtbaren Lichtspektrums erstreckt sich von etwa 380 nm bis 780 nm. Daraus berechnet sich ein Grenzwert der Auflösung von 200 – 300 nm. (Objektiv: olivgrün, Objekt: rot)
Sichtbarmachung von Nanostrukturen
Die Abbe’sche Formel zeigt: eine höhere Auflösung kann erreicht werden, wenn i) der Öffnungswinkel des Objektivs und/oder der Brechungsindex des Mediums, welches das Objekt umgibt, vergrößert wird und/oder ii) Strahlung mit niedrigerer Wellenlänge und damit höherer Energie angewendet wird.
- Im ersteren Fall wird das Objekt näher an das Objektiv herangebracht - dies führt aber (ebenso wie eine Erhöhung des Brechungsindex) nur zu mäßiger Zunahme der Auflösung (sin 45o = 0,707 bis hin zu sin 90o = 1).
- Die zweite Möglichkeit wird unter anderem in Methoden der Röntgenstreuung, Neutronenstreuung und insbesondere der Elektronenmikroskopie realisiert: es sind dies Standardtechniken der modernen Biowissenschaften, welche die Grundlage unserer Kenntnisse über den Aufbau von Biomolekülen geschaffen haben. Diese Methoden bieten die zur Analyse von Nanostrukturen – Molekülen bis hin zu Atomen - benötigte Auflösung. Die eingestrahlte Energie ist aber so hoch, die Versuchsbedingungen so lebensfeindlich – z.B. Arbeiten im Vakuum, bei sehr tiefen Temperaturen -, dass Messungen unter lebensnahen Bedingungen, direkte Beobachtungen biologischer Prozesse in lebenden Zellen nicht möglich sind.
Die Umgehung der Abbe‘schen Beugungsgrenze: Das Lichtmikroskop wird zum Nanoskop
In den letzten beiden Jahrzehnten wurden Methoden entwickelt, welche die bislang für absolut gehaltene, Abbe‘sche Grenze der optischen Auflösung in der Lichtmikroskopie - speziell in der in Biologie und Biomedizin sehr häufig verwendeten Fluoreszenmikroskopie - umgehen und Nanostrukturen und deren Dynamik sichtbar machen. Dies haben die US-Amerikaner Eric Betzig (Howard Hughes Medical Institute, Janelia Farm) und William Moerner (Stanford University, CA) und der Deutsche Stefan Hell (Max-Planck Institut für Biophysikalische Chemie/Göttingen) voneinander unabhängig und mit unterschiedlichen Strategien erreicht. Sie wurden dafür 2014 mit dem Nobelpreis für Chemie ausgezeichnet.
Prinzipiell werden in der Fluoreszenzmikroskopie Moleküle der Zelle mit fluoreszierenden Farbstoffen markiert. Ein Lichtstrahl (Laserlicht) einer bestimmten Wellenlänge regt diese Moleküle an –schaltet sie an –, sodass sie leuchten. Üblicherweise leuchtet dann die gesamte Probenoberfläche, sodass die Abstände zwischen den einzelnen angeregten Molekülen unter dem Abbeschen Grenzwert liegen und Strukturen unter 200 nm nicht mehr gesehen warden können.
Die STED-Mikroskopie
Stefan Hell hat hier mit der von ihm entwickelten STED-Mikroskopie (STimulated Emission Depletion, „stimulierte Emissions-Löschung“) einen Ausweg gefunden. Dem Lichtpuls, der die Fluoreszenzmoleküle anschaltet, schickt er sofort einen zweiten Lichtpuls – den STED-Puls – hinterher, der gezielt bis auf einen winzigen Ausschnitt alle angeregten Moleküle „abschaltet“ (d.h. zwingt ein Photon abzugeben = stimulierte Emission), sodass sie nicht mehr leuchten. Nur der winzige (Nanometer-kleine) leuchtende Ausschnitt wird detektiert – je kleiner dieser ist, desto höher wird die erzielte Auflösung. Nach diesem Anregung-Abschaltung Schema scannen die beiden Lichtpulse die gesamte Probenoberfläche. Aus den einzelnen detektierten Lichtpunkten wird dann via Computerprogramm das gesamte Bild zusammengesetzt. Mit dieser Technik werden Auflösungen von wenigen Nanometern, d.i. von der Größenordnung von Proteinen, erreicht.
Neben der enorm hohen räumlichen Auflösung wird auch eine sehr rasche zeitliche Auflösung erzielt. In einer eben erschienen Untersuchung** demonstriert die Hell-Gruppe die Dynamik von Vesikeln in der Nervenzelle einer lebenden Fliegenlarve und die Aufnahme von Viruspartikeln in lebende Zellen mit einer zeitlichen Auflösung von 5 – 10 Millisekunden.
Die SIM-Mikroskopie („Strukturierte Illuminationsmikroskopie“)
Auch die von Eric Betzig und William Moerner unabhängig entwickelten Strategien basieren auf der Fluoreszenzmikroskopie. Moerner, der als erster die Lichtabsoption an Einzelmolekülen gemessen hatte und Betzig experimentierten mit einem damals neu entdeckten, aus Quallen stammenden Protein, dem sogenannten Green Fluorescent Protein (GFP), dessen Fluoreszenz sich durch entsprechende Lichtpulse gezielt ein- und ausschalten lässt. GFP (aber auch andere fluoreszierende Proteine) können in Zellen eingeschleust und an andere Proteine gekoppelt werden: die Position und Dynamik dieser Proteine in der Zelle kann dann an Hand der angekoppelten anregbaren Fluoreszenz verfolgt werden.
Der Trick zur Umgehung der Abbe’schen Beugungsgrenze: die Probe wird mit einem sehr schwachen Lichtimpuls angeregt, welcher nur einen so kleinen Teil der Moleküle zum Fluoreszieren bringt, dass deren Abstände voneinander größer als 200 nm sind. Nach dem Ausbleichen dieser Moleküle wird die nächste kleine Untergruppe belichtet, dann die nächste, usf. Die resultierenden Bilder werden von Kameras aus verschiedenen Richtungen registriert und über Computeralgorithmen zu superaufgelösten Bildern zusammengesetzt und mit hoher zeitlicher Auflösung zu kurzen Videos. Mehr als lange Beschreibungen vermögen, zeigen diese Videos erstmals faszinierende Einblicke in die Vorgänge lebender Zellen in Echtzeit und auf molekularem Niveau (Video 1, Video 2).
Video 1. Dynamik der Verankerung des Zytoskeletts. Das fadenförmige Strukturprotein Aktin (violett fluorezierend: mApple-F-tractin) interagiert mit den bipolaren Kopfgruppen (EGFP, grün) des verankernden Proteins Paxillin . Aufnahmen:81 Zeitpunkte in Intervallen von 20 sec. (Movie 1, High NA SIM of actomyosin dynamics von HHMI NEWS PRO Mo 17. August 2015; weitere Details unter*)
Video 2. Dynamik von Mitochondrien in einer COS-7 Zelle. Markierung mit einem fluoreszierenden Membran-Marker (Skylan-NS-TOM20) Man kann beobachten, wie einzelne Mitochondrien sich kontrahieren, teilen und auch fusionieren, Aufnahmen: 20 Zeitpunkte in Intervallen von 2 min, der Farbcode zeigt die Entfernung vom Sunstrat. (Movie 10, 3D nonlinear SIM of mitochondrial dynamics von HHMI NEWS PRO vor 2 Wochen; weitere Details unter*)
Ausblick
Die faszinierenden neuen Techniken der „Nanoskopie“ stellen einen außerordentlichen Durchbruch dar für Grundlagen- und angewandte Forschung, dem weltweit Mit der Möglichkeit einzelne Biomoleküle in ihrem natürlichen Umfeld - auch in der Dynamik ihrer Wechselwirkungen - zu erfassen, wird die Basis zu einer wesentlich aussagekräftigeren systembiologischen Behandlung „Komplexer biologischer Systeme“ geschaffen. Die Pioniere in diesem Gebiet selbst wenden ihre Techniken an, um Einblicke in fundamentale biologische/pathologische Mechanismen zu gewinnen: Stefan Hell in die Mechanismen in Nervenzellen und an ihren Synapsen, W. E.Moerner in die Entstehung der Huntigton’schen Erkrankung und Eric Betzig in die Zellteilung in Embryonen.
*Dong Li e al., Extended-resolution structured illumination imaging of endocytic and cytoskeletal dynamics. Science (28. August 2015) 349 (6251)
** Jale Schneider et al., Ultrafast, temporally stochastic STED nanoscopy of milliseconds dynamics. Nature Methods, 12 (9) September 2015.
Weiterführende Links
How the optical microscope became a nanoscope. Populäre Information zum Nobelpreis in Chemie 2014 (PDF-Download; englisch)
Pushing the Envelope in Biological Fluorescence Microscopy. Distinguished Lecture by Dr. Eric Betzig (das Video zeigt großartige Bilder; in English) Video 36:45 min.
Nanoscopy with focused light. Stefan W. Hell: Nobel Lecture at Uppsala University (2014). Video 47:48 min.
Sehr weitreichende Informationen zu den Techniken der Superauflösenden Mikroskopie sind von den homepages der 3 Nobelpreisträger abrufbar:
Eric Betzig: http://janelia.org/lab/betzig-lab
Stefan W. Hell: www3.mpibpc.mpg.de/groups/hell
William W. Moerner: http://web.stanford.edu/group/moerner
